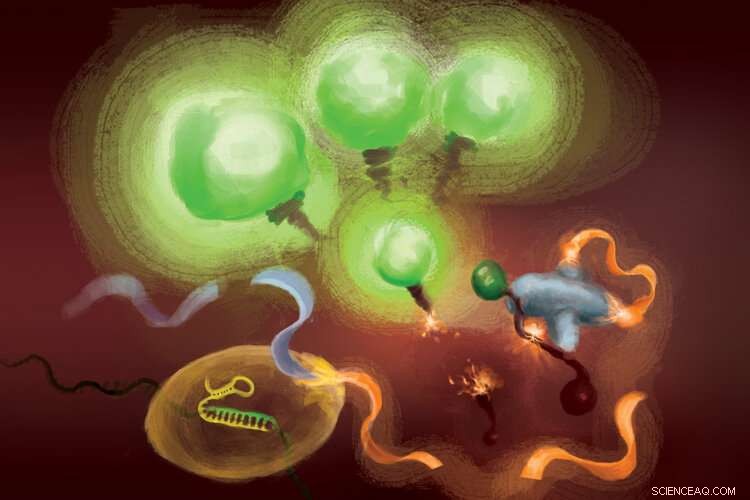
Le reazioni coinvolte nel test FIND-IT per rilevare l'infezione da virus SARS-CoV-2. Quando l'enzima Cas13 (in basso a sinistra) si lega al suo RNA bersaglio, taglia una molecola (nastro arancione e azzurro) per rilasciare un attivatore (arancione) che sovralimenta la nucleasi Csm6 (a destra) per scindere e rilasciare molecole fluorescenti che si illuminano (verde) e segnalano la presenza di RNA virale. Credito:Margaret L. Liu, Pritzker School of Medicine dell'Università di Chicago.
Frequente, test rapidi per COVID-19 sono fondamentali per controllare la diffusione dei focolai, soprattutto come nuovo, emergono varianti più trasmissibili.
Mentre il test diagnostico COVID-19 gold standard di oggi, che utilizza qRT-PCR - reazione a catena quantitativa della trascrittasi inversa-polimerasi (PCR) - è estremamente sensibile, rilevando fino a una copia di RNA per microlitro, richiede attrezzature specializzate, un'autonomia di diverse ore e una struttura di laboratorio centralizzata. Di conseguenza, il test richiede in genere almeno uno o due giorni.
Un team di ricerca guidato da scienziati nei laboratori di Jennifer Doudna, David Savage e Patrick Hsu dell'Università della California, Berkeley, mira a sviluppare un test diagnostico che sia molto più veloce e più facile da implementare rispetto a qRT-PCR. Ora ha combinato due diversi tipi di enzimi CRISPR per creare un test in grado di rilevare piccole quantità di RNA virale in meno di un'ora. Doudna ha condiviso il premio Nobel per la chimica 2020 per l'invenzione dell'editing del genoma CRISPR-Cas9.
Sebbene la nuova tecnica non sia ancora nella fase in cui rivaleggia con la sensibilità della qRT-PCR, in grado di rilevare solo poche copie del virus per microlitro di liquido, è già in grado di captare livelli di RNA virale – circa 30 copie per microlitro – sufficienti per essere utilizzati per sorvegliare la popolazione e limitare la diffusione delle infezioni.
"Non è necessaria la sensibilità della PCR per rilevare e diagnosticare sostanzialmente il COVID-19 nella comunità, se il test è abbastanza conveniente e abbastanza veloce, " ha detto il co-autore David Savage, professore di biologia molecolare e cellulare. "La nostra speranza era quella di portare la biochimica il più lontano possibile al punto in cui si potesse immaginare un formato molto conveniente in un ambiente in cui si potesse essere testati ogni giorno, dire, all'ingresso del lavoro."
I ricercatori riporteranno i loro risultati online il 5 agosto sulla rivista Natura chimica biologia .
Diversi test basati su CRISPR sono stati autorizzati per l'uso di emergenza dalla Food and Drug Administration, ma tutti richiedono una fase iniziale in cui l'RNA virale viene amplificato in modo che il segnale di rilevamento, che comporta il rilascio di una molecola fluorescente che si illumina sotto la luce blu, sia abbastanza luminoso da essere visto. Sebbene questa amplificazione iniziale aumenti la sensibilità del test a un livello simile a qRT-PCR, introduce inoltre passaggi che rendono più difficile l'esecuzione del test al di fuori di un laboratorio.
Il team guidato dall'UC Berkeley ha cercato di raggiungere una sensibilità e una velocità utili senza sacrificare la semplicità del test.
"Per le applicazioni point of care, vuoi avere una risposta rapida in modo che le persone possano sapere rapidamente se sono infette o meno, prima di prendere un volo, Per esempio, o andare a trovare i parenti, " ha detto il caposquadra Tina Liu, un ricercatore nel laboratorio di Doudna presso l'Innovative Genomics Institute (IGI), un centro incentrato su CRISPR che coinvolge scienziati dell'UC Berkeley e dell'UC San Francisco.
Oltre ad avere un passaggio in più, un altro svantaggio dell'amplificazione iniziale è che, perché produce miliardi di copie di RNA virale, esiste una maggiore possibilità di contaminazione incrociata tra i campioni dei pazienti. La nuova tecnica sviluppata dal team capovolge questo e invece aumenta il segnale fluorescente, eliminando una delle principali fonti di contaminazione incrociata.
La tecnica senza amplificazione, che chiamano Fast Integrated Nuclease Detection In Tandem (FIND-IT), potrebbe consentire test diagnostici rapidi ed economici per molte altre malattie infettive.
"Anche se abbiamo avviato questo progetto con l'esplicito scopo di avere un impatto sul COVID-19, Penso che questa particolare tecnica potrebbe essere applicabile a qualcosa di più di questa pandemia perché, in definitiva, CRISPR è programmabile, "Liu ha detto. "Quindi, potresti caricare l'enzima CRISPR con una sequenza mirata al virus dell'influenza o al virus dell'HIV o a qualsiasi tipo di virus a RNA, e il sistema ha il potenziale per funzionare allo stesso modo. Questo documento stabilisce davvero che questa biochimica è un modo più semplice per rilevare l'RNA e ha la capacità di rilevare quell'RNA in un lasso di tempo sensibile e veloce che potrebbe essere suscettibile di future applicazioni nella diagnostica del punto di cura".
I ricercatori stanno attualmente sviluppando una tale diagnostica utilizzando FIND-IT, che includerebbe passaggi per raccogliere ed elaborare i campioni e per eseguire il test su un dispositivo microfluidico compatto.
Utilizzo di proteine Cas in tandem
Per rimuovere l'amplificazione target dall'equazione, il team ha impiegato un enzima CRISPR, Cas13, per rilevare prima l'RNA virale, e un altro tipo di proteina Cas, chiamato Csm6, amplificare il segnale di fluorescenza.
Cas13 è una forbice multiuso per il taglio dell'RNA; una volta che si lega alla sua sequenza bersaglio, specificato da un RNA guida, è innescato per tagliare una vasta gamma di altre molecole di RNA. Questa attività di taglio innescata dal bersaglio può essere sfruttata per accoppiare il rilevamento di una specifica sequenza di RNA al rilascio di una molecola reporter fluorescente. Però, da solo, Cas13 può richiedere ore per generare un segnale rilevabile quando sono presenti quantità molto basse di RNA bersaglio.
L'intuizione di Liu è stata quella di utilizzare Csm6 per amplificare l'effetto di Cas13. Csm6 è un enzima CRISPR che rileva la presenza di piccoli anelli di RNA e si attiva per tagliare un'ampia gamma di molecole di RNA nelle cellule.
Per aumentare il rilevamento di Cas13, lei e i suoi colleghi hanno progettato una molecola attivatore appositamente progettata che viene tagliata quando Cas13 rileva l'RNA virale. Un frammento di questa molecola può legarsi e attivare Csm6 per tagliare e rilasciare una molecola fluorescente brillante da un pezzo di RNA. Normalmente, la molecola attivatore viene rapidamente scomposta da Csm6, limitando così la quantità di segnale fluorescente che può generare. Liu e i suoi colleghi hanno escogitato un modo per modificare chimicamente l'attivatore in modo che sia protetto dalla degradazione e possa sovraccaricare Csm6 per tagliare e rilasciare ripetutamente molecole fluorescenti legate all'RNA. Ciò si traduce in una sensibilità 100 volte migliore rispetto all'attivatore originale.
"Quando Cas13 viene attivato, fende questo piccolo attivatore, rimuovendo un segmento che lo protegge, " Liu ha detto. "Ora che è liberato, può attivare molte molecole diverse di quel secondo enzima, Csm6. E così, un bersaglio riconosciuto da Cas13 non porta solo all'attivazione della propria capacità di taglio dell'RNA; porta alla generazione di molti più enzimi attivi che possono poi scindere reporter ancora più fluorescenti".
Il team di ricercatori ha anche incorporato una combinazione ottimizzata di RNA guida che consente un riconoscimento più sensibile dell'RNA virale da parte di Cas13. Quando questo è stato combinato con Csm6 e il suo attivatore, il team è stato in grado di rilevare fino a 31 copie per microlitro di RNA SARS-CoV-2 in appena 20 minuti.
I ricercatori hanno anche aggiunto l'RNA estratto dai campioni dei pazienti al test FIND-IT in una cartuccia microfluidica, per vedere se questo test può essere adattato per essere eseguito su un dispositivo portatile. Utilizzando un piccolo dispositivo con una fotocamera, potrebbero rilevare l'RNA di SARS-CoV-2 estratto dai campioni dei pazienti con una sensibilità che catturerebbe le infezioni da COVID-19 al loro apice.
"Questo approccio nucleasi tandem—Cas13 più Csm6—combina tutto in un'unica reazione a una singola temperatura, 37 gradi Celsius, quindi non richiede riscaldamento ad alta temperatura o passaggi multipli, come è necessario per altre tecniche diagnostiche, " Liu ha detto. "Penso che questo apra le opportunità per più veloce, test più semplici che possono raggiungere una sensibilità paragonabile ad altre tecniche attuali e potrebbero potenzialmente raggiungere sensibilità ancora più elevate in futuro".
Lo sviluppo di questo metodo senza amplificazione per il rilevamento dell'RNA è il risultato di un riorientamento della ricerca all'interno dell'IGI quando la pandemia è iniziata verso i problemi di diagnosi e trattamento di COVID-19. In definitiva, cinque laboratori dell'UC Berkeley e due laboratori dell'UCSF sono stati coinvolti in questo progetto di ricerca, uno dei tanti all'interno dell'IGI.
"Quando abbiamo iniziato questo, speravamo di creare qualcosa che raggiungesse la parità con la PCR, ma non necessitava di amplificazione, quello sarebbe stato il sogno, "disse Selvaggio, che è stato il principale investigatore del progetto. "E dal punto di vista della sensibilità, avevamo circa diecimila volte il divario da saltare. L'abbiamo fatto circa mille volte; l'abbiamo abbassato di circa tre ordini di grandezza. Così, ci siamo quasi. Lo scorso aprile, quando stavamo davvero iniziando a mapparlo, sembrava quasi impossibile".