Unendo insieme le molecole, gli scienziati dell'Università di Montréal pensano di aver scoperto come i sistemi molecolari all'origine della vita si sono evoluti per creare complesse funzioni autoregolanti.
Pubblicato in Angewandte Chemie , le loro scoperte promettono di fornire a chimici e nanotecnologi una strategia semplice per creare la prossima generazione di nanosistemi dinamici.
La vita sulla Terra è sostenuta da milioni di diverse minuscole nanostrutture o nanomacchine che si sono evolute nel corso di milioni di anni, ha spiegato Alexis Vallée-Bélisle, professore dell'UdeM e ricercatore principale dello studio.
Queste strutture, spesso più piccole di 10.000 volte il diametro di un capello umano, sono tipicamente composte da proteine o acidi nucleici. Mentre alcuni sono realizzati da un singolo componente o parte (spesso polimeri lineari che si piegano in una struttura specifica), la maggior parte di essi è realizzata utilizzando diversi componenti che si assemblano spontaneamente in assiemi grandi e dinamici.
"Questi complessi molecolari sono altamente dinamici e si attivano o disattivano proprio in risposta a vari stimoli come una variazione di temperatura, ossigeno o sostanze nutritive", ha affermato Vallée-Bélisle.
"Analogamente alle auto che richiedono l'accensione sequenziale, il rilascio dei freni, il cambio di marcia e l'immissione di gas per andare avanti, i sistemi molecolari richiedono l'attivazione o la disattivazione sequenziale di varie nanomacchine per eseguire compiti specifici che vanno dal movimento, alla respirazione al pensiero."
I ricercatori hanno sollevato una domanda fondamentale:come sono stati creati, programmati e messi a punto gli assemblaggi molecolari dinamici per supportare la vita?
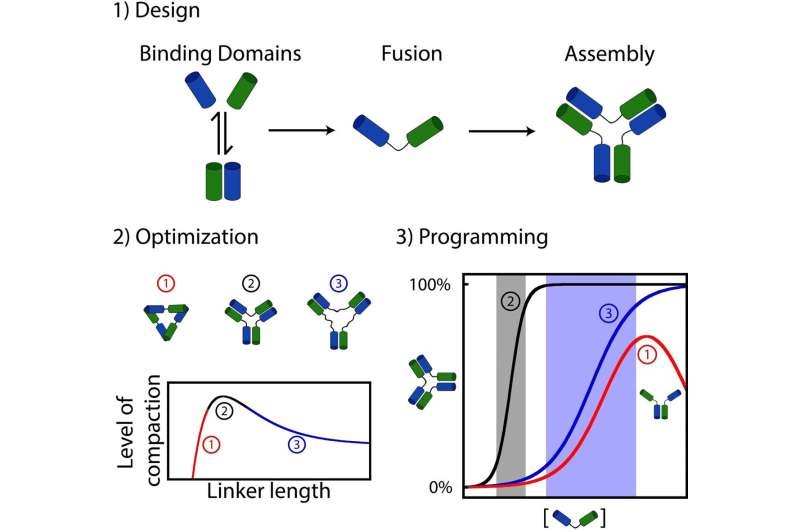
Ciò che hanno scoperto è che molti assemblaggi biologici erano probabilmente formati unendo in modo casuale molecole interagenti (ad esempio proteine o acidi nucleici come DNA o RNA) con linker che fungevano da "connettore" tra ciascuna parte.
"Poiché questi insiemi biomolecolari svolgono un ruolo cruciale nel consentire agli organismi viventi di rispondere al loro ambiente, abbiamo ipotizzato che la natura della connettività tra i componenti collegati possa anche contribuire all'evoluzione delle loro risposte dinamiche", ha affermato Vallée-Bélisle, titolare della Cattedra di ricerca canadese in Bioingegneria e Bionanotecnologia.
Per esplorare questa domanda, Dominic Lauzon, uno studente di dottorato all'epoca dello studio, ha deciso di sintetizzare e collegare insieme dozzine di molecole interagenti del DNA per esplorare l'impatto della connettività sulla dinamica dell'assemblaggio.
"La chimica programmabile e facile da usare degli acidi nucleici come il DNA lo rende una molecola conveniente per studiare questioni fondamentali legate all'evoluzione delle biomolecole", ha affermato Lauzon, il primo autore dello studio. "Inoltre si ritiene che gli acidi nucleici siano anche la molecola all'origine della vita sulla Terra."
Lauzon e Vallée-Bélisle hanno scoperto che una semplice variazione nella lunghezza del "linker" tra le molecole interagenti porta a variazioni significative nella loro dinamica di assemblaggio. Ad esempio, alcuni gruppi hanno mostrato un'elevata sensibilità alla variazione degli stimoli, mentre altri mancavano di tale sensibilità o addirittura richiedevano cambiamenti molto più ampi negli stimoli per promuovere l'assemblaggio.
Ancora più sorprendente, alcuni linker hanno persino creato nuove funzioni regolatrici complesse come le proprietà di autoinibizione, dove l’aggiunta di uno stimolo ne promuoverebbe sia l’assemblaggio che il disassemblaggio. Tutti questi diversi comportamenti reattivi sono spesso osservati anche nelle nanomacchine "viventi" naturali.
Utilizzando esperimenti ed equazioni matematiche, i ricercatori sono stati anche in grado di spiegare perché una variazione così semplice della lunghezza del linker era così efficace nel modificare la dinamica dell'assemblaggio molecolare.
"I linker che creavano gli assemblaggi più stabili erano quelli che creavano anche i meccanismi di attivazione più sensibili, mentre i linker che creavano gli assemblaggi meno stabili creavano i meccanismi di attivazione meno sensibili, fino al punto di introdurre l'autoinibizione," ha spiegato Lauzon.
La capacità di percepire con precisione i segnali molecolari è cruciale per gli assemblaggi biologici ma anche nello sviluppo della nanotecnologia che dipende dal rilevamento e dall'integrazione delle informazioni molecolari.
I ricercatori ritengono quindi che la loro scoperta possa anche fornire il quadro fondamentale per creare nanomacchine o nanosistemi più programmabili con attività regolate in modo ottimale, ad esempio semplicemente collegando molecole interagenti con diversi linker. Tali gruppi molecolari stanno già trovando applicazioni nel biosensing o nella somministrazione di farmaci.
Oltre a fornire una semplice strategia di progettazione per creare la prossima generazione di nanosistemi autoregolati, le scoperte degli scienziati fanno anche luce su come gli assemblaggi biomolecolari naturali potrebbero aver acquisito la loro dinamica ottimale.
"Una strategia di evoluzione molecolare ben nota degli organismi viventi è la fusione genetica, in cui il DNA che codifica per due domini proteici interagenti viene fuso in modo casuale", ha affermato Vallée-Bélisle.
"I nostri risultati forniscono anche la comprensione fondamentale necessaria per comprendere come una semplice variazione nella lunghezza del linker tra le proteine fuse possa aver creato in modo efficiente assemblaggi biologici che mostrano una varietà di dinamiche, alcune più adatte di altre per fornire un vantaggio agli organismi viventi." /P>
Ulteriori informazioni: Dominic Lauzon et al, Principi di progettazione e termodinamica per programmare la cooperazione degli assiemi molecolari, Angewandte Chemie International Edition (2023). DOI:10.1002/anie.202313944
Informazioni sul giornale: Edizione Internazionale Angewandte Chemie , Angewandte Chemie
Fornito dall'Università di Montreal