
Ogni subunità proteica è di colore diverso. Il dominio V1 è in alto, Vo in basso, gambi periferici sinistro e destro. Lo sfondo mostra la pompa dell'acqua azionata dal vento. Credito:IST Austria, 2019
Le cellule si affidano a complessi proteici noti come ATP sintasi o ATPasi per il loro fabbisogno energetico. Le molecole di adenosina trifosfato (ATP) alimentano la maggior parte dei processi che sostengono la vita. Il biologo strutturale Professor Leonid Sazanov e il suo gruppo di ricerca dell'Institute of Science and Technology Austria (IST Austria) a Klosterneuburg, L'Austria ha ora determinato la prima struttura atomica del rappresentante della famiglia V/A-ATPasi, colmare il vuoto nell'albero evolutivo di queste macchine molecolari essenziali. Questi risultati ottenuti utilizzando i più recenti metodi di microscopia crioelettronica hanno rivelato una struttura simile a turbina o mulino ad acqua dell'enzima e sono stati ora pubblicati sulla rivista Scienza .
Potenza rotativa
ATP sintasi/ATPasi sono grandi complessi proteici di membrana che condividono piani complessivi di costruzione lorda e meccanismi di catalisi rotativa. Questa famiglia di proteine comprende enzimi di tipo F che si trovano nei mitocondri (fabbriche energetiche della cellula), cloroplasti (organelli che conducono la fotosintesi nelle piante) e batteri; Il tipo V (vacuolare) si trova nei compartimenti intracellulari degli eucarioti (organismi superiori con un nucleo) e il tipo A (archeo) si trova nei procarioti:archaea (antichi microrganismi) e alcuni batteri.
Diversi gusti di ATPasi
Gli enzimi di tipo F e A di solito funzionano per produrre ATP, guidato dal flusso di protoni attraverso la membrana. Gli enzimi di tipo V di solito funzionano al contrario, utilizzando l'ATP per pompare protoni. V- e A-ATPasi sono simili strutturalmente ma differiscono dal tipo F per avere due o tre gambi periferici e subunità proteiche aggiuntive di collegamento tra V1 e Vo. Gli enzimi di tipo V probabilmente si sono evoluti dal tipo A e, a causa di queste somiglianze, il tipo A è anche chiamato V/A-ATPasi. Alcuni batteri, Compreso Thermus thermophilus , acquisito un enzima di tipo A. Zhou lungo, postdoc nel gruppo di ricerca Sazanov di IST Austria, ha purificato e studiato questo enzima (ThV1Vo) mediante crio-EM. A differenza del tipo F, per le ATPasi di tipo V solo le strutture dei domini V1 e Vo isolati sono state determinate in precedenza. Non si sapeva quindi come V1 fosse accoppiato a Vo, e mancava la conoscenza dell'intero ciclo catalitico.
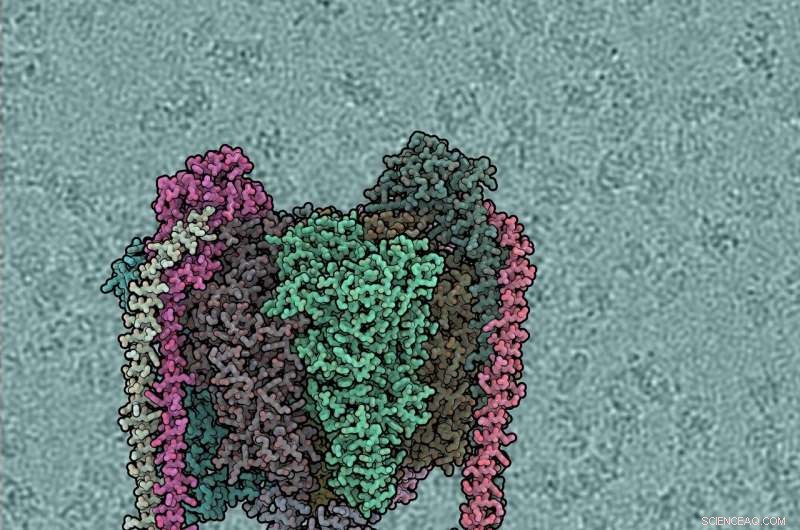
Ogni subunità proteica è di colore diverso. Il dominio V1 è in alto, Vo in basso, gambi periferici sinistro e destro. Lo sfondo mostra la micrografia crio-EM grezza, con singole molecole di ATPasi visibili. Credito:IST Austria, 2019
Plasticità e competizione
Gli scienziati non hanno determinato uno, ma in totale cinque strutture dell'intero enzima ThV1Vo, utilizzando metodi di crio-microscopia elettronica sviluppati di recente nella cosiddetta "rivoluzione della risoluzione" di questa tecnica. Le strutture rappresentano diversi stati conformazionali dell'enzima che differiscono per la posizione del rotore all'interno dello statore. La plasticità conformazionale globale di ThV1Vo si rivela come sostanziale oscillazione di V1 nello spazio in transizione da uno stato all'altro. È il risultato della competizione meccanica tra la rotazione del rotore centrale piegato e la rigidità dello statore. L'accoppiamento V1-Vo si ottiene tramite una stretta corrispondenza strutturale ed elettrostatica tra l'albero e la subunità specifica di tipo V che la collega all'anello a C. La visualizzazione del percorso del protone ha rivelato differenze significative nella distribuzione dei residui proteici carichi da quella nelle F-ATPasi, con un "check-point" più rigoroso che impedisce lo "scivolamento" dell'enzima.
Perché complessità aggiuntiva?
Invece di un singolo gambo periferico di enzimi di tipo F, I tipi A come ThV1Vo hanno due gambi periferici, mentre i tipi V eucariotici ne hanno tre. Ma qual è il vantaggio della complessità aggiuntiva nell'assemblaggio proteico già molto grande, insieme a ulteriori subunità che collegano V1 e Vo? Il dominio F1/V1 ha una simmetria tripla e quindi viene prodotta (o consumata) una molecola di ATP per ogni rotazione di 120° dello statore all'interno di F1/V1. Il professor Leonid Sazanov afferma:"In V/A-ATPases questo passaggio è una rotazione una tantum di 120°, in contrasto con l'F-ATP sintasi dove è diviso in diversi passaggi secondari. Così, può essere necessaria una maggiore plasticità in ThV1Vo per collegare questi passaggi di 120° in V1 a passaggi di subunità per c più piccoli nell'anello c12 di Vo. Questa flessibilità aggiuntiva può essere fornita nei tipi V dagli steli periferici aggiuntivi e dalle subunità di collegamento. Le nostre nuove strutture mostrano come questo si ottiene, fornendo un quadro per l'intera famiglia V-ATPasi".