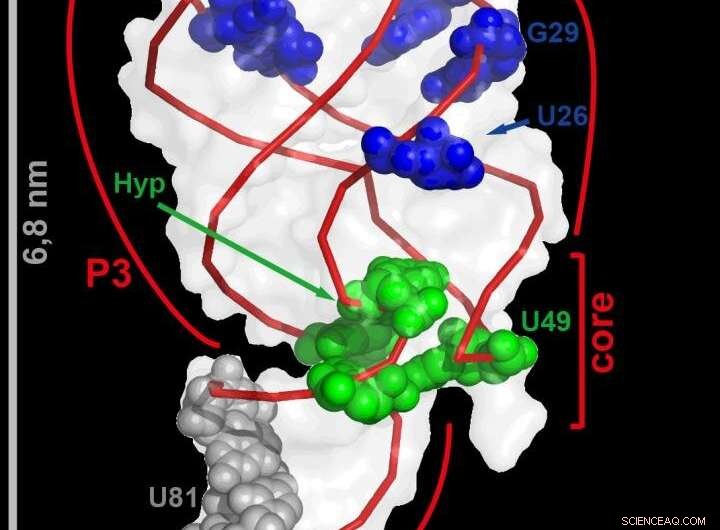
I ricercatori di Francoforte hanno seguito i movimenti di questa minuscola molecola, appena due millesimi dello spessore di un pezzo di carta. L'aptamero dell'RNA cambia la sua struttura quando si lega all'ipoxantina. Le basi azotate verdi cambiano forma particolarmente rapidamente, quelli colorati di blu più lentamente. Le regioni grigie non cambiano. Credito:Università Goethe
Allo stesso modo in cui un singolo pezzo di un puzzle si inserisce nel tutto, la molecola ipoxantina si lega a una catena di acido ribonucleico (RNA), che poi cambia la sua forma tridimensionale in un secondo e così facendo innesca nuovi processi nella cellula. Grazie a un metodo migliorato, i ricercatori sono ora in grado di seguire cambiamenti strutturali quasi inconcepibilmente piccoli nelle cellule mentre progrediscono, sia in termini di tempo che di spazio. Il gruppo di ricerca guidato dal professor Harald Schwalbe del Center for Biomolecular Magnetic Resonance (BMRZ) dell'Università di Goethe è riuscito, insieme a ricercatori israeliani, nell'accelerare di centomila volte il metodo della risonanza magnetica nucleare (NMR) per studiare l'RNA.
"Questo ci permette per la prima volta di seguire la dinamica dei cambiamenti strutturali nell'RNA alla stessa velocità con cui si verificano nella cellula, "dice Schwalbe, descrivendo questa scoperta scientifica, e sottolinea:"Il team guidato da Lucio Frydman dell'Istituto Weizmann in Israele ha dato un contributo importante qui".
I nuovi tipi di esperimenti NMR utilizzano molecole d'acqua i cui atomi possono essere seguiti in un campo magnetico. Schwalbe e il suo team producono acqua iperpolarizzata. Fare così, aggiungono un composto all'acqua che ha radicali elettronici permanentemente spaiati. Gli elettroni possono essere allineati nel campo magnetico mediante eccitazione con microonde a -271°C. Questo allineamento innaturale produce una polarizzazione che viene trasferita a +36°C alla polarizzazione degli atomi di idrogeno utilizzati nell'NMR. Le molecole d'acqua così polarizzate vengono riscaldate in pochi millisecondi e trasferite, insieme all'ipoxantina, alla catena dell'RNA. Il nuovo approccio può in generale essere applicato per osservare reazioni chimiche veloci e cambiamenti di ripiegamento nelle biomolecole a livello atomico.
In particolare i gruppi immino nell'RNA possono essere analizzati da vicino utilizzando questo metodo. In questo modo, i ricercatori sono stati in grado di misurare i cambiamenti strutturali nell'RNA in modo molto accurato. Hanno seguito un piccolo pezzo di RNA da Bacillus subtilis, che cambia la sua struttura durante il legame con l'ipoxantina. Questo cambiamento strutturale fa parte della regolazione del processo di trascrizione, in cui l'RNA viene prodotto dal DNA. Tali piccoli cambiamenti a livello molecolare guidano un gran numero di processi non solo nei batteri ma anche negli organismi multicellulari e persino nell'uomo.
Questo metodo migliorato consentirà in futuro di seguire il ripiegamento dell'RNA in tempo reale, anche se richiede meno di un secondo. Questo è possibile in condizioni fisiologiche, questo è, in ambiente liquido e con concentrazione di molecole naturali a temperature intorno ai 36 °C. "Il prossimo passo sarà ora non solo studiare i singoli RNA ma centinaia di essi, al fine di identificare le differenze biologicamente importanti nei loro tassi di ripiegamento, " afferma Boris Fürtig del gruppo di ricerca di Schwalbe.