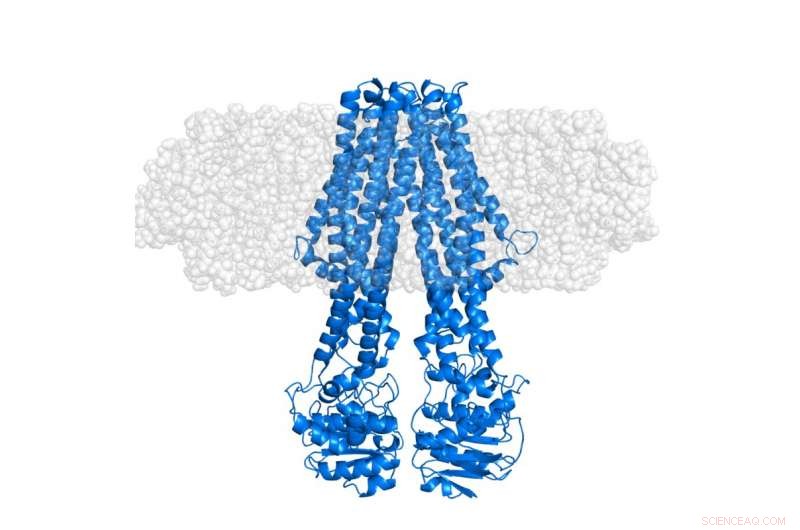
La proteina integrale di membrana MsbA (blu) incorporata in un nanodisco membranoso "semitrasparente" (grigio). Il nanodisco stesso è, attraverso la deuterazione selettiva, reso invisibile negli esperimenti SANS che si concentrano sulla conformazione della proteina nel suo contesto nativo. Credito:Institut Laue-Langevin
Le membrane che circondano le nostre cellule contengono un gran numero di proteine. Le proteine di membrana sono quindi una classe cruciale di macromolecole nei sistemi viventi. Svolgono ruoli chiave, come fornire gateway di trasporto dentro e fuori la cella, facilitando la segnalazione tra le cellule, oltre ad essere coinvolto nella catalisi enzimatica. Questi ruoli funzionali li rendono particolarmente importanti come bersagli farmacologici, con la maggior parte delle attuali terapie mirate alle proteine di membrana.
Però, gli studi strutturali delle proteine integrali di membrana (IMP) si sono rivelati estremamente impegnativi, poiché la maggior parte di essi è difficile da studiare correttamente in assenza del loro ambiente lipidico. Questo spesso impedisce loro di essere cristallizzati, un metodo comunemente usato nella biologia strutturale classica. Sono quindi necessari approcci alternativi per studi strutturali di IMP in ambienti membranosi. Per questo scopo, il Life Sciences Group dell'Institut Laue-Langevin (ILL), in collaborazione con l'Università di Copenaghen, ha aperto la strada con successo allo sviluppo di nanodischi carrier stealth. In questo approccio, viene utilizzato un sofisticato metodo di etichettatura del deuterio per rendere la membrana effettivamente invisibile alla diffrazione di neutroni a bassa risoluzione pur evidenziando la struttura degli IMP all'interno del loro consueto ambiente lipidico, come pubblicato in Acta Crystallographica D nel 2014.
Più recentemente, è stato appena completato il primo studio strutturale di una proteina integrale di membrana che utilizza questa strategia di deuterazione dei nanodischi vettore invisibile. Ciò è stato effettuato utilizzando la piattaforma Deuteration Laboratory (D-Lab) della Partnership for Structural Biology (PSB) in combinazione con lo scattering di neutroni a piccolo angolo (SANS) e lo scattering di raggi X (SAXS) forniti attraverso la piattaforma PSB SANS/SAXS . Come pubblicato sulla rivista Struttura di Josts et al, la squadra internazionale, guidato da Henning Tidow, Università di Amburgo, applicato questo metodo a una proteina trasportatrice ATP-binding cassette (ABC), MsbA – che svolge un ruolo importante nel trasporto dei lipidi nei batteri. I dati di scattering dei neutroni risultanti, per lo più acquisito utilizzando lo strumento D11 presso ILL, ha permesso l'osservazione diretta del segnale dalla proteina di membrana solubilizzata senza contributo dal lipide circostante. I dati SAXS hanno fornito un chiaro riferimento per la forma esterna del nanodisco, comprensivo del doppio strato lipidico.
Inoltre, sono stati studiati i cambiamenti conformazionali in MsbA, dimostrando la sensibilità del metodo e la sua applicabilità generale agli studi strutturali dei PIM.
È probabile che questo approccio diventi sempre più importante negli studi futuri di questi difficili, ma di fondamentale importanza, macromolecole biologiche, a sua volta supportando una migliore comprensione per lo sviluppo di farmaci mirati alle proteine di membrana.