In uno studio recentemente pubblicato sulla rivista Small , i ricercatori del Nano Life Science Institute (WPI NanoLSI), Università di Kanazawa, Giappone, in collaborazione con il professor Sarikaya, Seattle, Stati Uniti, hanno utilizzato la microscopia a forza atomica modulata in frequenza per rivelare l'architettura molecolare di peptidi geneticamente progettati e con mutazioni puntiformi e le loro auto-organizzazioni ciascuno formando cristalli biologici distinti e spessi a molecola singola su grafite atomicamente piatta e MoS2 superfici, offrendo una potenziale piattaforma per tecnologie ibride come la bioelettronica, i biosensori e gli array di proteine.
Le proteine sono i mattoni molecolari chiave della biologia, svolgono funzioni enzimatiche, trasportano ioni ed elettroni e costituiscono la principale infrastruttura delle architetture cellulari, come le pompe ioniche. Le funzioni delle proteine dipendono dalle loro sequenze di amminoacidi, che, a loro volta, ne determinano le strutture molecolari tridimensionali.
Le sequenze aminoacidiche delle proteine sono codificate dai geni (codice DNA) di una determinata cellula specializzata. Si prevede quindi che qualsiasi difetto nella sequenza aminoacidica, come una mutazione puntiforme, un cambiamento nella posizione di un amminoacido, un amminoacido mancante o la trasposizione di due o più amminoacidi, influenzi la struttura molecolare della proteina , che si riflette nella loro funzione, portando spesso a malattie genetiche.
Similmente alle proteine, anche i peptidi sono composti da unità di amminoacidi e ciascun peptide ha una sequenza unica; tuttavia, sono molto più brevi, variano da 10 a 30 unità, e sono codificati in modo simile dal DNA. Sono più facili da manipolare, progettare e utilizzare rispetto alle proteine, che sono molto più grandi (lunghe centinaia o migliaia di amminoacidi), difficili da sintetizzare e poco pratiche da utilizzare.
Pertanto, è imperativo che se si vuole sviluppare la tecnologia pratica del futuro sulla base delle lezioni della biologia su scala molecolare, la scienza fondamentale deve essere stabilita utilizzando peptidi, che possono poi essere implementati in applicazioni del mondo reale, come i biosensori , bioelettronica e dispositivi logici, nonché farmaci, tutti basati su lezioni di biologia.
Realizzando questa premessa, il professor Sarikaya ha avviato un nuovo approccio scientifico di convergenza due decenni fa, coinvolgendo ingegneria genetica, biologia molecolare, informatica, apprendimento automatico e scienza e ingegneria dei materiali, nella progettazione di peptidi leganti solidi e ne ha dimostrato l'uso in tecnologie ibride pratiche. /P>
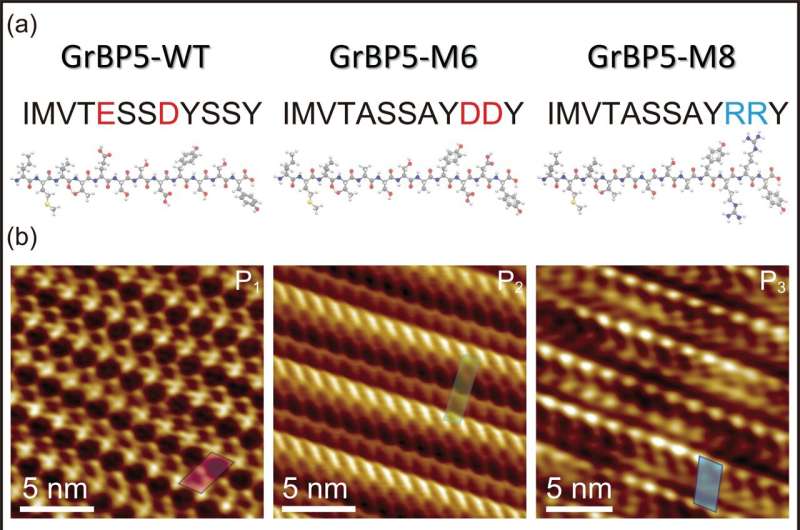
In questo nuovo studio, uno di questi peptidi, specificamente progettato come peptide legante la grafite, è mutato in due diversi peptidi, M6 e M8, ciascuno nominalmente dello stesso peso molecolare e dimensione ma contenente mutazioni puntiformi degli amminoacidi carichi, una negativa e l'altro positivo, rispettivamente.
Come previsto, avrebbero funzionato diversamente, e così è stato. Tuttavia, la questione impossibile da prevedere a priori era come esattamente avrebbero agito in modo diverso l'uno dall'altro e dal peptide wild type (l'originale), e quali sarebbero i dettagli della loro struttura molecolare e del loro comportamento cinetico sulla superficie del peptide. substrato di grafite.
Le risposte a queste domande, la cui mancanza finora limitava la comprensione della relazione tra mutanti e del loro comportamento prevedibile, richiedevano una risoluzione molto elevata e l'imaging diretto delle molecole nel loro stato nativo, nell'acqua per un periodo che va da secondi a ore. /P>
È qui che entra in gioco il laboratorio del professor Takeshi Fukuma presso il WPI Nano Life Science Institute, Università di Kanazawa, dove il suo team ha sviluppato strutture di microscopia a forza atomica modulata in frequenza in grado di interrogare oggetti su scala molecolare con una risoluzione dell'immagine vicina all'angstrom e per lunghi periodi di tempo all'interno di ambienti acquosi:una combinazione insolita di capacità per un sistema sperimentale, in particolare per la ricerca biologica.
Grazie alle capacità sperimentali altamente competenti dei ricercatori (con inesauribile pazienza nei test di lunga durata), guidati dal Dr. Ayhan Yurtsever, dal Dr. Kaito Hirata, dal Dr. Linhao Sun e dal Dr. Takeshi Fukuma, il team ha scoperto il comportamento dei mutanti con un dettaglio senza precedenti, fornendo risultati con un significato che attraversa diverse discipline tradizionalmente diverse, dalla scienza dei materiali alla fisica, alla chimica e alla biologia.
In primo luogo, nonostante le mutazioni e avendo individualmente una carica complessiva diversa, ciascun peptide non solo si legava alla superficie della grafite ma si assemblava anche, formando cristalli peptidici dello spessore di una singola molecola. Tuttavia, mentre il primo mutante, quello caricato negativamente, formava un reticolo cristallino obliquo con due molecole (avvolte l'una attorno all'altra) in ciascun reticolo, il secondo mutante, quello caricato positivamente, formava un diverso reticolo obliquo contenente un singolo peptide. /P>
Ciò che è veramente prezioso in questo caso è che il comportamento dei peptidi è semplicemente e direttamente correlato alla loro architettura molecolare, modellata utilizzando la dinamica molecolare. I ricercatori, quindi, sono stati in grado di descrivere matematicamente la relazione tra ciascuno dei peptidi (ciascuno che forma un diverso reticolo molecolare obliquo) e il reticolo esagonale del substrato di grafite, che è descritto esclusivamente come riconoscimento chirale.
In effetti, ciò significa che è dimostrato che ciascun peptide forma un'interfaccia ibrida con il substrato di grafite, meglio descritta come cristallograficamente coerente senza alcun intervallo evidente nel mezzo, non diversamente dalle interazioni ligando-recettore o proteina/DNA in biologia.
Né il riconoscimento molecolare di oggetti allo stato solido da parte di una molecola "vivente" è stato osservato così chiaramente prima, né i suoi dettagli sono stati descritti come chirali, entrambi elementi che accrescono il significato dei risultati presentati in questa pubblicazione.
Da un lato, dal punto di vista biologico, ora ci sono ampi dati e dettagli su come le mutazioni portano a cambiamenti conformazionali molecolari e alla funzione risultante dei peptidi, che possono essere espansi nel comportamento delle proteine, con significato nelle mutazioni genetiche ed efficaci progettazione di farmaci.
E d'altra parte, l'ibridazione coerente suggerisce che oggetti su scala nanometrica animati e inanimati sono stabilmente presenti nella stessa interfaccia morbida, formando un ponte molecolare esclusivo che potrebbe essere meglio descritto integrando perfettamente la biologia con dispositivi a stato solido.
Se è così, sulla base di questo studio, scienziati e tecnologi dovrebbero ampliare enormemente il repertorio delle basi molecolari dei futuri bio/nano dispositivi utilizzando il semplice strumento di biologia molecolare della mutazione.
Ulteriori informazioni: Ayhan Yurtsever et al, Dinamica dell'autoassemblaggio molecolare di peptidi corti su interfacce liquido-solido:effetto delle mutazioni puntiformi degli amminoacidi carichi, Piccolo (2024). DOI:10.1002/piccolo.202400653
Informazioni sul giornale: Piccolo
Fornito dall'Università di Kanazawa