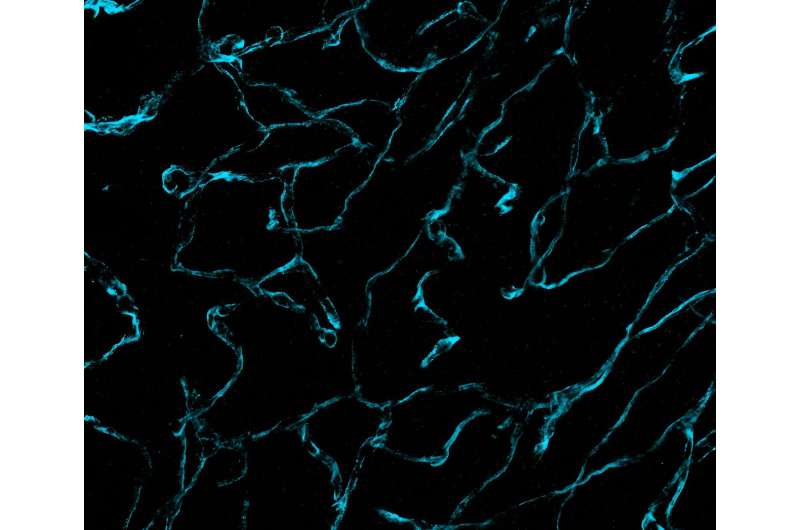
I ricercatori del Brigham and Women's Hospital e del Massachusetts Institute of Technology (MIT) hanno svelato immagini dettagliate senza precedenti del tessuto del cancro al cervello attraverso l'uso di una nuova tecnologia di microscopia chiamata patologia di espansione del decrowding (dExPath). I loro risultati, pubblicati su Science Translational Medicina , forniscono nuove informazioni sullo sviluppo del cancro al cervello, con potenziali implicazioni per il progresso della diagnosi e del trattamento di malattie neurologiche aggressive.
"In passato, ci siamo affidati a microscopi costosi e ad alta risoluzione che solo laboratori molto ben finanziati potevano permettersi, che richiedevano una formazione specializzata per l'utilizzo e sono spesso poco pratici per analisi ad alto rendimento dei tessuti cerebrali a livello molecolare", ha affermato Pablo Valdes, MD, Ph.D., alunno residente in neurochirurgia al Brigham e autore principale dello studio. "Questa tecnologia porta alla clinica un imaging affidabile e ad altissima risoluzione, consentendo agli scienziati di studiare le malattie neurologiche a un livello su scala nanometrica mai raggiunto prima su campioni clinici convenzionali con microscopi convenzionali."
I ricercatori in precedenza si affidavano a microscopi costosi e ad altissima risoluzione per acquisire immagini di strutture su scala nanometrica nelle cellule e nel tessuto cerebrale e, anche con la tecnologia più avanzata, spesso faticavano a catturare efficacemente queste strutture a livello di nanoscala.
Ed Boyden, Ph.D., professore di neurotecnologia Y. Eva Tan al MIT e co-autore senior di questo studio, ha iniziato ad affrontare questo problema etichettando i tessuti e quindi modificandoli chimicamente per consentire un'espansione fisica uniforme dei tessuti. Tuttavia, questa tecnologia di espansione era lungi dall’essere perfetta. Facendo affidamento su enzimi noti come proteasi per rompere i tessuti, gli scienziati hanno scoperto che questo trattamento chimico con enzimi distruggeva le proteine prima che potessero analizzarle, lasciando dietro di sé solo uno scheletro della struttura originale, conservando solo le etichette.
Lavorando insieme, Boyden ed E. Antonio Chiocca, MD, Ph.D., presidente di Neurochirurgia presso il Brigham and Women's Hospital e co-autore senior di questo studio, hanno fatto da mentore a Valdes durante la sua formazione come neurochirurgo-scienziato, per sviluppare nuove sostanze chimiche con dExPath per affrontare i limiti della tecnologia di espansione originale.
La loro nuova tecnologia modifica chimicamente i tessuti incorporandoli in un gel e "ammorbidendo" i tessuti con uno speciale trattamento chimico che separa le strutture proteiche senza distruggerle e che consente ai tessuti di espandersi. Ciò ha fornito risultati entusiasmanti ai ricercatori del MIT e di Brigham, che utilizzano abitualmente anticorpi disponibili in commercio per legarsi e illuminare i biomarcatori in un campione.
Gli anticorpi, tuttavia, sono grandi e molte volte non riescono a penetrare facilmente le strutture cellulari per raggiungere il loro bersaglio. Ora, separando le proteine con dExPath, questi stessi anticorpi utilizzati per la colorazione possono penetrare negli spazi per legare le proteine nei tessuti a cui non era possibile accedere prima dell'espansione, evidenziando strutture di dimensioni nanometriche o persino popolazioni cellulari precedentemente nascoste.
"Il cervello umano è dotato di diverse protezioni per proteggersi dagli agenti patogeni e dalle tossine ambientali. Ma questi elementi rendono difficile lo studio dell'attività cerebrale. Può essere un po' come guidare un'auto nel fango e nei fossati. Non possiamo accedere a determinate strutture cellulari nel cervello a causa delle barriere che si frappongono," ha affermato E. Antonio Chiocca, MD, Ph.D., presidente del Dipartimento di Neurochirurgia del Brigham. "Questo è solo uno dei motivi per cui questa nuova tecnologia potrebbe cambiare radicalmente la pratica. Se riusciamo a acquisire immagini più dettagliate e accurate del tessuto cerebrale, possiamo identificare più biomarcatori ed essere meglio attrezzati per diagnosticare e trattare malattie cerebrali aggressive."
Per convalidare l'efficacia di dExPath, il team di Boyden e Chiocca ha applicato la tecnologia al tessuto cerebrale umano sano, ai tessuti con cancro al cervello di alto e basso grado e ai tessuti cerebrali affetti da malattie neurodegenerative tra cui l'Alzheimer e il morbo di Parkinson. I ricercatori hanno colorato i tessuti per individuare biomarcatori specifici del cervello e della malattia e hanno acquisito immagini prima e dopo l'espansione dei campioni con dExPath.
I risultati hanno rivelato un'espansione uniforme e coerente del tessuto senza distorsioni, consentendo un'analisi accurata delle strutture proteiche. Inoltre, dExPath ha eliminato efficacemente i segnali fluorescenti nel tessuto cerebrale chiamato lipofuscina, il che rende molto difficile l’imaging delle strutture subcellulari nei tessuti cerebrali, migliorando ulteriormente la qualità dell’immagine. Inoltre, dExPath ha fornito segnali fluorescenti più forti per una migliore etichettatura nonché l'etichettatura simultanea di un massimo di 16 biomarcatori nello stesso campione di tessuto. In particolare, l'imaging dExPath ha rivelato che i tumori precedentemente classificati come "di basso grado" contenevano caratteristiche e popolazioni cellulari più aggressive, suggerendo che il tumore potrebbe diventare molto più pericoloso del previsto.
Sebbene promettente, dExPath richiede la convalida su campioni di dimensioni più ampie prima di poter contribuire alla diagnosi di condizioni neurologiche come il cancro al cervello. Valdes sottolinea che, sebbene sia ancora nelle fasi iniziali, il suo team aspira a che questa tecnologia possa eventualmente fungere da strumento diagnostico, migliorando in definitiva i risultati per i pazienti.
"Speriamo che con questa tecnologia possiamo comprendere meglio, a livello nanometrico, il complesso funzionamento dei tumori cerebrali e le loro interazioni con il sistema nervoso senza dipendere da attrezzature di laboratorio esorbitantemente costose", ha affermato Valdes, ora assistente professore di neurochirurgia e Jennie. Sealy Distinguished Chair in Neuroscienze presso la facoltà di medicina dell'Università del Texas.
"L'accessibilità di dExPath consentirà l'imaging a super risoluzione per comprendere l'elaborazione biologica a livello nanometrico nei tessuti umani in neuro-oncologia e nelle malattie neurologiche come l'Alzheimer e il Parkinson e, un giorno, potrebbe persino migliorare le strategie diagnostiche e gli esiti dei pazienti. "
Gli autori sono Pablo Valdes (BWH e MIT), Chih-Chieh (Jay) Yu, Jenna Aronson, Debarati Ghosh, Yongxin Zhao, Bobae An (MIT), Joshua D. Bernstock (BWH e MIT), Deepak Bhere (BWH), Michelle M. Felicella, Mariano S. Viapiano, Khalid Shah (BWH) e gli autori senior co-corrispondenti E. Antonio Chiocca e Edward S. Boyden.
Ulteriori informazioni: Pablo Valdes et al, Miglioramento dell'immunocolorazione di nanostrutture e cellule in campioni di cervello umano attraverso il deaffollamento proteico mediato dall'espansione, Science Translational Medicine (2024). DOI:10.1126/scitranslmed.abo0049
Informazioni sul giornale: Scienza Medicina Traslazionale
Fornito da Brigham and Women's Hospital