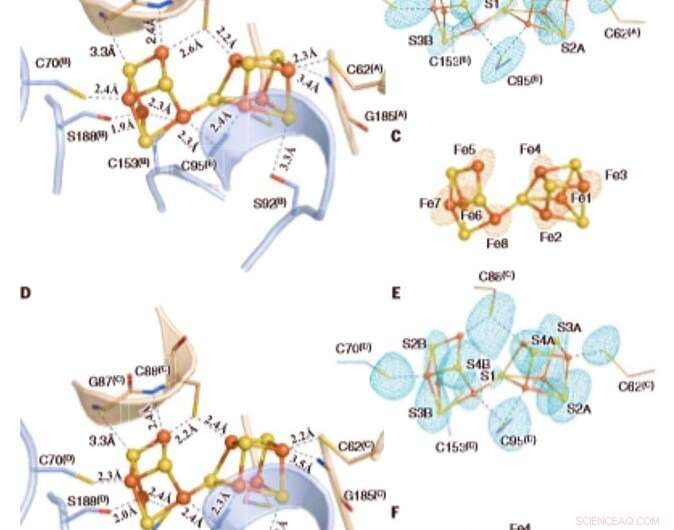
I P-cluster ossidati in Av1*. Strutture di P-cluster in (A to C) l'interfaccia delle catene A e B [P-cluster(A/B)] e (D a F) l'interfaccia delle catene C e D [P-cluster(C/D) ] di Av1*. Le catene A e C sono le subunità a, e le catene B e D sono le subunità b dei due dimeri ab di Av1*. [(A) e (D)] I cluster P sono mostrati in una presentazione con palla e bastone, e i residui chiave che interagiscono con i cluster P sono indicati come bastoncini. Le catene A e C sono mostrate come nastri di grano, e le catene B e D sono mostrate come nastri azzurri. [(B) e (C)] P-cluster(A/B) e [(E) e (F)] P-cluster(C/D) sovrapposti a [(B) e (E)] le mappe di densità anomala calcolato a 7100 eV con una risoluzione di 2,18 Å e contornato a 4,0 s, mostra la posizione degli atomi di zolfo (maglia blu menta); e con [(C) e (F)] le mappe di densità anomala calcolate a 7141 eV con una risoluzione di 2,1 e contornate a 15,0 s, che mostra la posizione degli atomi di ferro (maglia rossa). Gli atomi sono colorati come segue:Fe, arancia; S, giallo; Oh, rosso; N, blu. Le abbreviazioni a lettera singola per i residui di amminoacidi sono le seguenti:C, Cis; G, gli; H, Il suo; R, Argo; S, Ser. Credito:Scienza, doi:10.1126/science.aaz6748
L'enzima nitrogenasi è un catalizzatore biologico in grado di ridurre il diazoto (N2) ad ammoniaca in presenza di una serie di metallocofattori complessi. Però, i dettagli meccanicistici della reazione rimangono scarsi. In un nuovo rapporto su Scienza , Wonchull Kang e un gruppo di ricerca in chimica, biologia molecolare e biochimica presso l'Università della California-Irvine, NOI., hanno riportato una struttura cristallina di 1,83 angstrom per la proteina nitrogenasi molibdeno-ferro (MoFe), che hanno catturato in condizioni fisiologiche di ricambio dell'azoto. I risultati dello studio possono valutare i possibili meccanismi di N 2 riduzione e il ruolo dei siti di cintura-zolfo durante il processo.
La nitrogenasi è un catalizzatore per un passaggio critico nel ciclo globale dell'azoto, durante la riduzione ambientale del diazoto atmosferico (N 2 ) all'ammoniaca biodisponibile (NH 3 ). L'enzima molibdeno nitrogenasi contiene due componenti proteici:uno contenente la proteina ferro (Fe) in un ferro zolfo (Fe 4 S 4 ) cluster con un sito di legame dell'adenosina trifosfato (ATP) all'interno di ciascuna subunità. L'altro, proteina di ferro molibdeno (MoFe) contenente un α 2 ? 2 eterotetramero con due metallocluster complessi. Durante la catalisi della molibdeno-nitrogenasi (Mo-nitrogenasi), l'associazione e la dissociazione ripetute tra i due componenti proteici consentivano il trasferimento di elettroni ATP-dipendente dal Fe 4 S 4 cluster alla proteina MoFe per la riduzione del substrato. La capacità della nitrogenasi di trasportare molti elettroni al suo cofattore ha reso l'enzima altamente versatile durante la riduzione del substrato.
Comprensione del meccanismo d'azione dell'enzima nitrogenasi
Molti sforzi sono stati fatti per comprendere i meccanismi della nitrogenasi dalla sua scoperta, dove alcuni si erano concentrati sulle interazioni del substrato e dell'inibitore dell'enzima. Di questi sforzi, Kang et al. determinato una strategia degna di considerazione limitando le forniture di elettroni in eccesso che inavvertitamente guidavano l'N 2 processo di riduzione in avanti. Ciò ha riportato il substrato o lo stato legato intermedio dell'enzima a uno stato di riposo o ha ridotto l'enzima a uno stato misto indistinguibile. Il processo è stato rilevante poiché le proteine nitrogenasi sono normalmente isolate in presenza di un eccesso di ditionito come riducente fornito dall'esterno, e la rimozione di questa sorgente di elettroni artificiale in assenza di ossigeno potrebbe aiutare gli scienziati a catturare il diazoto (N 2 ) o dei suoi intermedi per l'analisi.
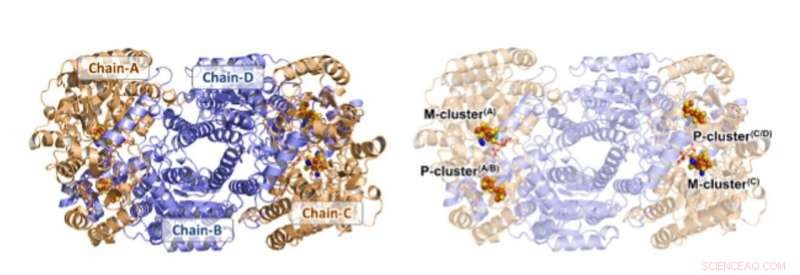
Struttura di Av1*. Presentazioni a nastro solido (a sinistra) e trasparente (a destra) dell'eterotetramero di Av1*, con le subunità a (Catena-A e Catena-C) e le subunità b (Catena-B e Catena-D) colorate in grano e blu, rispettivamente. I cluster M e P sono illustrati come modelli che riempiono lo spazio. Codice colore degli atomi:Fe, arancia; S, giallo; Oh, rosso; N, blu; Mo, ciano; C, grigio. Gli M-cluster in Chain-A e Chain-C sono designati M-cluster(A) e M-cluster(C), rispettivamente. I P-cluster alle interfacce Chain-A/Chain-B e Chain-C/Chain-D sono designati P-cluster(A/B) e P-cluster(C/D), rispettivamente. PYMOL è stato utilizzato per preparare questa figura. Credito:Scienza, doi:10.1126/science.aaz6748
Come prova del concetto, Kang et al. preparato l'estratto grezzo di un ceppo batterico anerobico Azotobacter vinelandii con o senza l'aggiunta di ditionito dopo la distruzione cellulare. Il ceppo di A. vinelandii esprimeva attivamente una Mo-nitrogenasi contenente una proteina MoFe marcata con istidina in entrambi i casi. Quando hanno analizzato l'attività di questi campioni, i campioni di estratto grezzo senza ditionite erano quasi inattivi durante la riduzione del substrato, a causa dell'esaurimento degli elettroni negli estratti grezzi durante la distruzione cellulare. Kang et al. potrebbe quindi ripristinare completamente l'attività dei campioni aggiungendo ditionite e ATP (ovvero fornendo elettroni).
Cluster di nitrogenasi-due metallocluster unici:il P-cluster e il M-cluster.
Sulla base delle condizioni descritte, quando una coltura che esprime azotosi che svolge attivamente N 2 la fissazione è soggetta a lisi cellulare senza ulteriori forniture di elettroni, la nitrogenasi è rimasta funzionale. Sebbene potenzialmente arrestato in uno stato "dormiente" o legato intermedio a causa del ritiro del flusso di elettroni in un metallocluster ferro-zolfo noto come M-cluster, situato all'interno dell'enzima nitrogenasi. Quando Kang et al. purificato l'estratto grezzo senza ditionite, la proteina MoFe marcata con istidina (designata come AV1* nello studio) era attiva durante N 2 riduzione e anche perfettamente funzionante. Quando il team ha cristallizzato AV1*, hanno osservato cristalli marroni che diffrangevano a una risoluzione di 1,83 angstrom (Å). Hanno confermato il riarrangiamento strutturale dei due cluster P di AV1* utilizzando dati di densità anomala e hanno utilizzato la risonanza paramagnetica elettronica per osservare l'assegnazione strutturale. I risultati hanno fornito risposte a lungo ricercate sulla rilevanza fisiologica di questo stato sperimentale e hanno indicato un flusso limitato di elettroni tra i due cluster metallici unici (cluster P e M) del composto in assenza di ditionite.
Un meccanismo plausibile di riduzione di N2 da parte della Mo-nitrogenasi. Credito:Markus Ribbe e Yilin Hu, Scienza , doi:10.1126/science.aaz6748 
Un meccanismo d'azione plausibile che concordava con le osservazioni sperimentali includeva la riduzione graduale del diazoto (N 2 ) nei tre siti di zolfo della cintura sul catalizzatore azotato basato sulla rotazione del cluster M. Il meccanismo proposto inizia con un legame stretto di N 2 in un sito specifico, seguito dalla rotazione del legato N 2 ad un altro sito successivo (siti designati come da S3A a S2B a S5A sul composto). Durante il processo, la riduzione/protonazione di N 2 al livello di diazene si è verificato attraverso il legame idrogeno, seguita da un'ulteriore riduzione/protonazione per la sua conversione in ammoniaca, prima del suo rilascio dalla struttura. La successiva rotazione del cluster ha portato un nuovo N 2 molecola al sito successivo per iniziare il prossimo ciclo di N . graduale 2 riduzione attraverso la rotazione continua dei cluster in un delicato meccanismo durante la catalisi. Tali cicli tra diversi siti di reazione erano vagamente analoghi al meccanismo dell'enzima ATP sintasi. Il metallocluster rotante ha quindi permesso efficacemente la riduzione multielettronica di N 2 attraverso un approccio divide et impera.
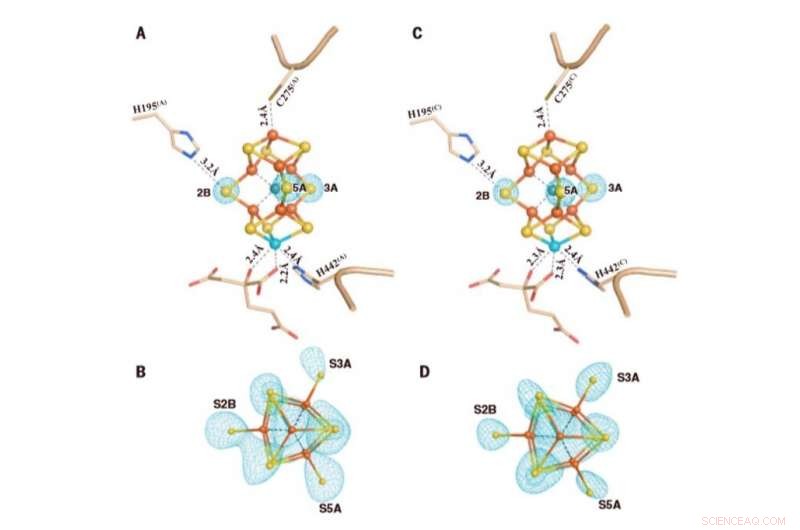
I cluster M in Av1*(TOD). Strutture di M-cluster nella (A e B) catena A [M-cluster(A)] e (C e D) catena C [M-cluster(C)] raffinate a una risoluzione di 1,73 . Vista laterale di (A) M-cluster(A) e (C) M-cluster(C) con residui chiave che interagiscono con i cluster indicati come bastoncini. M-cluster(A) e M-cluster(C) sono sovrapposti alle mappe di omissione Fo-Fc degli zolfi della cintura contornate a 13s (maglia blu menta). View along the Fe1-C-Mo direction of (B) M-cluster(A) and (D) M-cluster(C) superimposed with the anomalous density maps calculated at 7100 eV at a resolution of 2.17 Å and contoured at 4.0s, showing the presence of the anomalous sulfur density (mint-blue mesh) at all belt sulfur positions (S2B, S3A, and S5A) in (B) M-cluster(A) and (D) M-cluster(C). Credit:Science, doi:10.1126/science.aaz6748
To understand the sulfur-displaced conformation of AV1* under limited electron flux, the team formed AV1* turnover with dithionite (designated as AV1*TOD), to yield brown crystals that diffracted to a resolution of 1.73 Å. The observations were consistent with the mechanism of bound dinitrogen species on the compound and illustrated the physiological relevance of the conformation during catalysis. The capacity to displace three different sites by a dinitrogen species was consistent with previous investigations on catalysis-dependent selenium. Kang et al. proposed many mechanisms to explain the observations, however they seek further experimental support to verify them. The team highlighted the possibility for all belt-sulfur sites to be involved in the process of catalysis due to the presence of asymmetric belt-sulfur displacements in the compound. The results aim to provoke a paradigm shift in the mechanistic thinking of nitrogenase activity, ultimately to understand the intricate mechanism of the enzyme.
© 2020 Scienza X Rete