
Un modello basato su agenti di risposta immunitaria innata simula meccanicamente la sepsi in 2-D. Credito:Lawrence Livermore National Laboratory
Un approccio di deep learning originariamente progettato per insegnare ai computer come giocare ai videogiochi meglio degli umani potrebbe aiutare nello sviluppo di cure mediche personalizzate per la sepsi, una malattia che provoca circa 300, 000 decessi all'anno e per i quali non esiste una cura nota.
Lawrence Livermore National Laboratory (LLNL), in collaborazione con ricercatori dell'Università del Vermont, sta esplorando il modo in cui l'apprendimento per rinforzo profondo può scoprire strategie farmacologiche terapeutiche per la sepsi utilizzando una simulazione del sistema immunitario innato di un paziente come piattaforma per esperimenti virtuali. L'apprendimento per rinforzo profondo è un approccio di apprendimento automatico all'avanguardia originariamente sviluppato da Google DeepMind per insegnare a una rete neurale come giocare ai videogiochi, dati solo pixel come input e il punteggio del gioco come segnale di apprendimento. Gli algoritmi spesso superano le prestazioni umane, nonostante non sia stata data alcuna conoscenza delle meccaniche del gioco.
L'approccio di deep learning di LLNL tratta la simulazione del sistema immunitario sviluppata dai suoi collaboratori come un videogioco. Utilizzando gli output della simulazione, un "punteggio" basato sulla salute del paziente e un algoritmo di ottimizzazione, la rete neurale impara a manipolare 12 diversi mediatori di citochine, regolatori del sistema immunitario, per riportare la risposta immunitaria all'infezione a livelli normali. La ricerca appare in un articolo pubblicato dalla International Conference on Machine Learning.
"È un sistema complesso, " ha affermato il ricercatore LLNL Dan Faissol, ricercatore principale del progetto. "Finora le indagini precedenti si sono basate sulla manipolazione di un singolo mediatore/citochina, generalmente somministrato con una singola dose o in un ciclo molto breve. Crediamo che il nostro approccio abbia un grande potenziale perché esplora cose molto più complesse, strategie terapeutiche pronte all'uso che trattano ogni paziente in modo diverso in base alle misurazioni del paziente nel tempo".
La strategia di trattamento proposta dai ricercatori è adattiva e personalizzata, migliorarsi su un ciclo di feedback osservando continuamente i livelli di citochine e prescrivendo farmaci specifici per il singolo paziente. Ogni esecuzione della simulazione rappresenta un diverso tipo di paziente e diverse condizioni iniziali di infezione.
"La sfida era mantenere le cose clinicamente rilevanti, " ha spiegato il ricercatore LLNL Brenden Petersen, il responsabile tecnico del progetto. "Dovevamo assicurarci che tutti gli aspetti del problema simulato fossero rilevanti nel mondo reale, che il computer non utilizzasse alcuna informazione che non sarebbe stata realmente disponibile in un ospedale. Quindi, abbiamo fornito alla rete neurale solo informazioni che possono essere effettivamente misurate clinicamente, come i livelli di citochine e la conta cellulare da un prelievo di sangue".
Utilizzando il modello basato su agenti con apprendimento per rinforzo profondo, i ricercatori hanno identificato una politica di trattamento che raggiunge un tasso di sopravvivenza del 100% per i pazienti su cui è stata addestrata, e una mortalità inferiore all'1% su 500 pazienti selezionati casualmente.
"La simulazione è di natura meccanicistica, il che significa che possiamo virtualmente sperimentare farmaci e combinazioni di farmaci che non sono stati testati prima per vedere se potrebbero essere promettenti, "Faissol ha detto. "Il numero di possibili strategie di trattamento è enorme, soprattutto se si considerano strategie multifarmaco che variano nel tempo. Senza usare la simulazione, non c'è modo di valutarli tutti. La parte difficile è scoprire una strategia che funzioni per tutti i tipi di pazienti. L'infezione di ognuno è diversa, e il corpo di ognuno è diverso."
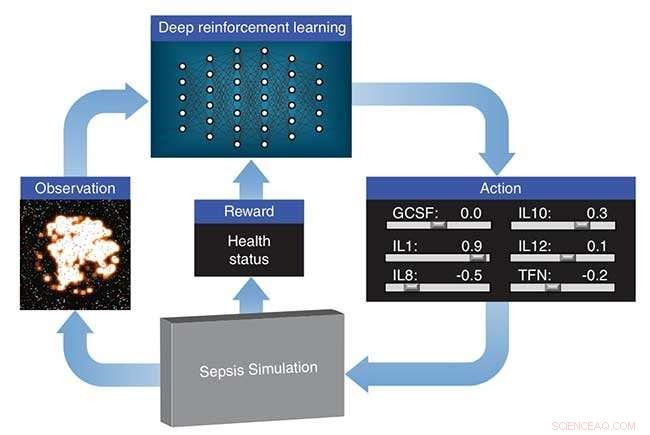
L'approccio di deep learning di LLNL tratta la simulazione del sistema immunitario sviluppata dai suoi collaboratori come un videogioco. Utilizzando gli output della simulazione, un “punteggio” basato sulla salute del paziente e un algoritmo di ottimizzazione, la rete neurale impara a manipolare 12 diversi mediatori di citochine - regolatori del sistema immunitario - per riportare la risposta immunitaria all'infezione a livelli normali. Credito:Lawrence Livermore National Laboratory
La ricerca del team ha dimostrato che questo approccio adattivo può portare a nuove intuizioni, ei ricercatori sperano di convincere gli altri ad adottare l'approccio sulla sepsi e altre malattie.
"Il nostro grande, la visione a lungo termine è un sistema al posto letto "a circuito chiuso" in cui le misurazioni di un paziente vengono inserite in uno strumento di supporto decisionale, che poi somministra i farmaci corretti alle dosi corrette ai tempi giusti, " ha detto Petersen. " Tali strategie di trattamento dovrebbero prima essere controllate e messe a punto in laboratorio umido e modelli animali, eventualmente informare i trattamenti reali."
Petersen ha affermato che la maggior parte dell'hardware per eseguire un tale sistema a ciclo chiuso esiste già, come con sistemi più semplici come le pompe per insulina che monitorano costantemente il sangue e somministrano l'insulina al momento giusto.
L'approccio di apprendimento per rinforzo profondo del laboratorio deve ancora essere testato nel mondo reale, ma in base al successo utilizzando la simulazione, il National Institutes of Health ha assegnato a LLNL e ai ricercatori dell'Università del Vermont una sovvenzione quinquennale per continuare il lavoro, principalmente sulla sepsi ma anche sul cancro.
"Questo è un progetto entusiasmante, " ha detto Gary An, un medico di terapia intensiva presso l'Università del Vermont e scienziato computazionale che ha sviluppato la versione originale della simulazione della sepsi. "Questo è un progetto incredibilmente nuovo che riunisce tre aree all'avanguardia della ricerca computazionale:simulazioni multiscala ad alta risoluzione di processi biologici, l'estensione dell'apprendimento per rinforzo profondo alla ricerca biomedica e l'uso del calcolo ad alte prestazioni per riunire il tutto."
Il direttore di Bioingegneria di LLNL Shankar Sundaram ha descritto l'approccio come "un esempio illustrativo del laboratorio che contribuisce allo sviluppo di una potenziale soluzione terapeutica a un problema di salute complesso critico per la nostra missione di biosicurezza, applicando e facendo progredire le nostre capacità all'avanguardia nell'apprendimento automatico scientifico e mirando a una migliore causalità, comprensione meccanicistica".
I ricercatori LLNL hanno anche avviato una collaborazione con il Moffitt Cancer Center in Florida per vedere se un approccio simile potrebbe apprendere strategie di terapia farmacologica efficaci utilizzando una simulazione del cancro. Moffitt ha rilasciato una versione per videogiochi della loro simulazione chiamata "Cancer Crusade" che funziona sui telefoni cellulari.
"Una strategia è quella di crowdsourcing dell'apprendimento analizzando i trattamenti registrati dai migliori giocatori di tutto il mondo, " ha detto Petersen. "Abbiamo applicato il nostro approccio di apprendimento profondo e vogliamo vedere come i nostri trattamenti computerizzati si accumulano contro i migliori giocatori:una resa dei conti 'uomo contro macchina'".
Il progetto sulla sepsi ha anche portato a un nuovo sforzo presso LLNL nella ricerca di strategie di ciberdifesa adattive e autonome utilizzando la simulazione e l'apprendimento per rinforzo profondo.