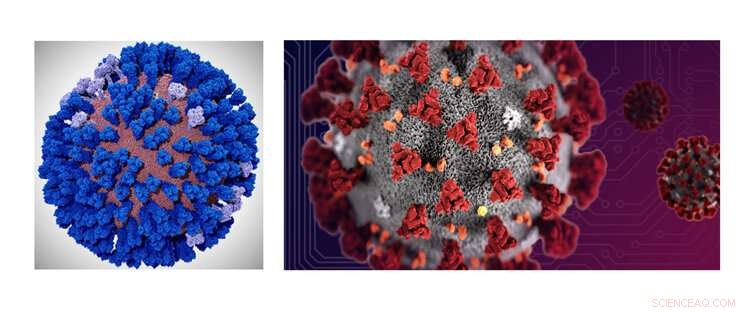
Un modello di computer interamente atomico con busta di coronavirus è stato sviluppato dall'Amaro Lab dell'UC San Diego sul supercomputer Frontera del TACC, finanziato dall'NSF, a UT Austin. La biochimica Rommie Amaro spera di sfruttare il suo recente successo con le simulazioni del virus dell'influenza su tutti gli atomi (a sinistra) e applicarle al coronavirus (a destra). Credito:Lorenzo Casalino (UCSD), TACC
Gli scienziati stanno preparando un enorme modello computerizzato del coronavirus che si aspettano possa fornire informazioni su come infetta il corpo. hanno mosso i primi passi, testare le prime parti del modello e ottimizzare il codice sul supercomputer Frontera presso l'Università del Texas presso il Texas Advanced Computing Center (TACC) di Austin. Le conoscenze acquisite dal modello completo possono aiutare i ricercatori a progettare nuovi farmaci e vaccini per combattere il coronavirus.
Rommie Amaro sta guidando gli sforzi per costruire il primo modello completo di tutti gli atomi dell'involucro del coronavirus SARS-COV-2, la sua componente esterna. "Se abbiamo un buon modello per come appare l'esterno della particella e come si comporta, avremo una buona visione dei diversi componenti coinvolti nel riconoscimento molecolare." Il riconoscimento molecolare riguarda il modo in cui il virus interagisce con i recettori dell'enzima di conversione dell'angiotensina 2 (ACE2) e possibilmente altri bersagli all'interno della membrana della cellula ospite. Amaro è professore di chimica e biochimica all'Università della California, San Diego.
Il modello del coronavirus è previsto da Amaro per contenere circa 200 milioni di atomi, un'impresa ardua, come l'interazione di ogni atomo con l'altro deve essere calcolata. Il flusso di lavoro del suo team prende un ibrido, o approccio di modellizzazione integrativa.
"Stiamo cercando di combinare dati a risoluzioni diverse in un modello coeso che può essere simulato su strutture di classe dirigente come Frontera, "Ha detto Amaro. "In pratica partiamo dai singoli componenti, dove le loro strutture sono state risolte a risoluzione atomica o quasi atomica. Facciamo in modo che ciascuno di questi componenti sia installato e funzionante e in uno stato in cui sono stabili. Quindi possiamo introdurli nelle simulazioni di inviluppi più grandi con molecole vicine".
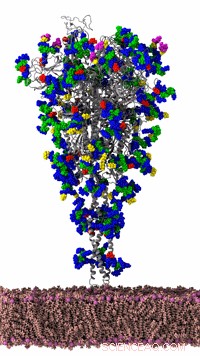
La proteina spike SARS-CoV-2 del coronavirus è stata simulata dal Romaro Lab dell'UC San Diego sul supercomputer Frontera del TACC finanziato dall'NSF a UT Austin. È la principale proteina virale coinvolta nell'infezione da coronavirus della cellula ospite. Le simulazioni di dinamica molecolare basate sulla fisica possono prevedere come si muove la macchina molecolare del coronavirus, che consente ai ricercatori di comprendere le sue vulnerabilità a potenziali vaccini e farmaci. Testa di Spike di Coronavirus basata su pdb 6vsb; gambo a punta utilizzando modelli di omologia; glicoprofilo progettato secondo Walls et al. 2019 Cella, e Watanabe et al. 2020 bioRxiv. Credito:Rommie Amaro, UCSD
Il 12-13 marzo, 2020, l'Amaro Lab ha eseguito simulazioni di dinamica molecolare fino a 4, 000 nodi, o circa 250, 000 core di elaborazione, su Frontera. Frontera, il supercomputer numero 5 al mondo e il supercomputer accademico numero 1 secondo la classifica di novembre 2019 dell'organizzazione Top500, è il sistema di elaborazione ad alte prestazioni di classe leader supportato dalla National Science Foundation.
"Simulazioni di quelle dimensioni possono essere eseguite solo su una macchina come Frontera o su una macchina possibilmente presso il Dipartimento dell'Energia, Ha detto Amaro. "Abbiamo subito contattato la squadra di Frontera, e sono stati molto gentili nel darci lo stato di priorità per il benchmarking e nel cercare di ottimizzare il codice in modo che queste simulazioni possano essere eseguite nel modo più efficiente possibile, una volta che il sistema è effettivamente installato e funzionante."
"È emozionante lavorare su una di queste macchine nuove di zecca, di sicuro. La nostra esperienza finora è stata molto buona. I benchmark iniziali sono stati davvero impressionanti per questo sistema. Continueremo a ottimizzare i codici per questi sistemi ultra grandi in modo da poter ottenere prestazioni ancora migliori. Direi che anche lavorare con il team di Frontera è stato fantastico. Sono pronti ad aiutare e sono stati estremamente reattivi durante questa finestra temporale critica. È stata un'esperienza molto positiva, " ha detto Amaro.
"TACC è orgogliosa di supportare questa ricerca critica e innovativa, " disse Dan Stanzione, Direttore Esecutivo di TACC e Principal Investigator del progetto di supercomputer Frontera. "Continueremo a supportare le simulazioni di Amaro e altri importanti lavori relativi alla comprensione e alla ricerca di un modo per sconfiggere questa nuova minaccia".

Il supercomputer Frontera del Texas Advanced Computing Center di UT Austin, finanziato dalla NSF, è al quinto posto più veloce al mondo e al primo posto per i sistemi accademici, secondo la classifica Top500 di novembre 2019. Credito:TACC
Il lavoro di Amaro con il coronavirus si basa sul suo successo con una simulazione di tutti gli atomi dell'involucro del virus dell'influenza, pubblicato in Scienze Centrali ACS , Febbraio 2020. Ha detto che il lavoro sull'influenza avrà un numero notevole di somiglianze con ciò che stanno ora perseguendo con il coronavirus.
"È un brillante test dei nostri metodi e delle nostre capacità di adattarci ai nuovi dati e di metterli in funzione immediatamente, Amaro ha detto. "Ci è voluto un anno o più per costruire l'involucro virale dell'influenza e farlo funzionare sui supercomputer nazionali. Per l'influenza, abbiamo usato il supercomputer Blue Waters, che era in qualche modo il predecessore di Frontera. Il lavoro, però, con il coronavirus ovviamente sta procedendo molto, ritmo molto più veloce. Questo è abilitato, in parte a causa del lavoro che abbiamo fatto in precedenza su Blue Waters."
Amaro ha detto:"Queste simulazioni ci daranno nuove informazioni sulle diverse parti del coronavirus necessarie per l'infettività. E perché ci interessa questo è perché se possiamo comprendere queste diverse caratteristiche, gli scienziati hanno maggiori possibilità di progettare nuovi farmaci; per capire come funzionano i farmaci attuali e le potenziali combinazioni di farmaci. Le informazioni che otteniamo da queste simulazioni sono sfaccettate e multidimensionali e saranno utili agli scienziati in prima linea immediatamente e anche a lungo termine. Si spera che il pubblico capisca che ci sono molti diversi componenti e aspetti della scienza da spingere in avanti per comprendere questo virus. Queste simulazioni su Frontera sono solo una di quelle componenti, ma si spera importante e redditizio".