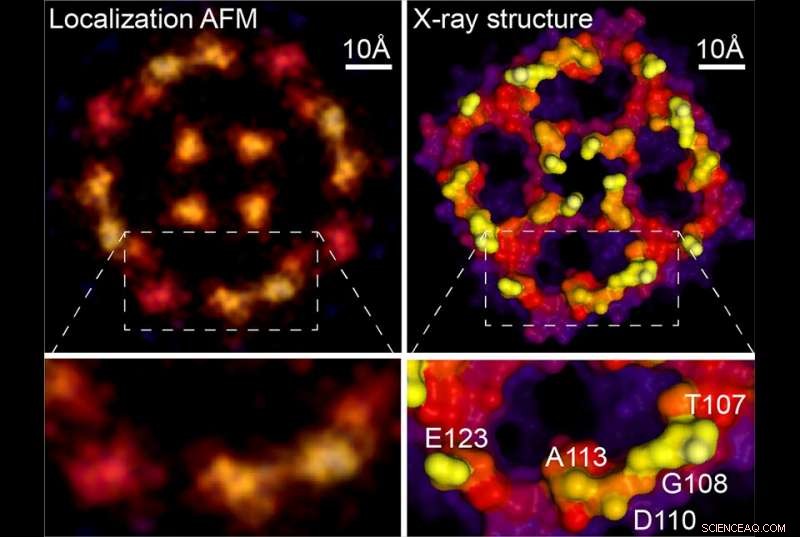
Localizzazione AFM e struttura a raggi X. Credito:Weill Cornell Medicine, Università di Leeds e Università di Washington
Gli scienziati della Weill Cornell Medicine hanno sviluppato una tecnica computazionale che aumenta notevolmente la risoluzione della microscopia a forza atomica, un tipo specializzato di microscopio che "sente" gli atomi su una superficie. Il metodo rivela dettagli a livello atomico su proteine e altre strutture biologiche in condizioni fisiologiche normali, aprendo una nuova finestra sulla biologia cellulare, virologia e altri processi microscopici.
In uno studio, pubblicato il 16 giugno in Natura , gli investigatori descrivono la nuova tecnica, che si basa su una strategia utilizzata per migliorare la risoluzione in microscopia ottica.
Per studiare proteine e altre biomolecole ad alta risoluzione, i ricercatori si sono a lungo affidati a due tecniche:la cristallografia a raggi X e la microscopia crioelettronica. Mentre entrambi i metodi possono determinare le strutture molecolari fino alla risoluzione dei singoli atomi, lo fanno su molecole che sono o impalcate in cristalli o congelate a temperature ultra-fredde, eventualmente alterandoli dalle loro normali forme fisiologiche. La microscopia a forza atomica (AFM) può analizzare le molecole biologiche in condizioni fisiologiche normali, ma le immagini risultanti sono state sfocate e a bassa risoluzione.
"La microscopia a forza atomica può facilmente risolvere gli atomi in fisica, su superfici solide di silicati e su semiconduttori, quindi significa che in linea di principio la macchina ha la precisione per farlo, " ha detto l'autore senior Dr. Simon Scheuring, professore di fisiologia e biofisica in anestesiologia presso Weill Cornell Medicine. "La tecnica è un po' come prendere una penna e scansionare le Montagne Rocciose, in modo da ottenere una mappa topografica dell'oggetto. In realtà, la nostra penna è un ago affilato fino a pochi atomi e gli oggetti sono singole molecole proteiche".
Però, le molecole biologiche hanno molte piccole parti che si muovono, sfocando le loro immagini AFM. Per affrontare quel problema, Il dottor Scheuring ei suoi colleghi hanno adattato un concetto della microscopia ottica chiamato microscopia a super risoluzione. "In teoria non era possibile con la microscopia ottica risolvere due molecole fluorescenti che fossero più vicine tra loro della metà della lunghezza d'onda della luce, " ha detto. Tuttavia, stimolando le molecole adiacenti a diventare fluorescenti in tempi diversi, i microscopisti possono analizzare la diffusione di ogni molecola e individuare le loro posizioni con alta precisione.
Invece di stimolare la fluorescenza, Il team del Dr. Scheuring ha notato che le fluttuazioni naturali delle molecole biologiche registrate nel corso delle scansioni AFM producono spread simili di dati posizionali. Primo autore Dr. George Heath, che era un associato post-dottorato presso Weill Cornell Medicine al momento dello studio ed è ora membro di facoltà presso l'Università di Leeds, impegnati in cicli di esperimenti e simulazioni computazionali per comprendere il processo di imaging AFM in modo più dettagliato ed estrarre il massimo di informazioni dalle interazioni atomiche tra punta e campione.
Utilizzando un metodo come l'analisi a super risoluzione, sono stati in grado di estrarre immagini a risoluzione molto più elevata delle molecole in movimento. Continuando l'analogia topografica, Il dottor Scheuring ha spiegato che "se le rocce (cioè, atomi) oscillano un po' su e giù, puoi rilevare questo, poi quello, e poi fai la media di tutti i rilevamenti nel tempo e ricevi informazioni ad alta risoluzione."
Poiché i precedenti studi AFM hanno regolarmente raccolto i dati necessari, la nuova tecnica può essere applicata retroattivamente alle immagini sfocate che il campo ha generato per decenni. Come esempio, il nuovo documento include un'analisi di una scansione AFM di una proteina di membrana di acquaporina, originariamente acquisito durante la tesi di dottorato del Dr. Scheuring. La rianalisi ha generato un'immagine molto più nitida che corrisponde strettamente alle strutture della cristallografia a raggi X della molecola. "In pratica ora ottieni una risoluzione quasi atomica su queste superfici, " ha detto il dottor Scheuring. Per mostrare la potenza del metodo, gli autori forniscono nuovi dati ad alta risoluzione sull'annessina, una proteina coinvolta nella riparazione della membrana cellulare, e su un antiporter protone-cloruro di cui riportano anche modifiche strutturali legate al suo funzionale.
Oltre a consentire ai ricercatori di studiare molecole biologiche in condizioni fisiologicamente rilevanti, il nuovo metodo ha altri vantaggi. Per esempio, La cristallografia a raggi X e la microscopia crioelettronica si basano su dati medi di un gran numero di molecole, ma l'AFM può generare immagini di singole molecole. "Invece di avere osservazioni di centinaia di molecole, osserviamo una molecola cento volte e calcoliamo una mappa ad alta risoluzione, " ha detto il dottor Scheuring.
L'imaging di singole molecole mentre svolgono le loro funzioni potrebbe aprire tipi di analisi completamente nuovi. "Supponiamo che tu abbia una proteina spike [virale] che si trova in una conformazione e poi si attiva e va in un'altra conformazione, " ha detto il dottor Scheuring. "In linea di principio saresti in grado di calcolare una mappa ad alta risoluzione da quella stessa molecola mentre transita da una conformazione all'altra, non da migliaia di molecole nell'una o nell'altra conformazione." Tali dati di una singola molecola ad alta risoluzione potrebbero fornire informazioni più dettagliate ed evitare i risultati potenzialmente fuorvianti che possono verificarsi quando si calcola la media dei dati di molte molecole. Inoltre, la mappa potrebbe rivelare nuove strategie per reindirizzare o interrompere con precisione tali processi.