I ricercatori del NIST mirano a trasformare la cellula in una fabbrica di computer biologici progettando e inserendo il DNA nel genoma di una cellula. Le proteine cellulari produrrebbero RNA basato sul DNA attraverso la trascrizione. Il filamento di RNA si sarebbe quindi piegato, legandosi a se stesso e diviso in due, grazie a una speciale sequenza di RNA auto-scissione chiamata ribozima. La struttura risultante, una porta del circuito dell'RNA, verrebbe annullata e innescherà ulteriori reazioni chimiche solo in determinate condizioni. Credito:N. Hanacek/NIST
Piccoli computer biologici fatti di DNA potrebbero rivoluzionare il modo in cui diagnostichiamo e curiamo una serie di malattie, una volta che la tecnologia sarà completamente sviluppata. Tuttavia, uno dei principali ostacoli per questi dispositivi basati sul DNA, che possono funzionare sia nelle cellule che nelle soluzioni liquide, è stato la loro breve durata. Un solo utilizzo e i computer sono esauriti.
Ora, i ricercatori del National Institute of Standards and Technology (NIST) potrebbero aver sviluppato computer biologici di lunga durata che potrebbero potenzialmente persistere all'interno delle cellule. In un articolo pubblicato sulla rivista Science Advances , gli autori rinunciano al tradizionale approccio basato sul DNA, optando invece per utilizzare l'RNA dell'acido nucleico per costruire computer. I risultati dimostrano che i circuiti dell'RNA sono affidabili e versatili quanto le loro controparti basate sul DNA. Inoltre, le cellule viventi potrebbero essere in grado di creare questi circuiti di RNA continuamente, cosa che non è prontamente possibile con i circuiti del DNA, posizionando ulteriormente l'RNA come un candidato promettente per computer biologici potenti e di lunga durata.
Proprio come il computer o il dispositivo intelligente su cui stai leggendo questo articolo, i computer biologici possono essere programmati per svolgere diversi tipi di attività.
"La differenza è che, invece di codificare con uno e zero, si scrivono stringhe di A, T, C e G, che sono le quattro basi chimiche che compongono il DNA", ha affermato Samuel Schaffter, ricercatore post-dottorato del NIST e autore principale dello studio .
Assemblando una sequenza specifica di basi in un filamento di acido nucleico, i ricercatori possono dettare a cosa si lega. Un filamento potrebbe essere progettato per attaccarsi a specifici frammenti di DNA, RNA o alcune proteine associate a una malattia, quindi innescare reazioni chimiche con altri filamenti nello stesso circuito per elaborare le informazioni chimiche e alla fine produrre una sorta di output utile.
Tale output potrebbe essere un segnale rilevabile che potrebbe aiutare la diagnostica medica o potrebbe essere un farmaco terapeutico per curare una malattia.
Tuttavia, il DNA non è il materiale più robusto e può rompersi rapidamente in determinate condizioni. Le cellule possono essere ambienti ostili, poiché spesso contengono proteine che sminuzzano gli acidi nucleici. E anche se le sequenze di DNA rimangono abbastanza a lungo per rilevare il loro bersaglio, i legami chimici che formano le rendono inutili in seguito.
"Non possono fare cose come monitorare continuamente i modelli nell'espressione genica. Sono un uso, il che significa che ti danno solo un'istantanea", ha detto Schaffter.
Essendo anche un acido nucleico, l'RNA condivide molti dei problemi del DNA quando si tratta di essere un elemento costitutivo di un computer biologico. È suscettibile di una rapida degradazione e dopo che un filamento si lega chimicamente a una molecola bersaglio, quel filamento è finito. Ma a differenza del DNA, l'RNA potrebbe essere una risorsa rinnovabile nelle giuste condizioni. Per sfruttare questo vantaggio, Schaffter e i suoi colleghi dovevano prima dimostrare che i circuiti dell'RNA, che le cellule sarebbero teoricamente in grado di produrre, potrebbero funzionare altrettanto bene del tipo basato sul DNA.
Il vantaggio dell'RNA sul DNA deriva da un processo cellulare naturale chiamato trascrizione, in cui le proteine producono RNA su base continua utilizzando il DNA di una cellula come modello. Se il DNA nel genoma di una cellula fosse codificato per i componenti del circuito in un computer biologico, la cellula produrrebbe continuamente i componenti del computer.
Nel processo di calcolo biologico, singoli filamenti di acidi nucleici in un circuito possono facilmente finire legati ad altri filamenti nello stesso circuito, un effetto indesiderato che impedisce ai componenti del circuito di legarsi ai bersagli previsti. Il design di questi circuiti spesso significa che i diversi componenti si adatteranno in modo naturale l'uno all'altro.
Per prevenire il legame indesiderato, le sequenze di DNA che fanno parte di computer noti come circuiti di spostamento del filamento vengono solitamente sintetizzate (in macchine anziché in cellule) separatamente e in una forma a doppio filamento. Con ogni base chimica su ogni filamento legato a una base sull'altro, questo doppio filamento funge da cancello bloccato che si sbloccherebbe solo se la sequenza bersaglio arrivasse e prendesse il posto di uno dei filamenti.
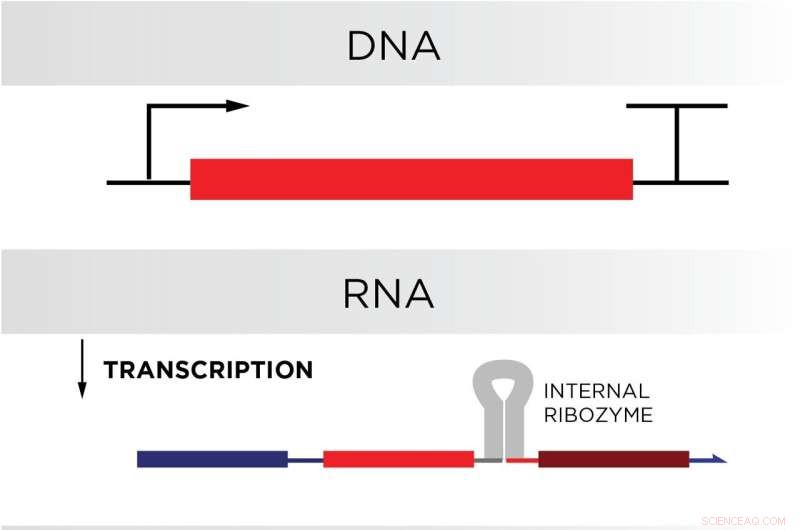
Schaffter ed Elizabeth Strychalski, leader del Cellular Engineering Group del NIST e coautrice dello studio, hanno cercato di imitare questa funzione di "cancello bloccato" nel loro circuito dell'RNA, tenendo presente che, in definitiva, le cellule avrebbero dovuto produrre queste porte bloccate da sole. Per preparare le cellule al successo, i ricercatori hanno scritto le sequenze in modo che una metà dei fili potesse legarsi a filo con l'altra metà. Legandosi in questo modo, le sequenze di RNA si ripiegherebbero su se stesse come un panino da hot dog, assicurandosi che siano in uno stato bloccato.
Ma per funzionare correttamente, i cancelli dovrebbero essere due fili chimicamente legati ma distinti, più simili a un panino o un panino per hamburger che a un panino con hotdog. Il team ha ottenuto il design a doppio filamento nei propri cancelli codificando un tratto di RNA chiamato ribozima vicino al punto di piegatura dei cancelli. Questo particolare ribozima, tratto dal genoma di un virus dell'epatite, si reciderebbe da solo dopo che il filamento di RNA in cui era stato incorporato piegato, creando due filamenti separati.
Gli autori hanno testato se i loro circuiti potessero eseguire operazioni logiche di base, come sbloccare le loro porte solo in scenari specifici, come se fosse presente una delle due sequenze di RNA specifiche o solo se entrambe fossero contemporaneamente. Hanno anche costruito ed esaminato circuiti costituiti da più porte che eseguivano diverse operazioni logiche in serie. Solo quando questi circuiti incontravano la giusta combinazione di sequenze, i loro cancelli si sbloccavano uno per uno come le tessere del domino.
Gli esperimenti hanno comportato l'esposizione di diversi circuiti a pezzi di RNA, alcuni dei quali i circuiti sono stati progettati per attaccarsi, e la misurazione dell'uscita dei circuiti. In questo caso, l'uscita alla fine di ogni circuito era una molecola reporter fluorescente che si accendeva una volta sbloccato il gate finale.
I ricercatori hanno anche monitorato la velocità con cui le porte si sono sbloccate mentre i circuiti elaboravano gli input e hanno confrontato le loro misurazioni con le previsioni dei modelli computerizzati.
"Per me, questi dovevano funzionare in una provetta in modo predittivo come il calcolo del DNA. La cosa bella con i circuiti del DNA è la maggior parte delle volte, puoi semplicemente scrivere una sequenza su un pezzo di carta e funzionerà in questo modo vuoi", ha detto Schaffter. "La cosa fondamentale qui è che abbiamo scoperto che i circuiti dell'RNA erano molto prevedibili e programmabili, molto più di quanto pensassi, in realtà."
Le somiglianze nelle prestazioni tra i circuiti del DNA e dell'RNA potrebbero indicare che potrebbe essere vantaggioso passare a quest'ultimo, poiché l'RNA può essere trascritto per ricostituire i componenti di un circuito. E molti circuiti del DNA esistenti che i ricercatori hanno già sviluppato per svolgere vari compiti potrebbero teoricamente essere sostituiti con versioni di RNA e comportarsi allo stesso modo. Per essere sicuri, però, gli autori dello studio devono spingere ulteriormente la tecnologia.
In questo studio, gli autori hanno dimostrato che i circuiti trascrivibili funzionano, ma non li hanno ancora prodotti utilizzando il vero meccanismo cellulare della trascrizione. Invece, le macchine sintetizzavano gli acidi nucleici attraverso un processo simile a quello utilizzato per produrre il DNA per la ricerca. Il passaggio successivo richiederebbe l'inserimento del DNA nel genoma di un organismo, dove servirebbe da modello per i componenti del circuito dell'RNA.
"Siamo interessati a inserirli successivamente nei batteri. Vogliamo sapere:possiamo impacchettare i progetti di circuiti in materiale genetico usando la nostra strategia? Possiamo ottenere lo stesso tipo di prestazioni e comportamento quando i circuiti sono all'interno delle cellule?" ha detto Schaffter. "Abbiamo il potenziale per". + Esplora ulteriormente