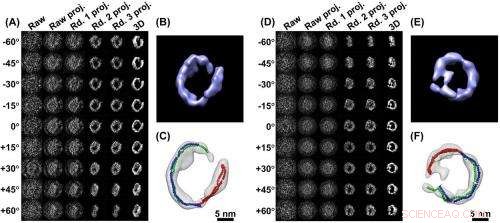
Immagini 3D da una singola particella (A) una serie di immagini di una particella proteica ApoA-1, presa da diverse angolazioni come indicato. Una successione di quattro miglioramenti del computer (proiezioni) chiarisce il segnale. Nella colonna di destra è l'immagine 3-D compilata dai dati chiariti. B) è un primo piano dell'immagine 3D ricostruita. C) L'analisi mostra come la struttura delle particelle sia formata da tre proteine ApoA-1 (rosso, verde, modelli simili a pasta blu)
(PhysOrg.com) -- Quando Gang Ren fa roteare i controlli del suo microscopio crioelettronico, lo paragona alla messa a punto del cambio e dei freni di una bicicletta da corsa. Ma questa macchina del Lawrence Berkeley National Laboratory (Berkeley Lab) del Dipartimento dell'Energia degli Stati Uniti (DOE) è un po' più complessa. Costa quasi 1,5 milioni di dollari, opera alla temperatura gelida dell'azoto liquido, e consente agli scienziati di vedere ciò che nessuno ha mai visto prima.
Alla Fonderia Molecolare, L'acclamato centro di ricerca sulle nanotecnologie del Berkeley Lab, Ren ha spinto il suo microscopio Zeiss Libra 120 Cryo-Tem a risoluzioni mai immaginate dai suoi produttori tedeschi, produrre istantanee dettagliate delle singole molecole. Oggi, lui e il suo collega Lei Zhang stanno riportando le prime immagini 3D di una singola proteina mai ottenute con sufficiente chiarezza per determinarne la struttura.
Gli scienziati creano regolarmente modelli di proteine usando la diffrazione dei raggi X, risonanza magnetica nucleare, e imaging al microscopio crioelettronico convenzionale (cryoEM). Ma questi modelli richiedono una "media" computerizzata dei dati dall'analisi di migliaia, o anche milioni di molecole simili, perché è così difficile risolvere le caratteristiche di una singola particella. Ren e Zhang hanno fatto proprio questo, generazione di modelli dettagliati utilizzando immagini al microscopio elettronico di una singola proteina.
Egli chiama la sua tecnica "tomografia elettronica a particelle individuali, ” o IPET. Il lavoro è descritto nel numero del 24 gennaio di PLoS One , la rivista scientifica open source, in un articolo intitolato "IPET e FETR:approccio sperimentale per lo studio della dinamica della struttura molecolare mediante tomografia crioelettronica di una struttura a singola molecola".
Le immagini 3D riportate nel documento includono quelle di un singolo anticorpo IgG e apolipoproteina A-1 (ApoA-1), una proteina coinvolta nel metabolismo umano. L'obiettivo di Ren è produrre immagini 3D individuali di proteine significative dal punto di vista medico, come l'HDL, il "colesterolo buono" protettivo per il cuore la cui struttura è sfuggita agli sforzi di legioni di scienziati armati di strumenti di modellazione proteica molto più potenti. “Siamo sulla buona strada, "dice Ren.
Ren ha le credenziali di uno che sa cosa può fare. È stato reclutato per lavorare al Berkeley Lab nell'agosto 2010 dall'Università della California a San Francisco, dove aveva usato un microscopio crioelettronico e tecniche di calcolo della media più convenzionali per discernere la struttura 3-D di LDL - il "colesterolo cattivo" pensato per essere un importante fattore di rischio per le malattie cardiache.
Le sue immagini di singole proteine sono un po' confuse, anche dopo essere stati ripuliti da complessi filtri del computer, ma molto informativo per l'osservatore addestrato. Queste singole particelle sono straordinariamente minuscole, richiedendo a Ren di azzerare un punto di meno di 20 nanometri. Ha riportato immagini proteiche di appena 70 kDa. sono kilodalton, una scala lillipuziana (espressa in unità di massa) destinata a misurare gli atomi, molecole, e frammenti di DNA. È un modo più utile per dimensionare oggetti morbidi come proteine che possono essere raggruppate, filante, o floscio.
A differenza delle immagini scultoree dei modelli proteici, una suite di queste fotografie può trasmettere un senso di queste particelle in tutta la loro floscicità su nanoscala. All'interno della complessa struttura di queste proteine si trovano i segreti della loro funzione, e forse chiavi per farmaci che bloccano quelli cattivi e promuovono quelli buoni. Con alcuni filtri aggiuntivi del computer, un modello di proteina ad alto contrasto può essere generato dalle immagini e animato per mostrare le sue parti mobili in 3-D.
“Questo ti permette di vedere la personalità di ogni proteina, '' dice Ren. "È una prova di concetto per qualcosa che la gente pensava fosse impossibile."
Osservando la struttura delle singole proteine, è possibile capire la loro flessibilità, parti mobili. “Questo apre una porta per lo studio della dinamica delle proteine, "dice Ren. “Anticorpi, Per esempio, non sono solidi. Sono molto flessibili, molto dinamico».
In che modo Ren ha ottenuto così tanta versatilità dal suo Libra 120? “Non è un modello di fascia alta, '' ammette. Molto ha a che fare con gli accessori che monta sulla macchina, e con la sua maestria e pazienza. Ha dotato il microscopio di 300 dollari, 000 telecamera CCD, un potente software di elaborazione delle immagini, agenti speciali di contrasto, e un dispositivo chiamato "filtro energetico" che setaccia i dati digitalizzati della fotocamera e seleziona i segnali deboli. Conoscenza approfondita della sua macchina personalizzata, impiega anche un elemento di olio di gomito, lavorando lunghe ore per tirare fuori le potenti immagini da un torrente di rumore digitale.

Lo scienziato dello staff Gang Ren (in piedi) e il collega postdoc Lei Zhang possono controllare le immagini delle singole proteine dal loro microscopio crioelettronico presso la fonderia molecolare del Berkeley Lab.
Gli angoli multipli utilizzati per creare il ritratto 3D aiutano a risolvere l'immagine molecolare debole. “Tutte le immagini sono rumorose, ” spiega Ren. “In fisica, il rumore è incoerente tra le immagini, ma il segnale - l'oggetto o la proteina - è coerente. Utilizzando questo approccio, troviamo che la porzione consistente (il segnale) può essere potenziata, mentre la parte inconsistente (il rumore) sarà ridotta sostanzialmente.”
I microscopi elettronici focalizzano flussi di elettroni piuttosto che la luce per vedere cose incredibilmente piccole. La corta lunghezza d'onda di un fascio di elettroni consente una risoluzione e un ingrandimento molto più elevati rispetto alla luce visibile. Potenti microscopi elettronici sono stati utilizzati per decenni per sondare materiali su scala atomica; e proprio accanto alla Molecular Foundry si trova il National Center for Electron Microscopy del Berkeley Lab, che ospita i microscopi più potenti del mondo. Il microscopio TEAM 0.5 è in grado di distinguere oggetti piccoli quanto il raggio di un atomo di idrogeno. Ma questi microscopi pesanti tirano fuori questa risoluzione su scala atomica con impulsi di energia che cancellerebbero la maggior parte delle proteine biologiche morbide. I microscopi elettronici ad alta potenza sono utilizzati principalmente per sondare la struttura atomica di forti, materiali solidi, come il grafene, un reticolo di carbonio dello spessore di un solo atomo.
Il laboratorio di Ren è specializzato in cryoEM, che esamina oggetti congelati a -180 °C (-292 °F). Un bagno di azoto liquido congela i campioni così rapidamente che non si formano cristalli di ghiaccio. “È amorfo, come il vetro, '' dice Ren. I campioni di proteine vengono congelati su un disco delle dimensioni dell'unghia di un bambino, riempito con piccoli pozzetti di 2 micron di diametro. Il disco viene inserito nel microscopio su un supporto rotante che può inclinare il campione fino a 140° all'interno del vuoto, angoli di ripresa sufficienti per produrre una prospettiva 3D. “La sfida è isolarla dall'aria, e per farlo girare senza vibrazioni, anche le vibrazioni del gorgogliamento dell'azoto liquido, '' dice Ren.
La temperatura estremamente bassa fissa i campioni e impedisce che si secchino nel vuoto necessario per la scansione elettronica. Crea condizioni favorevoli per l'imaging a dosi di elettroni molto più basse, abbastanza basse da mantenere intatta una singola proteina morbida mentre vengono scattate più di 100 immagini in un periodo di una o due ore.