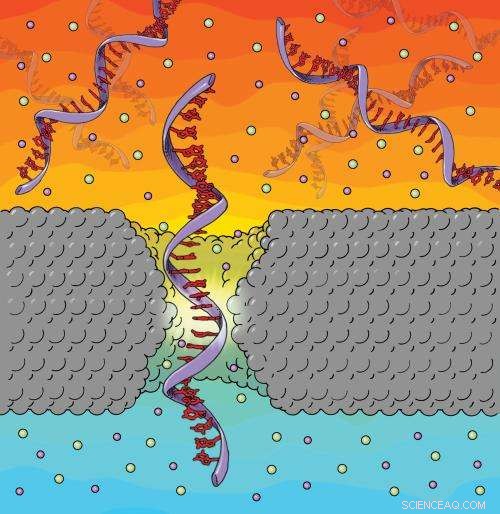
Questa è un'illustrazione di un omopolimero di DNA a filamento singolo che trasloca attraverso un nanoporo di nitruro di silicio. Lo spessore della membrana si assottiglia all'interno del nanoporo, esaltandone la sensibilità. Credito:Università della Pennsylvania
(Phys.org) —Il fascino della medicina personalizzata ha reso nuovi, modi più efficienti di sequenziare i geni una priorità assoluta della ricerca. Una tecnica promettente prevede la lettura delle basi del DNA utilizzando i cambiamenti nella corrente elettrica mentre vengono infilate attraverso un foro nanoscopico.
Ora, un team guidato da fisici dell'Università della Pennsylvania ha utilizzato nanopori allo stato solido per differenziare molecole di DNA a singolo filamento contenenti sequenze di una singola base ripetitiva.
Lo studio è stato condotto da Marija Drndić, professore associato presso il Dipartimento di Fisica e Astronomia della Scuola delle Arti e delle Scienze, insieme agli studenti laureati Kimberly Venta e Matthew Puster e ai ricercatori post-dottorato Gabriel Shemer, Julio A. Rodriguez-Manzo e Adrian Balan. Hanno collaborato con l'assistente professore Jacob K. Rosenstein della Brown University e il professor Kenneth L. Shepard della Columbia University.
I loro risultati sono stati pubblicati sulla rivista ACS Nano .
In questa tecnica, note come misurazioni della traslocazione del DNA, filamenti di DNA in una soluzione salina sono guidati attraverso un'apertura in una membrana da un campo elettrico applicato. Quando ciascuna base del filo passa attraverso il poro, blocca contemporaneamente il passaggio di alcuni ioni; gli amplificatori collegati al chip a nanopori possono registrare il conseguente calo di corrente elettrica. Poiché ogni base ha una dimensione diversa, i ricercatori sperano di utilizzare questi dati per dedurre l'ordine delle basi durante il passaggio del filo. Le differenze nelle dimensioni della base sono così piccole, però, che le proporzioni sia dei nanopori che delle membrane devono essere vicine a quelle dei filamenti di DNA stessi, una sfida importante.
I dispositivi nanopori più vicini ad essere un'opzione commercialmente valida per il sequenziamento sono costituiti da pori proteici e doppi strati lipidici. Tali pori proteici hanno proporzioni desiderabili, ma le membrane a doppio strato lipidico in cui sono inserite sono simili a una pellicola di sapone, che lascia molto a desiderare in termini di durata e robustezza.
Dispositivi nanopori allo stato solido, che sono costituiti da sottili membrane allo stato solido, offrono vantaggi rispetto alle loro controparti biologiche - possono essere più facilmente spediti e integrati con altri dispositivi elettronici - ma le dimostrazioni di base della sensibilità della prova del principio a diverse basi del DNA sono state più lente.
"Mentre i nanopori biologici hanno mostrato la capacità di risolvere singoli nucleotidi, le alternative allo stato solido sono rimaste indietro a causa di due sfide:produrre effettivamente i pori delle giuste dimensioni e ottenere un segnale elevato, misurazioni a basso rumore e ad alta larghezza di banda, " Drndić ha detto. "Stiamo attaccando queste due sfide qui."
Poiché il meccanismo mediante il quale il nanoporo differenzia tra un tipo di base e un altro è dovuto alla quantità di apertura del poro bloccata, più piccolo è il diametro di un poro, più è preciso. Affinché il nanoporo sia efficace nel determinare una sequenza di basi, il suo diametro deve avvicinarsi al diametro del DNA e il suo spessore deve avvicinarsi a quello dello spazio tra una base e l'altra, o circa 0,3 nanometri.
Per ottenere nanopori e membrane allo stato solido in queste minuscole proporzioni, ricercatori, compreso il gruppo di Drndić, stanno studiando materiali all'avanguardia, come il grafene. Un singolo strato di atomi di carbonio in un reticolo esagonale, le membrane di grafene possono essere realizzate con uno spessore di circa 0,5 nanometri, ma hanno i loro svantaggi da affrontare. Per esempio, il materiale stesso è idrofobo, rendendo più difficile il passaggio di filamenti di DNA attraverso di essi.
In questo esperimento, Drndić e i suoi colleghi hanno lavorato con un materiale diverso, il nitruro di silicio, piuttosto che tentare di creare membrane di grafene spesse un atomo per i nanopori. Il nitruro di silicio trattato è idrofilo e ha prontamente consentito traslocazioni di DNA, come misurato da molti altri ricercatori nell'ultimo decennio. E mentre la loro membrana è più spessa, circa 5 nanometri, i pori di nitruro di silicio possono anche avvicinarsi al grafene in termini di sottigliezza a causa del modo in cui sono fabbricati.
"Il modo in cui realizziamo i nanopori nel nitruro di silicio li fa assottigliare, in modo che lo spessore effettivo sia circa un terzo del resto della membrana, "Drndic ha detto.
Drndić e i suoi colleghi hanno testato il loro nanoporo di nitruro di silicio su omopolimeri, oppure singoli filamenti di DNA con sequenze costituite da una sola base ripetuta più volte. I ricercatori sono stati in grado di effettuare misurazioni distinte per tre delle quattro basi:adenina, citosina e timina. Non hanno tentato di misurare la guanina poiché gli omopolimeri realizzati con quella base si legano su se stessi, rendendo più difficile il loro passaggio attraverso i nanopori.
"Mostriamo che questi piccoli pori sono sensibili al contenuto di base, "Drndic ha detto, "e abbiamo visto questi risultati in pori con diametri compresi tra 1 e 2 nanometri, il che è in realtà incoraggiante perché suggerisce che una certa variabilità di produzione potrebbe andare bene".