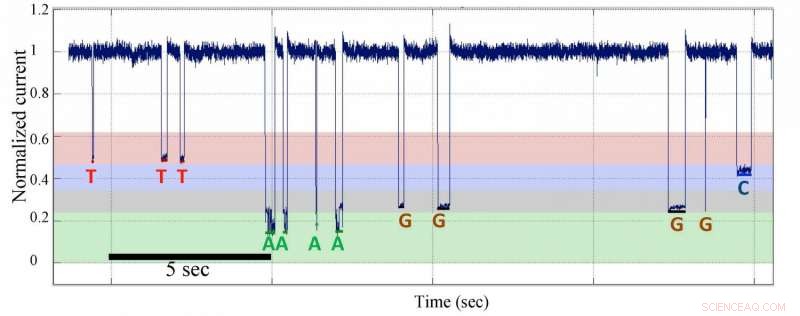
Sequenziamento di DNA a nanopori a singola molecola mediante sintesi di dati da uno stampo con sequenze di omopolimeri. Credito:Jingyue Ju, Columbia Engineering
Ricercatori della Columbia University, con i colleghi di Genia Technologies (Roche), L'Università di Harvard e il National Institute of Standards and Technology (NIST) riferiscono di aver ottenuto il sequenziamento elettronico del DNA di una singola molecola in tempo reale con una risoluzione a base singola utilizzando una matrice di nanopori proteici.
Il sequenziamento del DNA è la tecnologia chiave per iniziative di medicina personalizzata e di precisione, consentendo rapide scoperte nella scienza biomedica. La sequenza completa del genoma di un individuo fornisce importanti marcatori e linee guida per la diagnostica medica, assistenza sanitaria, e mantenere una vita sana. Ad oggi, il costo e la velocità necessari per ottenere sequenze di DNA altamente accurate hanno rappresentato una sfida importante. Sebbene negli ultimi dieci anni siano stati fatti diversi progressi, gli strumenti di sequenziamento ad alto rendimento ampiamente utilizzati oggi dipendono dall'ottica per il rilevamento di quattro elementi costitutivi del DNA:A, C, G e T. Per esplorare capacità di misurazione alternative, è stato sviluppato il sequenziamento elettronico di un insieme di modelli di DNA per l'analisi genetica. Sequenziamento del filamento nanopore, in cui un singolo filamento di DNA viene fatto passare attraverso i pori su scala nanometrica sotto una tensione elettrica applicata per produrre segnali elettronici per la determinazione della sequenza a livello di singola molecola, è stato recentemente sviluppato; però, perché i quattro nucleotidi sono molto simili nelle loro strutture chimiche, non possono essere facilmente distinti usando questo metodo. I ricercatori stanno quindi perseguendo attivamente la ricerca e lo sviluppo di un'accurata piattaforma di sequenziamento elettronico del DNA a singola molecola in quanto ha il potenziale per produrre un sequenziatore di DNA miniaturizzato in grado di decifrare il genoma per facilitare la medicina di precisione personalizzata.
Un team di ricercatori della Columbia Engineering, diretto da Jingyue Ju (Samuel Ruben-Peter G. Viele Professore di Ingegneria, Professore di Ingegneria Chimica e Farmacologia, Direttore del Centro per la tecnologia del genoma e l'ingegneria biomolecolare), con i colleghi della Harvard Medical School, guidato da George Church (professore di genetica); Tecnologie Genia, guidato da Stefan Roever (CEO di Genia); e John Kasianowicz, il Principal Investigator al NIST, hanno sviluppato un sistema completo per sequenziare il DNA in nanopori elettronicamente a livello di singola molecola con risoluzione a base singola. Questo lavoro, intitolato, "Sequenziamento elettronico del DNA di singole molecole in tempo reale mediante sintesi utilizzando nucleotidi etichettati con polimeri su un array di nanopori, " è pubblicato sulla rivista, Atti dell'Accademia Nazionale delle Scienze ( PNAS ) Prima edizione.
In precedenza, ricercatori dei laboratori di Ju alla Columbia e Kasianowicz al NIST hanno riportato il principio generale del sequenziamento dei nanopori per sintesi (SBS), la fattibilità della progettazione e sintesi di nucleotidi marcati con polimeri come substrati per la DNA polimerasi, il rilevamento e la differenziazione dei tag polimerici per nanopori a livello di singola molecola [ Rapporti scientifici 2, 684 (2012) DOI:10.1038/srep00684]. Il corrente PNAS l'articolo descrive la costruzione del sistema SBS a nanopori completo per produrre dati di sequenziamento elettronico di singole molecole con risoluzione a base singola. Questa strategia SBS distingue accuratamente quattro basi di DNA rilevando e differenziando elettronicamente quattro diversi tag polimerici attaccati al 5'-fosfato dei nucleotidi durante la loro incorporazione in un filamento di DNA in crescita catalizzato dalla polimerasi, un enzima di sintesi del DNA. I ricercatori hanno progettato e sintetizzato nuovi nucleotidi etichettati al fosfato terminale con polimeri a base di oligonucleotidi per eseguire SBS a nanopori su una piattaforma di array di nanopori di proteina α-emolisina. I tag sui nucleotidi marcati con polimeri, che sono stati verificati come substrati attivi per la DNA polimerasi, producono diversi livelli di blocco della corrente elettrica. Hanno costruito un array di nanopori su un chip elettronico con più elettrodi; l'array è composto da canali proteici che sono stati accoppiati a una DNA polimerasi che era legata a uno stampo di DNA innescato. L'aggiunta di nucleotidi etichettati con polimeri distinti progettati su misura all'array di nanopori innesca la sintesi del DNA. Bloccando la corrente ionica del canale a diversi livelli, i tag distinti forniscono una lettura della sequenza del modello in tempo reale con risoluzione a base singola.
Come Carl Fuller, autore principale, Adjunct Senior Research Scientist presso il Ju Laboratory del Dipartimento di Ingegneria Chimica della Columbia e Direttore di Chimica presso Genia, sottolinea, "La novità del nostro approccio SBS a nanopori inizia con il design, sintesi, e selezione di quattro diversi nucleotidi marcati con polimeri. Usiamo una DNA polimerasi attaccata covalentemente al nanoporo e ai nucleotidi etichettati per eseguire SBS. Durante la replicazione del DNA legato alla polimerasi, il tag di ciascun nucleotide complementare viene catturato nel poro per produrre un segnale elettrico unico. Per la determinazione della sequenza vengono utilizzati quattro distinti tag polimerici che producono firme distinte che vengono riconosciute dal rivelatore elettronico nel chip array di nanopori. Così, Le sequenze di DNA vengono ottenute per molte singole molecole in parallelo e in tempo reale. I quattro tag polimerici sono progettati per offrire distinzioni molto migliori tra loro, in contrasto con le piccole differenze tra i quattro nucleotidi nativi del DNA, superando così la grande sfida affrontata da altri metodi di sequenziamento diretto dei nanopori." Inoltre, i tag possono essere ulteriormente ottimizzati rispetto alle dimensioni, carica, e struttura per fornire una risoluzione ottimale nel sistema SBS a nanopori.
"Questo entusiasmante progetto riunisce scienziati e ingegneri sia del mondo accademico che dell'industria con competenze combinate nell'ingegneria molecolare, nanotecnologia, genomica, l'elettronica e la scienza dei dati per produrre prodotti rivoluzionari, piattaforme di diagnostica genetica economicamente vantaggiose con un potenziale senza precedenti per la medicina di precisione, " dice Ju. "Siamo estremamente grati per il generoso supporto del NIH che ci ha permesso di fare rapidi progressi nella ricerca e nello sviluppo della tecnologia SBS a nanopori, e gli eccezionali contributi di tutti i membri del nostro consorzio di ricerca."
Secondo Ju, i ricercatori si sono già spinti oltre quanto dimostrato nel PNAS studio in cui i dati di sequenziamento sono stati ottenuti su un primo prototipo di sequenziatore basato su nanopori SBS. Il throughput e le prestazioni dell'attuale sequencer sono andati oltre quanto riportato nel PNAS carta. Recentemente è stata raggiunta la fattibilità di raggiungere lunghezze di lettura di oltre 1000 basi di DNA. Andando avanti, il team di ricerca collaborativa continuerà a ottimizzare i tag modificando i linker, struttura, e carica a livello molecolare, e messa a punto della polimerasi e dell'elettronica per il sistema SBS a nanopori con l'obiettivo di sequenziare accuratamente un intero genoma umano rapidamente e a basso costo, consentendo così di essere utilizzato nelle diagnosi mediche di routine.