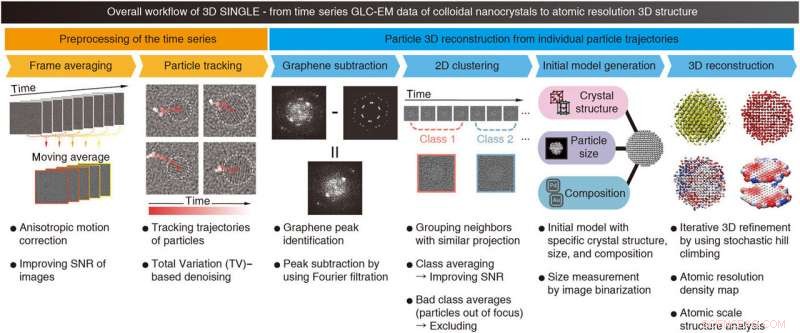
Flusso di lavoro complessivo di SINGLE. Vengono mostrati il flusso di lavoro generale e le descrizioni di ogni passaggio di SINGLE. SINGLE si compone di due fasi principali:elaborazione preliminare delle serie temporali (arancione), tra cui (i) media del frame della finestra temporale con correzione del movimento anisotropico e (ii) tracciamento della traiettoria delle particelle con l'utilizzo del denoising basato sulla variazione totale (TV), e ricostruzione 3D di particelle da singole traiettorie di particelle (blu), tra cui (i) l'identificazione e la sottrazione dello sfondo di grafene, (ii) clustering 2D a tempo limitato con esclusione di immagini fuori fuoco, (iii) generazione iniziale del modello, e (iv) ricostruzione 3D e analisi della struttura su scala atomica. Credito:progressi scientifici, doi:10.1126/sciadv.abe6679
Gli scienziati dei materiali utilizzano in genere la microscopia elettronica a trasmissione in fase di soluzione (TEM) per rivelare le proprietà fisiochimiche uniche delle strutture tridimensionali (3D) dei nanocristalli. In un nuovo rapporto su Progressi scientifici , Cyril F. Reboul e un gruppo di ricerca della Monash University, Australia, Università Nazionale di Seul, Corea del Sud, e il Lawrence Berkeley National Laboratory negli Stati Uniti, sviluppato un metodo di ricostruzione 3-D browniano a particella singola. Per realizzare questo, hanno ripreso insiemi di nanocristalli colloidali utilizzando la microscopia elettronica a trasmissione di cellule liquide di grafene. Il team ha ottenuto immagini di proiezione di nanocristalli ruotati in modo diverso utilizzando un rivelatore di elettroni diretto per ottenere un insieme di ricostruzioni 3D. In questo lavoro, hanno introdotto metodi computazionali per ricostruire con successo i nanocristalli 3-D a risoluzione atomica e hanno raggiunto questo obiettivo tracciando le singole particelle nel tempo, sottraendo lo sfondo interferente. Il metodo potrebbe anche identificare/rifiutare immagini di bassa qualità per facilitare strategie su misura per l'allineamento 2-D/3-D che differivano da quelle della microscopia crioelettronica biologica. Il team ha reso disponibili gli sviluppi tramite un pacchetto software open source noto come SINGLE. Il software gratuito è disponibile su GitHub.
Utilizzo di SINGLE per la cristallografia
I ricercatori hanno sostenuto progressi nella cristallografia negli ultimi 50 anni per trasformare la comprensione esistente della chimica e della biologia. Tuttavia, alcuni bersagli, inclusi i nanocristalli solubilizzati, rimangono intrattabili con i metodi cristallografici tradizionali. Ad esempio, i nanocristalli colloidali contengono da decine a centinaia di atomi e mantengono una varietà di applicazioni in campi multidisciplinari tra cui l'elettronica, catalisi e sensori biologici. La versatilità deriva dall'elevata sensibilità delle proprietà dei nanocristalli alle dimensioni, composizione chimica e altre variabili durante la sintesi. Tipicamente, gli scienziati usano la singola particella, Ricostruzione 3D in biologia strutturale per determinare la struttura delle proteine. La tecnica è relativamente nuova per la ricostruzione 3-D in situ di singoli nanocristalli solubilizzati. In questo lavoro, Reboul et al. sviluppato SINGOLO; un metodo che si basava sulla ricostruzione 3D indipendente di singoli nanocristalli solubilizzati, incluso il movimento browniano. La tecnica è un primo progresso nello studio per risolvere strutture atomiche 3-D di nanocristalli direttamente dalla fase di soluzione.
Tracciamento delle traiettorie dei singoli nanocristalli. Monitoraggio dei risultati della particella 1 (A e B) e 2 (C e D) per tutto il film (dal blu al rosso). Sono mostrate le medie temporali rappresentative delle immagini di particelle grezze non allineate (50 fotogrammi) (B e D). Barre della scala, 1 nanometro. Credito:progressi scientifici, doi:10.1126/sciadv.abe6679 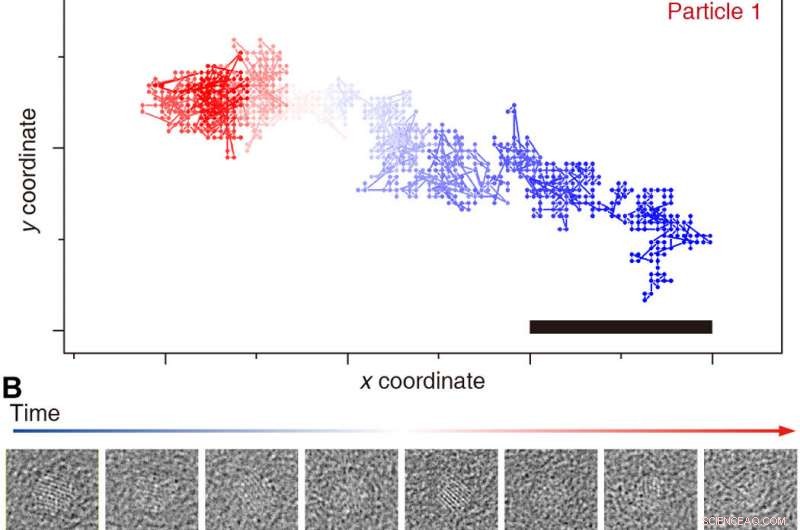
Gli scienziati hanno introdotto nuovi metodi di pre-elaborazione per migliorare il rapporto segnale-rumore (SNR) per tracciare le traiettorie delle particelle rimuovendo il segnale di fondo indotto dal grafene. Metodi computazionali avanzati potrebbero ricostruire con successo in 3-D dai dati di microscopia elettronica a trasmissione di cellule liquide di grafene (GLC) in situ. Rispetto alle tecniche esistenti, il lavoro ha presentato l'applicabilità di un metodo computazionale senza precedenti per ottenere ricostruzioni 3-D a risoluzione atomica per nanocristalli dispersi in soluzione. Hanno diviso il flusso di lavoro SINGLE in due fasi principali (1) pre-elaborazione e (2) ricostruzione 3-D delle particelle. Gli scienziati miravano a fornire le massime prestazioni ed efficienza possibili su qualsiasi hardware della CPU, compresi i supercomputer alle workstation o persino ai laptop.
All'inizio, il team ha fatto la media della finestra temporale su diversi fotogrammi con movimento anisotropico per migliorare il rapporto segnale-rumore, portando a particelle visibili e un segnale di grafene potenziato. Il team ha quindi identificato manualmente le posizioni delle particelle nella prima media della finestra temporale. Successivamente, il team ha sviluppato un modello di partenza basato sulla struttura cristallografica prevista, diametro delle particelle e degli elementi costitutivi e ha prodotto ricostruzioni 3D con coordinate atomiche adattate per l'analisi della struttura su scala atomica.
Clustering 2D a tempo limitato. (A) Frazione della variazione angolare lungo la serie temporale. La linea tratteggiata rossa è la linea di tendenza. Le direzioni di proiezione cambiano rapidamente nelle regioni comprese tra i frame 1500 e 1600 (arancione), cornici da 3800 a 3900 (verde), e cornici da 5600 a 5700 (blu). I riquadri sono grafici che rappresentano la differenza angolare nella direzione di proiezione in quelle regioni. (B) Rappresentazione schematica della media di classe 2D limitata nel tempo. (C) Grafico che mostra le classi allocate per i singoli frame nella regione da 1 a 400. L'inserto è un grafico che mostra le classi allocate su tutti i frame. (D) Medie di classe ottenute con il raggruppamento e l'allineamento 2D a tempo limitato. Credito:progressi scientifici, doi:10.1126/sciadv.abe6679 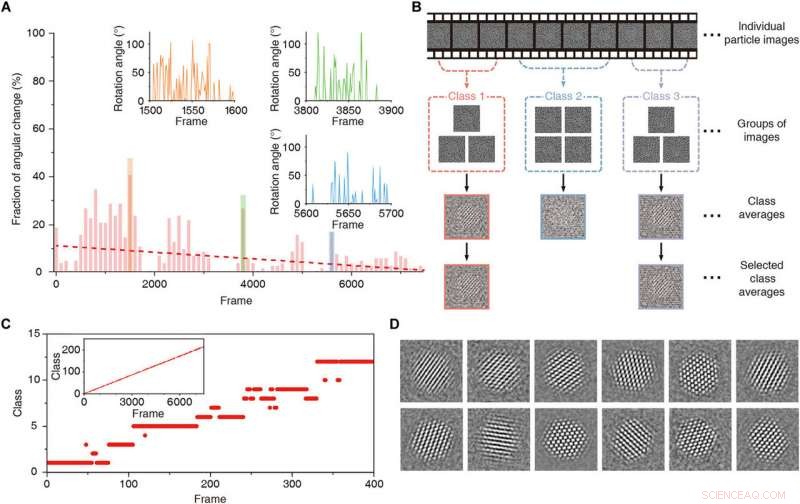
Reboul et al. ha introdotto un nuovo metodo di tracciamento che utilizza le trasformate di Fourier veloci e la correlazione di fase per identificare un massimo di correlazione con precisione sub-pixel. Il team ha denoizzato la finestra temporale di estrazione utilizzando il denoising basato sulla variazione totale (TV) e combinando il denoising e la media temporale per fornire un metodo robusto per tracciare il movimento dei singoli nanocristalli in tutto il campione. Il metodo ha permesso loro di discernere la forma complessiva dei nanocristalli e/o le loro caratteristiche cristalline, il che ha attestato la robustezza dell'algoritmo di tracciamento. Utilizzando il metodo, hanno anche recuperato traiettorie precedentemente impegnative per ottenere ricostruzioni 3D e hanno impiegato una traiettoria di particelle sottratte dallo sfondo in tutte le fasi di elaborazione delle immagini per la sottrazione di grafene del GLC (cella liquida di grafene). Il team ha ulteriormente caratterizzato la natura delle rotazioni dei nanocristalli nello spazio altamente ristretto della cella liquida di grafene. Il metodo non era banale a causa della natura probabilistica dell'algoritmo di ricostruzione 3-D. Il team ha quindi incorporato un approccio deterministico per migliorare l'accuratezza del cluster, migliorando il rapporto segnale-rumore rispetto ai singoli frame.
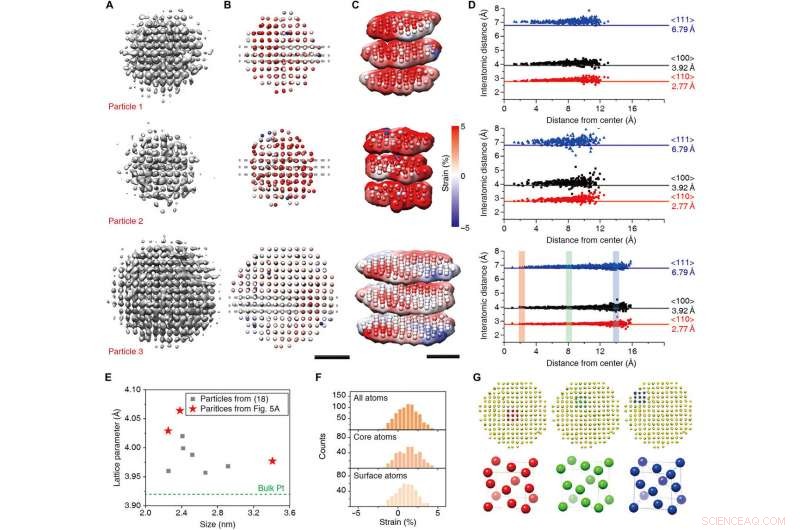
Risultati della ricostruzione 3D e analisi della struttura a livello atomico. (da A a C) mappe di densità 3D (A), mappe di deformazione radiale da coordinate atomiche adattate (B), e la loro rappresentazione della fetta (C). (D) Distanze interatomiche nelle direzioni di <110> (rosso), <100> (Nero), e <111> (blu) per tre nanocristalli. (E) Parametri reticolari adattati di nanocristalli precedentemente riportati (18) (quadrati grigi) e nuovi risultati (stelle rosse). (F) Istogramma della deformazione radiale di tutti gli atomi (in alto), atomi del nucleo (al centro), atomi di superficie (fondo) della particella 3. (G) Struttura cellulare unitaria del nucleo (rosso), mezzo (verde), e superficie (blu) della particella 3. Barre della scala, 1 nanometro. Credito:progressi scientifici, doi:10.1126/sciadv.abe6679
Generazione di modelli
I ricercatori hanno quindi sviluppato un modello di partenza utilizzando la conoscenza che le particelle hanno una disposizione di posizione atomica approssimativamente cubica. Hanno simulato le densità atomiche utilizzando fattori di diffusione atomica 5-gaussiana. Le proiezioni 2-D della densità 3-D simulata rappresentavano il carattere delle proiezioni nel nucleo del nanocristallo, per superare i problemi legati alla simmetria traslazionale e un segnale di fondo interferente. Il metodo di raffinamento 3-D in uso per la microscopia crioelettronica biologica non può essere applicato direttamente ai dati delle serie temporali dei nanocristalli; perciò, Reboul et al. introdotto modifiche critiche. Hanno usato uno schema di raffinamento a due fasi per stabilire la forma corretta del nanocristallo per consentire agli atomi e alle loro forme di guidare l'allineamento 3D. I ricercatori hanno scelto tre nanocristalli di varie dimensioni che non erano stati precedentemente ricostruiti per il benchmarking, quindi utilizzando mappe atomiche prodotte con il metodo, Reboul et al. ottenuto dettagli strutturali microscopici a livello atomico. Il lavoro ha anche facilitato le mappe atomiche che dettagliano l'analisi della deformazione e l'analisi della struttura cellulare unitaria.
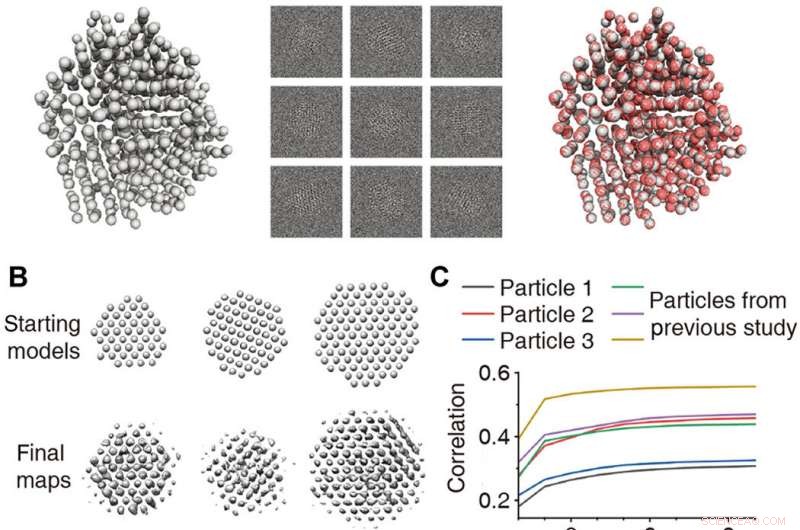
Validazione di ricostruzioni 3D. (A) Ricostruzione 3D di particelle disordinate simulate con struttura atomica nota. A sinistra:modello di un nanocristallo disordinato ottenuto mediante simulazione di dinamica molecolare. Medio:5000 immagini simulate multistrato con rumore aggiunto per ottenere un SNR =0,1. A destra:mappa atomica (rosso) come risultato della ricostruzione 3D sovrapposta al modello della verità al suolo (grigio). (B) Modelli 3D di partenza e mappe di densità 3D finali ottenute da dati sperimentali. (C) Correlazione tra le riproiezioni della mappa di densità 3D raffinata e le viste sperimentali delle particelle tracciate in funzione dell'iterazione per la prima fase di raffinamento 3D. Le particelle presentate in questo articolo (nero, rosso, e colore blu) e presentato in uno studio precedente (18) (verde, viola, e colore ocra) sono tracciati. (D) Confronto delle medie di classe (indicate come proiezione) con le riproiezioni per la validazione delle tre strutture. (E) Rappresentazione atomica dipendente dal tempo delle direzioni di proiezione per le tre strutture:dal bianco (inizio) al rosa (centro) al rosso (fine). Rosso, giallo, e le frecce blu indicano x, si, e z assi, rispettivamente. Credito:progressi scientifici, doi:10.1126/sciadv.abe6679
Convalida delle ricostruzioni 3D
I ricercatori hanno inoltre generato un modello di un nanocristallo disordinato utilizzando simulazioni di dinamica molecolare per comprendere l'applicabilità di SINGLE a nanocristalli altamente disordinati. Utilizzando simulazioni multi-slice, hanno applicato il movimento traslazionale e variazioni di sfocatura casuali per rappresentare il movimento realistico delle particelle. Hanno quindi ottenuto una mappa di densità 3-D del nanocristallo disordinato da 500 immagini simulate con un rapporto segnale-rumore di 0,1 e un modello di partenza con un ordine cristallino perfetto per concordare in modo eccellente con le particelle originali. Il team ha ottenuto la distribuzione delle direzioni di proiezione dei nanocristalli rotanti per convalidare la qualità della ricostruzione 3-D e richiederà ulteriori studi per capire come le strutture atomiche effettive dei nanocristalli influenzano la dinamica di rotazione.
In questo modo, Cyril F. Reboul e colleghi hanno dimostrato metodi computazionali in SINGLE per ottenere mappe di densità di nanocristalli a risoluzione atomica. Utilizzando una configurazione avanzata di celle liquide come celle liquide al grafene con nanocamere ordinate, il team ha consentito il controllo dello spessore del liquido per estendere l'applicabilità di SINGLE per un'acquisizione dati efficiente. La suite SINGLE ha fornito una prima piattaforma analitica efficiente per comprendere l'origine strutturale delle proprietà fisiche e chimiche uniche dei nanocristalli nella loro fase di soluzione nativa.
© 2021 Science X Network