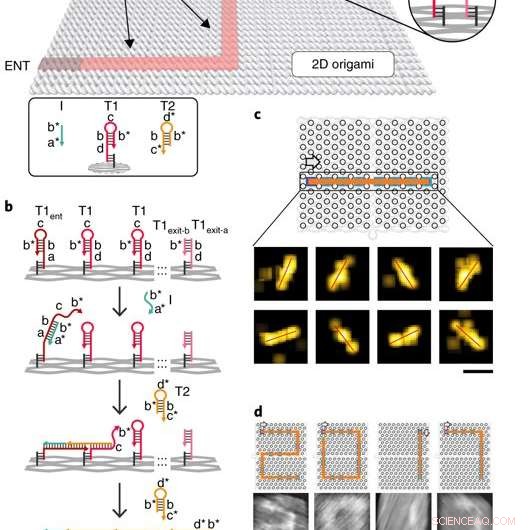
Implementazione del navigatore del DNA a singola molecola. a) Illustrazione schematica del sistema PSEC. Un grafo connesso aciclico (cioè un albero) è costruito su un substrato di origami di DNA rettangolare di 100 x 70 nm^2. T1 e T2 sono due tipi di combustibili che guidano il PSEC sull'albero. L'iniziatore I è stato utilizzato per attivare l'avvio del PSEC dal vertice di ingresso ENT. b) meccanismo di propagazione del sistema PSEC. L'iniziatore I riconosce e apre il tornante T1ent al vertice ENT. Il tornante aperto quindi cattura e apre un tornante T2 dall'ambiente per avviare la cascata. c) Una linea retta pavimentata da PSEC e visualizzata con DNA-PAINT. d) Quattro cifre pavimentate di "2017" acquisite mediante microscopia a forza atomica (AFM). I tornanti T1 nei punti d'angolo sono stati modificati per evitare campate indesiderate. Le frecce indicano la direzione di propagazione. Credito: Materiali della natura , doi:10.1038/s41563-018-0205-3.
Il campo della nanorobotica intelligente si basa sulla grande promessa di dispositivi molecolari con capacità di elaborazione delle informazioni. In un nuovo studio che supporta la tendenza dei portatori di informazioni basati sul DNA, gli scienziati hanno progettato un sistema di navigazione del DNA in grado di eseguire singole molecole, parallelo, operazioni di ricerca in profondità su una piattaforma di origami bidimensionale.
Le operazioni di individuazione del percorso con i navigatori del DNA utilizzano un processo a cascata di scambio di filamenti localizzato avviato in un sito di innesco unico sulla piattaforma origami. La progressione automatica lungo i percorsi è resa possibile da forcine di DNA contenenti una sequenza trasversale universale. In base alla progettazione, ogni navigatore di singola molecola può esplorare autonomamente uno qualsiasi dei possibili percorsi attraverso un albero radicato a 10 vertici costruito nello studio. I labirinti erano equivalenti a un albero con un'entrata alla radice e un'uscita attraverso una delle foglie. Lo studio condotto da Jie Chao e collaboratori ha portato all'esplorazione di tutti i percorsi intrapresi dai navigatori del DNA per estrarre un percorso di soluzione specifico che collegasse una data coppia di vertici di inizio e di fine nel labirinto. Di conseguenza, il percorso della soluzione è stato posato chiaramente sulla piattaforma dell'origami e illustrato utilizzando l'imaging a singola molecola. L'approccio è ora pubblicato in Materiali della natura , dettagliare la realizzazione di materiali molecolari con funzioni computazionali biomolecolari incorporate per operare a livello della singola molecola con il potenziale per progettare nanorobot intelligenti per future applicazioni nell'industria e nella medicina.
Sofisticati strumenti molecolari sono stati utilizzati in passato per creare macchine molecolari che convertono sostanze chimiche, energia fotonica o elettrica in movimenti rotatori o lineari su scala nanometrica. Ad esempio, Il movimento browniano su scala nanometrica può essere convertito in modo controllabile in movimenti diretti all'interno di nanomacchine basate sul DNA utilizzando reazioni di ibridazione del DNA. Tali macchine basate sul DNA operano in modo autonomo seguendo un "programma molecolare" incorporato pre-progettato come una reazione a cascata innescata manualmente tramite uno stimolo esterno per ogni fase dell'operazione.
Il focus del campo si è progressivamente spostato per realizzare circuiti logici basati sul DNA utilizzando aptameri e DNAzymes per progettare porte logiche molecolari. Per esempio, nel 2006, Stojanovic e i suoi collaboratori hanno integrato più di 100 porte logiche del DNA per progettare un'automazione chiamata MAYA-II per giocare a Tic-Tac-Toe. Studi precedenti hanno dimostrato un sistema di calcolo privo di enzimi basato su reazioni a catena di ibridazione (HCR) per creare porte logiche e circuiti logici per prestazioni più robuste ed efficienti rispetto ai sistemi originali.
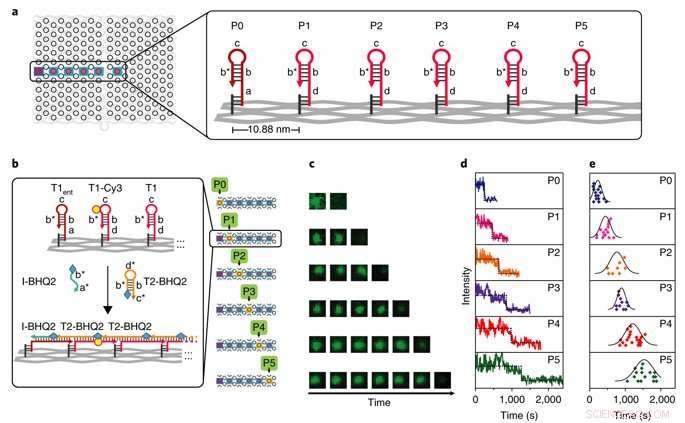
Caratterizzazione di singole molecole della cinetica della PSEC. a) Come banco di prova è stata utilizzata una linea retta nel mezzo dell'origami del DNA. Il vertice P0 è il punto di partenza e i vertici P1-P5 sono i punti intermedi. b) Dettagli del progetto di sei prove parallele per misurare la cinetica con TIRF risolta nel tempo. T2 etichettato con BHQ2 è stato utilizzato per spegnere T1 etichettato con Cy3, assemblando un profilo cinetico ad ogni passo. L'esempio illustrato mostra il meccanismo di tempra. c) Le immagini TIRF di esempio mostrano la fluorescenza che cambia nel tempo nei sei test paralleli da P0 a P5. d) Tipiche tracce di fluorescenza a singola molecola utilizzate per monitorare gli eventi di spegnimento che si verificano ai vertici da P0 a P5. e) Grafici a dispersione che mostrano l'analisi statistica della distribuzione della lunghezza per percorso. Credito: Materiali della natura , doi:10.1038/s41563-018-0205-3.
Nel presente studio di Chao et al, lo stesso principio di base dello schema di reazione HCR è stato utilizzato in un diverso contesto computazionale per sviluppare un sistema di navigazione del DNA a singola molecola. La piattaforma ha esplorato tutti i possibili percorsi attraverso un grafico ad albero disegnato su una struttura origami come un labirinto semplicemente connesso senza percorsi ciclici. Tali strutture di origami di DNA sono nanostrutture portatrici di informazioni per natura con una geometria su nanoscala ben definita. Il labirinto potrebbe essere esplorato mediante la cascata di scambio del filamento prossimale (PSEC) basata su reazioni a catena di ibridazione. I ricercatori hanno dimostrato che un sistema con un gran numero di navigatori di DNA a molecola singola potrebbe condurre collettivamente una ricerca parallela in profondità (PDFS) sull'albero per eseguire in modo efficiente la risoluzione di labirinti all'interno di origami 2-D. Inizialmente i ricercatori hanno condotto studi per testare il design del PSEC.
Il sistema PSEC (proximal strand exchange cacade) (principio di funzionamento del navigatore del DNA) è stato facilitato su un substrato di origami rettangolare composto da tre componenti, che includeva l'implementazione fisica di un grafo ad albero, filamenti completi e un filamento iniziatore. Aree vuote senza estensioni di base corrispondevano a muri nel labirinto, preventing propagation of the strand exchange cascade. The entrance and exit were defined and denoted as ENT and EXIT respectively. In the second component, two types of DNA hairpins, T1 and T2, were used as fuels to drive the PSEC on the tree graph. The two hairpins coexisted metastably in solution to hybridize and fuel the PSEC process with free energy .
In base alla progettazione, information only propagated through the network in the presence of an initiator (Initiator I). Upon addition of initiator I, PSEC was conducted and observed using atomic force microscopy (AFM). To visualize an established formation, the scientists enabled DNA-navigator-based formation of the number 2017 as a proof-of-principle. Another technique known as DNA-PAINT was employed as a single-molecule, super-resolution imaging technique to reveal molecular features at the nanoscale to further substantiate the PSEC-based path paving process. The on-origami PSEC was highly specific, without intra- or inter-origami crosstalk.
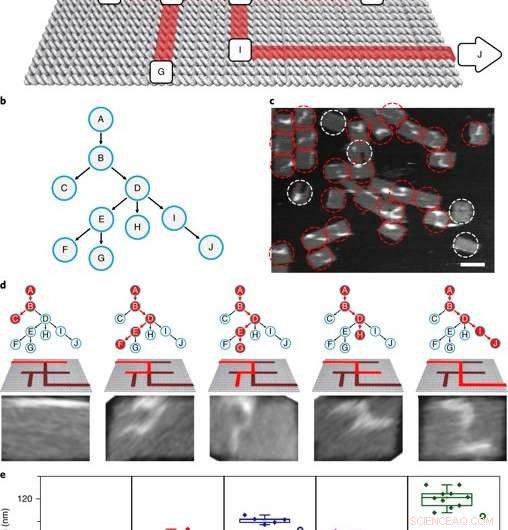
The PSEC-driven graph traversal on a maze. a) A maze design with 10 vertices. Arrows indicate the entrance vertex A and exit vertex J. b) The maze is equivalent to a rooted tree with 10 vertices. The entrance vertex A corresponds to the root of the tree. c) An AFM image showed the result of a transversal experiment generating all possible paths. In this DNA computing implementation of a PDFS algorithm, a vast number of PSEC events simultaneously occurred to realize the graph traversal on the maze. PSEC ending at the exit or deadends were highlighted in red circles. Invalid structures were highlighted with white circles. d) Typical paths found in the mixture seen from left to right. Only P ABDIJ was the correct solution to the maze. e) Scatter plots showing the statistical analysis of the length distribution for each path. Credito: Materiali della natura , doi:10.1038/s41563-018-0205-3.
Kinetics of the process were investigated at the single-molecule level in the study using time-resolved total internal reflection fluorescence microscopy (TIRF) in a setup with a prescribed starting point (P0) and five intermediate steps (P1-P5). Fluorescence in the setup was quenched using fluorescence resonance energy transfer (FRET), and the cascade was observed in real time by recording fluorescence signals continuously. The average speed of propagation was recorded to be 2.46 nm per minute, propagation across the straight line (54.4 nm) took approximately 22 minutes.
The scientists then constructed the main model maze with 10 vertices that included an entrance vertex A and an exit vertex J, three junctions (B, D, E) an intermediate vertex (I) and four dead ends (C, F, G, H). Each path of the maze that was equivalent to a tree with 10 vertices was investigated using DNA investigators starting at root A. The PSEC reactions produced a mixture of various paths on the maze, confirmed with AFM. Each individual PSEC could progress on one of the five possible paths. Statistical analysis of the length distribution showed that the measured paths coincided well with the values predicted.
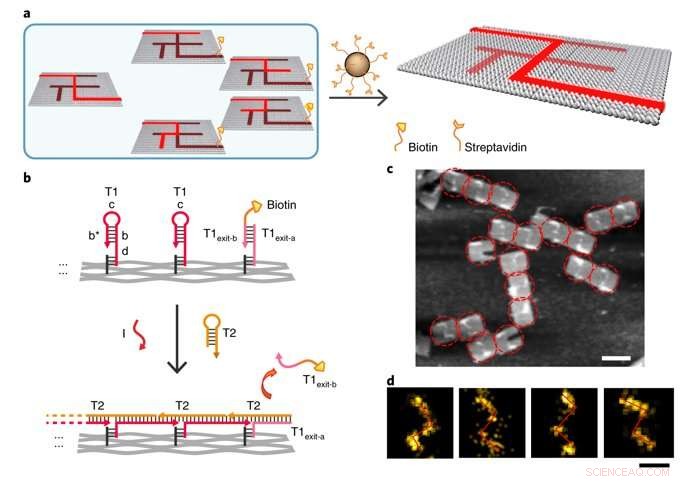
Single-molecule DNA navigators for maze-solving. a) Schematic illustration of magnetic bead-based selection. Exit vertex J is labelled with biotin to enable differentiation between the correct and wrong paths. Only if the PSEC reaches the correct exit J, would the biotin-modified T1exit-B strand be released. All wrong paths could be captured and removed by the Streptavidin-modified magmatic beads therefore. b) Details of the release of biotin modification at the exit J by the PSEC. c) AFM characterization of the correct solution after selection. The remaining structures all showed the correct solution path PABDIJ. d) Single-molecule and class-averaged DNA-PAINT characterization of the correct solution after selection. Credito: Materiali della natura , doi:10.1038/s41563-018-0205-3.
To prevent the navigators from propagating through a wrong path with dead ends, the scientists designed a streptavidin-biotin tag-based method to selectively eliminate inaccurate path navigation. Only the correct path (P ABDIJ ) was followed in the maze therefore. The computational context used in the study allowed exploration of paths through tree graphs defined on the origami. The autonomous path explored by the DNA navigators proceeded unidirectionally and irreversibly, turning at junctions and corners on the origami platform as they were designed to. The design enabled parallel depth first search (PDFS) allowing each DNA navigator to individually explore any one of the paths through the given graph at a defined speed, greater than that previously achieved.
The main advantage of the described biomolecular computer schemes in comparison to conventional electronic computing is they can be interfaced directly with biologically relevant processes. Di conseguenza, the scientists envision translational biomedical sensing and decision-making platforms with DNA origami and single-molecule diagnostics using decision trees. Future applications will also include simple sensors or those coupled to a molecular actuator to trigger downstream molecular cascades.
© 2018 Science X Network