Progettazione del motore e configurazione sperimentale. a, b, Schemi rispettivamente di un piedistallo e di una piattaforma triangolare. I cilindri indicano doppie eliche del DNA. c, Illustrazione schematica delle fasi di assemblaggio del motore. d,e, Componenti del braccio del rotore. f, a sinistra, illustrazione schematica della configurazione sperimentale per l'osservazione della dinamica motoria in un microscopio TIRF invertito. Il piedistallo è fissato attraverso diversi collegamenti biotina-neutravidina a un vetrino coprioggetto. Stella arancione, coloranti Cy5. Stelle blu, posizioni di etichettatura per i filamenti di imager DNA-PAINT. A destra, due elettrodi di platino sono immersi nella camera del liquido dall'alto e collegati ad un generatore di funzioni che genera una corrente alternata ad onda quadra per creare una modulazione energetica ad asse fisso che agisce su tutti i motori. Credito:Natura (2022). DOI:10.1038/s41586-022-04910-y
Un gruppo di ricerca guidato dall'Università Tecnica di Monaco (TUM) è riuscito per la prima volta a produrre un motore elettrico molecolare utilizzando il metodo dell'origami del DNA. La minuscola macchina fatta di materiale genetico si autoassembla e converte l'energia elettrica in energia cinetica. I nuovi nanomotori possono essere accesi e spenti ei ricercatori possono controllare la velocità di rotazione e la direzione di rotazione.
Che si tratti delle nostre auto, trapani o macinacaffè automatici, i motori ci aiutano a svolgere il lavoro nella nostra vita quotidiana per svolgere un'ampia varietà di compiti. Su scala molto più piccola, i motori molecolari naturali svolgono compiti vitali nei nostri corpi. Ad esempio, una proteina motoria nota come ATP sintasi produce la molecola adenosina trifosfato (ATP), che il nostro corpo utilizza per immagazzinare e trasferire energia a breve termine.
Sebbene i motori molecolari naturali siano essenziali, è stato abbastanza difficile ricreare motori su questa scala con proprietà meccaniche più o meno simili a quelle dei motori molecolari naturali come l'ATP sintasi. Un team di ricerca ha ora costruito un motore rotativo molecolare funzionante su scala nanometrica utilizzando il metodo dell'origami del DNA e ha pubblicato i risultati su Natura . Il team era guidato da Hendrik Dietz, professore di nanotecnologia biomolecolare al TUM, Friedrich Simmel, professore di fisica dei sistemi biologici sintetici al TUM e Ramin Golestanian, direttore del Max Planck Institute for Dynamics and Self-Organization.
Un nanomotore autoassemblante
Il nuovo motore molecolare è costituito da DNA, materiale genetico. I ricercatori hanno utilizzato il metodo dell'origami del DNA per assemblare il motore dalle molecole di DNA. Questo metodo è stato inventato da Paul Rothemund nel 2006 e successivamente è stato ulteriormente sviluppato dal team di ricerca del TUM. Diversi lunghi singoli filamenti di DNA servono come base a cui si attaccano ulteriori filamenti di DNA come controparti. Le sequenze di DNA sono selezionate in modo tale che i filamenti e le pieghe attaccati creino le strutture desiderate.
"Abbiamo avanzato questo metodo di fabbricazione per molti anni e ora possiamo sviluppare oggetti molto precisi e complessi, come interruttori molecolari o corpi cavi che possono intrappolare i virus. Se metti in soluzione i filamenti di DNA con le giuste sequenze, gli oggetti autoassemblare", afferma Dietz.
Il nuovo nanomotore realizzato in materiale DNA è composto da tre componenti:base, piattaforma e braccio del rotore. La base è alta circa 40 nanometri ed è fissata a una lastra di vetro in soluzione tramite legami chimici su una lastra di vetro. Un braccio del rotore lungo fino a 500 nanometri è montato sulla base in modo che possa ruotare. Un altro componente è fondamentale affinché il motore funzioni come previsto:una piattaforma che si trova tra la base e il braccio del rotore. Questa piattaforma contiene ostacoli che influenzano il movimento del braccio del rotore. Per superare gli ostacoli e ruotare, il braccio del rotore deve piegarsi leggermente verso l'alto, come un cricchetto.

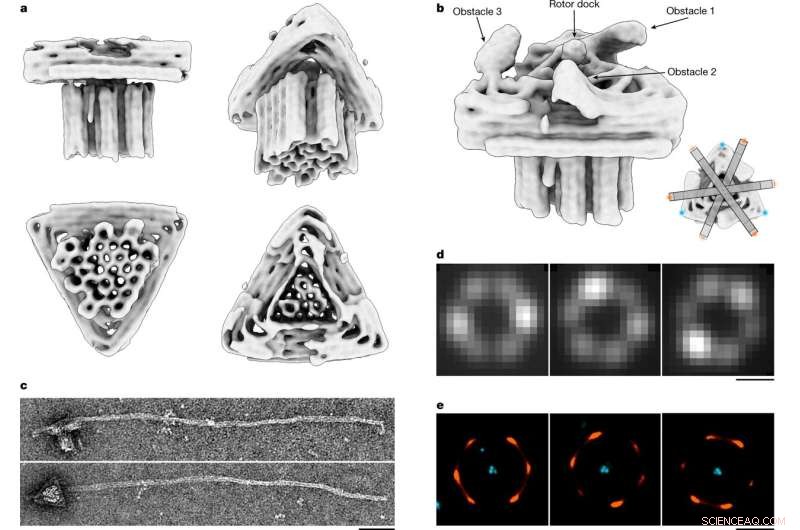
Analisi strutturale del motore dell'origami del DNA. a, Viste diverse di una mappa di densità elettronica 3D del blocco motore determinata mediante crio-EM a particella singola (vedere anche Dati estesi Fig. 4 e nella banca dati di microscopia elettronica (EMDB) con il codice EMD-14358). b, Dettagli della mappa crio-EM del blocco motore raffigurati a diverse soglie di densità alle quali è possibile distinguere i tre ostacoli e il dock del rotore. Riquadro, schematico che mostra i sei siti preferiti di abitazione del braccio del rotore. c, Immagini TEM esemplificative con colorazione negativa di una variante del motore con braccio del rotore lungo attaccato. Barra della scala, 50 nm. d, Esempi di immagini di fluorescenza a particella singola. Barra della scala, 500 nm. Le immagini mostrano la deviazione standard dell'intensità media per pixel calcolata su tutti i fotogrammi dei video TIRF registrati. e, immagini DNA-PAINT che mostrano le posizioni della punta del braccio del rotore rispetto alla piattaforma triangolare. Barra della scala, 500 nm. Credito:Natura (2022). DOI:10.1038/s41586-022-04910-y
Movimento mirato tramite tensione CA
Senza alimentazione di energia, i bracci del rotore dei motori si muovono casualmente in una direzione o nell'altra, azionati da collisioni casuali con le molecole del solvente circostante. Tuttavia, non appena la tensione CA viene applicata tramite due elettrodi, i bracci del rotore ruotano in modo mirato e continuo in una direzione.
"Il nuovo motore ha capacità meccaniche senza precedenti:può raggiungere coppie nell'intervallo di 10 piconewton per nanometri. E può generare più energia al secondo di quella che viene rilasciata quando due molecole di ATP vengono scisse", spiega Ramin Golestanian, che ha guidato l'analisi teorica del meccanismo del motore.
Il movimento mirato dei motori risulta dalla sovrapposizione delle forze elettriche fluttuanti con le forze subite dal braccio del rotore a causa degli ostacoli a cricchetto. Il meccanismo sottostante realizza un cosiddetto "cricchetto browniano lampeggiante". I ricercatori possono controllare la velocità e la direzione della rotazione tramite la direzione del campo elettrico e anche tramite la frequenza e l'ampiezza della tensione CA.
"Il nuovo motore potrebbe anche avere applicazioni tecniche in futuro. Se sviluppiamo ulteriormente il motore, potremmo eventualmente usarlo in futuro per guidare reazioni chimiche definite dall'utente, ispirandosi al modo in cui l'ATP sintasi rende l'ATP guidato dalla rotazione. Quindi, ad esempio , le superfici potrebbero essere densamente rivestite con tali motori. Quindi aggiungeresti materiali di partenza, applicheresti una piccola tensione CA e i motori produrranno il composto chimico desiderato", afferma Dietz. + Esplora ulteriormente