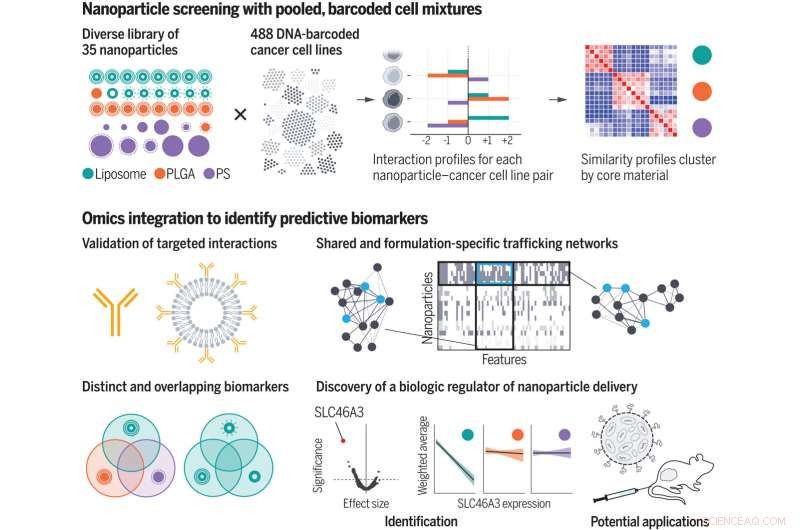
Utilizzando una libreria di nanoparticelle curata, i ricercatori hanno esaminato contemporaneamente i profili di interazione nanoparticella-cellula di centinaia di cellule tumorali. Incorporando l'annotazione omica, hanno identificato le caratteristiche biologiche, o biomarcatori, che mediano il rilascio di nanoparticelle alle cellule. Hanno generato reti di traffico e hanno scoperto un regolatore biologico della consegna di nanoparticelle a base di lipidi. PLGA, polilattide-co-glicolide; PS, polistirolo. Credito:Natalie Boehnke et al, Scienza (2022). DOI:10.1126/science.abm5551
L'uso di nanoparticelle per fornire farmaci contro il cancro offre un modo per colpire i tumori con grandi dosi di farmaci evitando gli effetti collaterali dannosi che spesso derivano dalla chemioterapia. Tuttavia, finora, solo una manciata di farmaci antitumorali a base di nanoparticelle è stata approvata dalla FDA.
Un nuovo studio del MIT e dei ricercatori del Broad Institute del MIT e di Harvard potrebbe aiutare a superare alcuni degli ostacoli allo sviluppo di farmaci a base di nanoparticelle. L'analisi del team delle interazioni tra 35 diversi tipi di nanoparticelle e quasi 500 tipi di cellule tumorali ha rivelato migliaia di tratti biologici che influenzano se quelle cellule assorbono diversi tipi di nanoparticelle.
I risultati potrebbero aiutare i ricercatori ad adattare meglio le loro particelle di somministrazione di farmaci a specifici tipi di cancro o progettare nuove particelle che sfruttano le caratteristiche biologiche di particolari tipi di cellule tumorali.
"Siamo entusiasti delle nostre scoperte perché è davvero solo l'inizio:possiamo utilizzare questo approccio per mappare quali tipi di nanoparticelle sono i migliori per colpire determinati tipi di cellule, dal cancro alle cellule immunitarie e altri tipi di cellule d'organo sane e malate. Stiamo imparando in che modo la chimica di superficie e altre proprietà dei materiali svolgono un ruolo nel targeting", afferma Paula Hammond, professoressa del MIT Institute, capo del Dipartimento di ingegneria chimica e membro del Koch Institute for Integrative Cancer Research del MIT.
Hammond è l'autore senior del nuovo studio, che appare in Scienza . Gli autori principali del documento sono Natalie Boehnke, un postdoc del MIT che presto si unirà alla facoltà dell'Università del Minnesota, e Joelle Straehla, Charles W. e Jennifer C. Johnson Clinical Investigator presso il Koch Institute, istruttrice presso la Harvard Medical School, e un oncologo pediatrico presso il Dana-Farber Cancer Institute.
Interazioni cellula-particella
Il laboratorio di Hammond ha precedentemente sviluppato molti tipi di nanoparticelle che possono essere utilizzate per somministrare farmaci alle cellule. Gli studi nel suo laboratorio e in altri hanno dimostrato che diversi tipi di cellule tumorali spesso rispondono in modo diverso alle stesse nanoparticelle. Anche Boehnke, che stava studiando il cancro alle ovaie quando si è unita al laboratorio di Hammond, e Straehla, che stava studiando il cancro al cervello, hanno notato questo fenomeno nei loro studi.
I ricercatori hanno ipotizzato che le differenze biologiche tra le cellule potrebbero guidare la variazione nelle loro risposte. Per capire quali potrebbero essere queste differenze, hanno deciso di condurre uno studio su larga scala in cui avrebbero potuto osservare un numero enorme di cellule diverse che interagiscono con molti tipi di nanoparticelle.
Straehla aveva recentemente appreso della piattaforma PRISM del Broad Institute, progettata per consentire ai ricercatori di eseguire rapidamente lo screening di migliaia di farmaci su centinaia di diversi tipi di cancro contemporaneamente. Con la collaborazione strumentale di Angela Koehler, una professoressa associata di ingegneria biologica del MIT, il team ha deciso di provare ad adattare quella piattaforma per schermare le interazioni cellula-nanoparticella invece delle interazioni cellula-farmaco.
"Utilizzando questo approccio, possiamo iniziare a pensare se c'è qualcosa nella firma genotipica di una cellula che predice quante nanoparticelle occuperà", afferma Boehnke.
Per il loro screening, i ricercatori hanno utilizzato 488 linee cellulari tumorali provenienti da 22 diversi tessuti di origine. Ogni tipo di cellula è "codificato a barre" con una sequenza di DNA univoca che consente ai ricercatori di identificare le cellule in un secondo momento. Per ogni tipo di cellula, sono inoltre disponibili set di dati estesi sui loro profili di espressione genica e altre caratteristiche biologiche.
Sul lato delle nanoparticelle, i ricercatori hanno creato 35 particelle, ognuna delle quali aveva un nucleo costituito da liposomi (particelle costituite da molte molecole grasse chiamate lipidi), un polimero noto come PLGA o un altro polimero chiamato polistirene. I ricercatori hanno anche rivestito le particelle con diversi tipi di molecole protettive o mirate, inclusi polimeri come polietilenglicole, anticorpi e polisaccaridi. Ciò ha permesso loro di studiare l'influenza sia della composizione del nucleo che della chimica superficiale delle particelle.
Lavorando con gli scienziati del Broad Institute, tra cui Jennifer Roth, direttrice del laboratorio PRISM, i ricercatori hanno esposto pool di centinaia di cellule diverse a una delle 35 diverse nanoparticelle. Ogni nanoparticella aveva un tag fluorescente, quindi i ricercatori potevano utilizzare una tecnica di smistamento cellulare per separare le cellule in base alla quantità di fluorescenza che emanavano dopo un'esposizione di quattro o 24 ore.
Sulla base di queste misurazioni, a ciascuna linea cellulare è stato assegnato un punteggio che ne rappresentava l'affinità per ciascuna nanoparticella. I ricercatori hanno quindi utilizzato algoritmi di apprendimento automatico per analizzare quei punteggi insieme a tutti gli altri dati biologici disponibili per ciascuna linea cellulare.
Questa analisi ha prodotto migliaia di caratteristiche, o biomarcatori, associati all'affinità per diversi tipi di nanoparticelle. Molti di questi marcatori erano geni che codificano per il macchinario cellulare necessario per legare le particelle, portarle in una cellula o elaborarle. Alcuni di questi geni erano già noti per essere coinvolti nel traffico di nanoparticelle, ma molti altri erano nuovi.
"Abbiamo trovato alcuni marcatori che ci aspettavamo e abbiamo anche trovato molto di più che è stato davvero inesplorato. Speriamo che altre persone possano utilizzare questo set di dati per espandere la loro visione di come interagiscono le nanoparticelle e le cellule", afferma Straehla.
Assorbimento di particelle
I ricercatori hanno scelto uno dei biomarcatori che hanno identificato, una proteina chiamata SLC46A3, per ulteriori studi. Lo schermo PRISM aveva mostrato che livelli elevati di questa proteina erano correlati con un assorbimento molto basso di nanoparticelle a base di lipidi. Quando i ricercatori hanno testato queste particelle in modelli murini di melanoma, hanno trovato la stessa correlazione. I risultati suggeriscono che questo biomarcatore potrebbe essere utilizzato per aiutare i medici a identificare i pazienti i cui tumori hanno maggiori probabilità di rispondere alle terapie a base di nanoparticelle.
Ora, i ricercatori stanno cercando di scoprire il meccanismo di come SLC46A3 regola l'assorbimento di nanoparticelle. Se potessero scoprire nuovi modi per diminuire i livelli cellulari di questa proteina, ciò potrebbe aiutare a rendere i tumori più suscettibili ai farmaci veicolati dalle nanoparticelle lipidiche. I ricercatori stanno anche lavorando per esplorare ulteriormente alcuni degli altri biomarcatori che hanno trovato.
Questo approccio di screening potrebbe essere utilizzato anche per studiare molti altri tipi di nanoparticelle che i ricercatori non hanno esaminato in questo studio.
"Il cielo è il limite in termini di ciò che altri biomarcatori sconosciuti sono là fuori che non abbiamo semplicemente catturato perché non li abbiamo schermati", afferma Boehnke. "Speriamo che sia un'ispirazione per gli altri iniziare a guardare i loro sistemi di nanoparticelle in un modo simile". + Esplora ulteriormente
Questa storia è stata ripubblicata per gentile concessione di MIT News (web.mit.edu/newsoffice/), un popolare sito che copre notizie sulla ricerca, l'innovazione e l'insegnamento del MIT.