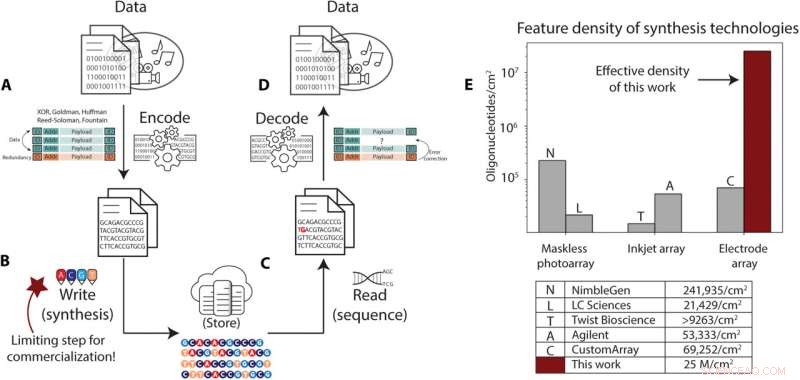
L'archiviazione dei dati del DNA richiede un throughput di sintesi più elevato rispetto a quanto è possibile con le tecniche attuali. (A-D) Panoramica della pipeline di archiviazione dei dati del DNA. (A) I dati digitali sono codificati dalla loro rappresentazione binaria in sequenze di basi di DNA, con un identificatore che li correla con un oggetto dati, indirizzando le informazioni che vengono utilizzate per riordinare i dati durante la lettura e le informazioni ridondanti utilizzate per la correzione degli errori. (B) Queste sequenze sono sintetizzate in oligonucleotidi di DNA e conservate. (C) Al momento del recupero, le molecole di DNA vengono selezionate e copiate tramite PCR o altri metodi e sequenziate nuovamente in rappresentazioni elettroniche delle basi in queste sequenze. (D) Il processo di decodifica prende questo insieme rumoroso e talvolta incompleto di letture di sequenziamento, corregge gli errori e le sequenze mancanti e decodifica le informazioni per recuperare i dati. (E) Riepilogo dei processi di sintesi commerciale e corrispondenti densità oligonucleotidiche stimate, come riportato in letteratura o dalle stesse società. La densità del nostro metodo elettrochimico è evidenziata in rosso scuro. Credito:Progressi scientifici , 10.1126/sciadv.abi6714
I genetisti possono archiviare i dati nel DNA sintetico come mezzo per l'archiviazione a lungo termine grazie alla sua densità, facilità di copia, longevità e sostenibilità. La ricerca nel campo è recentemente avanzata con nuovi algoritmi di codifica, automazione, conservazione e sequenziamento. Tuttavia, l'ostacolo più impegnativo nell'implementazione dello storage del DNA rimane il throughput di scrittura, che può limitare la capacità di storage dei dati. In un nuovo rapporto, Bichlien H. Nguyen e un team di scienziati della Microsoft Research e dell'informatica e dell'ingegneria dell'Università di Washington, Seattle, USA, hanno sviluppato il primo scrittore di archiviazione del DNA su scala nanometrica. Il team intendeva ridimensionare la densità di scrittura del DNA a 25 x 10 6 sequenze per centimetro quadrato, una capacità di archiviazione migliorata rispetto agli array di sintesi del DNA esistenti. Gli scienziati hanno scritto e decodificato con successo un messaggio nel DNA per stabilire un pratico sistema di archiviazione dei dati del DNA. I risultati sono ora pubblicati in Science Advances .
Archivi di DNA a lungo termine
L'attuale ritmo di generazione dei dati supera le capacità di archiviazione esistenti, il DNA è una soluzione promettente a questo problema con una densità pratica prevista di oltre 60 petabyte per centimetro cubo. Il materiale è durevole in una serie di condizioni, rilevante e facile da copiare, con la promessa di essere più sostenibile o più ecologico dei media commerciali. Durante il processo, i dati digitali sotto forma di sequenze di bit possono essere codificati in sequenze delle quattro basi naturali del DNA:guanina, adenina, tiamina e citosina, sebbene siano possibili anche basi aggiuntive. Il team può quindi scrivere le sequenze in forma molecolare tramite la sintesi di oligonucleotidi di DNA de novo per creare molecole specifiche basate su una serie di passaggi chimici ripetuti. Gli oligonucleotidi risultanti possono essere conservati e conservati dopo la sintesi. Per accedere ai dati, la memoria del DNA può essere amplificata utilizzando reazioni a catena della polimerasi e sequenziata per restituire le sequenze di base del DNA al dominio digitale, quindi le sequenze di base del DNA possono essere decodificate per recuperare la sequenza originale di bit.
Panoramica dell'array da 650 nm con un passo di 2 μm. (A) L'analisi agli elementi finiti della generazione e diffusione di acido anodico su un elettrodo di 650 nm di diametro con un pozzo da 200 nm è rappresentata con una vista in sezione trasversale lungo il piano y =x e (B) vista dall'alto verso il basso sul z =0 piano. I colori blu e giallo rappresentano rispettivamente le regioni con concentrazioni di acido relativamente basse e alte. (C) Una panoramica dell'array di sintesi del DNA su scala nanometrica con immagini di microscopia elettronica a scansione dell'array di elettrodi da 650 nm e vista ingrandita di un elettrodo. (D) Un'immagine fluorescente in cui il pozzo che circonda ciascun anodo attivato è modellato con fluoresceina AAA. Il diagramma del fumetto mostra quali elettrodi nel layout sono stati attivati. (E) Illustrazione dei pozzetti modellati con AAA-fluoresceina e AAA-AquaPhluor e (F) sovrapposizione di immagini corrispondenti dei due fluorofori all'estremità del DNA sintetizzato sulla stessa matrice di elettrodi da 650 nm. Credito:Progressi scientifici , 10.1126/sciadv.abi6714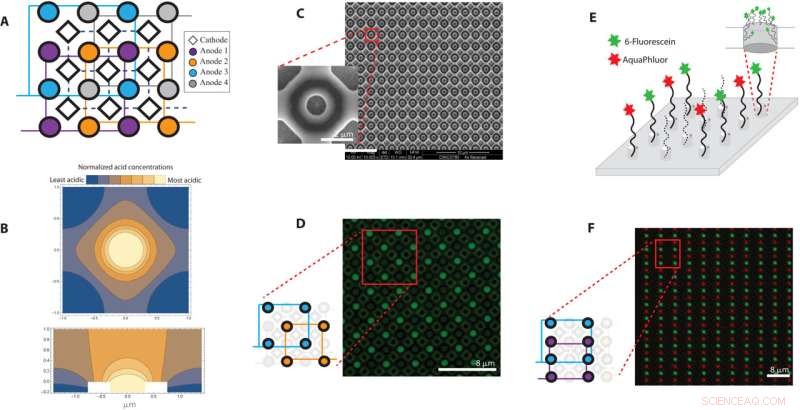
In questo studio, Nguyen et al. ha prodotto un array di elettrodi che ha dimostrato un controllo specifico dell'elettrodo indipendente della sintesi del DNA con dimensioni e passi degli elettrodi per stabilire una densità di sintesi di 25 milioni di oligonucleotidi per cm 2 . Questo valore è stimato come la densità dell'elettrodo richiesta per raggiungere l'obiettivo minimo di kilobyte al secondo di archiviazione dati nel DNA. Il team ha spinto lo stato dell'arte nel controllo chimico-elettronico e ha fornito prove sperimentali alla larghezza di banda di scrittura necessaria per l'archiviazione dei dati del DNA.
Il team ha introdotto un controller molecolare proof-of-concept sotto forma di un minuscolo meccanismo di scrittura di archiviazione del DNA su un chip. Il chip potrebbe racchiudere strettamente la sintesi del DNA a 3 ordini di grandezza in più rispetto a prima per ottenere una maggiore velocità di scrittura del DNA. Memorizzare le informazioni nel DNA alla scala necessaria per l'uso commerciale richiedeva due processi cruciali. Per prima cosa il team ha dovuto tradurre bit digitali (uno e zero) in filamenti di DNA sintetico che rappresentano bit con un software di codifica e un sintetizzatore di DNA. Quindi devono essere in grado di leggere e decodificare le informazioni sui suoi bit per recuperare nuovamente tali informazioni in forma digitale con un sequenziatore di DNA e un software di decodifica.
Sviluppo di array elettrochimici per funzionalità su scala nanometrica
Durante la sintesi tradizionale delle catene di DNA, gli scienziati utilizzano un metodo multifase noto come chimica della fosforamidite, in cui una catena di DNA può essere cresciuta in sequenza mediante l'aggiunta di basi di DNA. Ciascuna base di DNA contiene un gruppo di blocco per impedire aggiunte multiple di basi di DNA alla catena in crescita. All'attaccamento a una catena di DNA, l'acido può essere fornito nella configurazione per scindere il gruppo bloccante e innescare la catena di DNA per aggiungere la base successiva. Durante la sintesi elettrochimica del DNA, ogni punto nell'array contiene un elettrodo e quando viene applicata una tensione, viene generato acido sull'elettrodo di lavoro (anodo) per sbloccare le catene di DNA in crescita, mentre una base equivalente viene generata sul controelettrodo (catodo) . Il team ha impedito la diffusione dell'acido nella configurazione progettando un array di elettrodi, in cui ogni elettrodo funzionante attorno al quale si è verificata la formazione di acido durante la sintesi del DNA è stato immerso in un pozzo e circondato da quattro controelettrodi comuni, ovvero catodi che guidavano la formazione di basi, per confinare l'acido a regioni specifiche. Nguyen et al. verificato l'efficacia del progetto mediante l'analisi agli elementi finiti. Durante gli esperimenti, quando presentato in concentrazione sufficiente, l'acido ha sbloccato i nucleotidi legati alla superficie per consentire al nucleotide successivo di accoppiarsi. Utilizzando la configurazione di chip contenenti punti caratteristici per confinare gli acidi, hanno sviluppato array elettrochimici con quattro elettrodi singoli per regolare la sintesi del DNA. Il team ha quindi eseguito esperimenti con due basi etichettate in modo fluorescente in verde e rosso. Come prova del concetto, hanno mostrato la capacità del dispositivo di scrivere dati sintetizzando quattro filamenti di DNA unici, ciascuno lungo 100 basi con un messaggio codificato, senza errori.
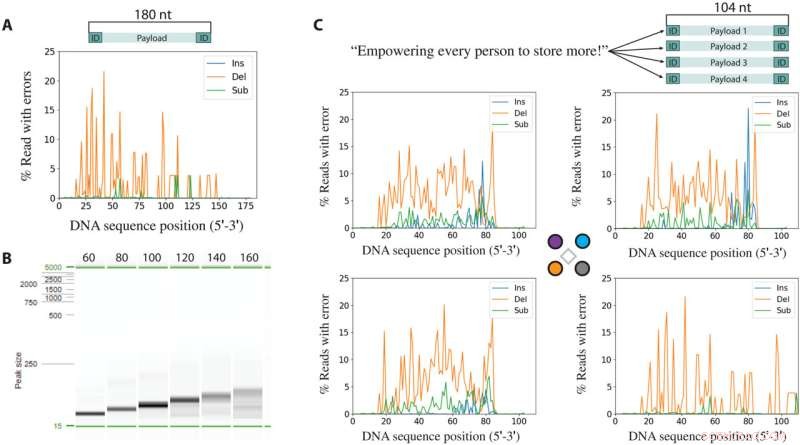
Errori derivanti dalla sintesi seguiti dal sequenziamento. (A) Inserzioni (Ins), eliminazioni (Del) e sostituzioni (Sub) per posizione per una sequenza di 180 basi sintetizzata e amplificata dalla PCR. (B) Immagine di elettroforesi di prodotti di sintesi dopo l'amplificazione PCR. (C) Messaggio codificato in 64 byte suddivisi in quattro sequenze univoche di 104 basi (in alto). Inserzioni, eliminazioni e sostituzioni per locus di ciascuna delle quattro sequenze nell'esecuzione della sintesi multiplex. In ogni grafico di analisi degli errori, le basi del terminale 20 alle estremità 3′ e 5′ provengono dai primer utilizzati nella PCR e non sono rappresentative degli errori sintetizzati. Credito:Progressi scientifici , 10.1126/sciadv.abi6714
Ridimensionamento dell'archiviazione dei dati del DNA con pozzetti per elettrodi su scala nanometrica. Minuscolo meccanismo di scrittura di archiviazione del DNA su un chip. Credito:Blog di ricerca Microsoft, Progressi scientifici , 10.1126/sciadv.abi6714
Prospettiva:sintesi di oligonucleotidi corti sull'array di elettrodi per l'archiviazione dei dati
Utilizzando la configurazione, Nguyen et al. ha anche dimostrato la sintesi spazialmente controllata di oligonucleotidi corti sulla matrice di elettrodi per valutare la lunghezza massima del DNA che potrebbe essere formato. Gli scienziati hanno creato una singola sequenza di DNA con 180 nucleotidi e prodotti di varie lunghezze amplificati dalla PCR dalla lunghezza completa degli oligonucleotidi. Man mano che l'amplicone si allungava, i prodotti PCR attesi apparivano più deboli e meno ben definiti, mentre gli ampliconi più corti mostravano bande più forti e ben definite indicative di errori di sintesi più elevati. Sulla base dei risultati, i ricercatori hanno selezionato la lunghezza della sequenza pari a 100 basi per facilitare la purificazione per fornire una dimostrazione pratica dell'archiviazione dei dati del DNA senza ulteriore ottimizzazione. In questo modo, il metodo proof-of-concept dimostrato in questo lavoro da Bichlien H. Nguyen e colleghi ha aperto la strada alla generazione in parallelo di sequenze di DNA uniche e su larga scala per l'archiviazione dei dati. Il lavoro ha superato i precedenti rapporti sulle sequenze di DNA sintetico denso per fornire una prima indicazione sperimentale per ottenere la larghezza di banda di scrittura richiesta per l'archiviazione dei dati a dimensioni delle caratteristiche su scala nanometrica. Gli scienziati si aspettano applicazioni immediate dei dispositivi nella tecnologia dell'informazione e prevedono le loro applicazioni pratiche nella scienza dei materiali, nella biologia sintetica e nei saggi di biologia molecolare su larga scala. + Esplora ulteriormente
© 2021 Rete Scienza X