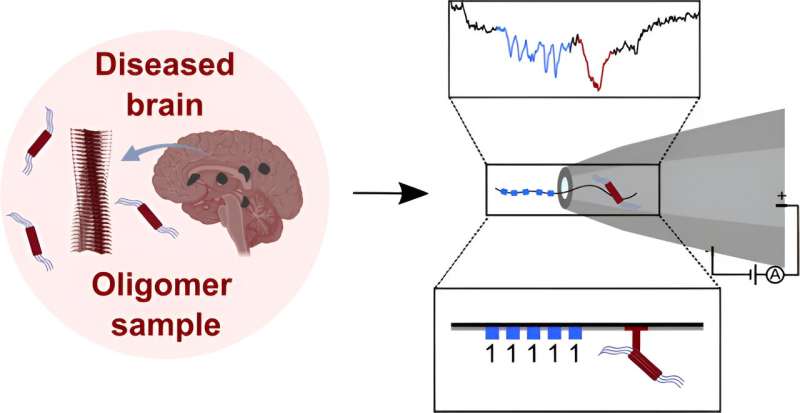
Un team di chimici, microbiologi e fisici dell’Università di Cambridge nel Regno Unito ha sviluppato un modo per utilizzare nanopori allo stato solido e codici a barre del DNA multiplex per identificare proteine mal ripiegate come quelle coinvolte nei disturbi neurodegenerativi nei campioni di sangue. Nel loro studio, riportato nel Journal of American Chemical Society , il gruppo ha utilizzato tecniche di codifica a barre del DNA multiplex per superare i problemi con le tecniche di filtraggio dei nanopori per isolare oligomeri dannosi.
Ricerche precedenti hanno dimostrato che la presenza di oligomeri dannosi nel cervello può portare al ripiegamento errato delle proteine associate a malattie neurodegenerative come il morbo di Parkinson e il morbo di Alzheimer. I ricercatori medici sono alla ricerca di un modo per rilevarli nel sangue al fine di diagnosticare una malattia neurodegenerativa e monitorarne la progressione una volta confermata.
Sfortunatamente, trovarli in miscele complesse come il sangue si è rivelato un compito arduo. Un approccio si è rivelato promettente, ovvero l’utilizzo di sensori a nanopori, ma ad oggi non sono in grado di monitorare gli oligomeri target mentre sfrecciano attraverso sensori a nanopori personalizzabili. In questo nuovo sforzo, il gruppo di ricerca ha superato questo problema utilizzando nanostrutture di DNA personalizzabili.
Nel loro lavoro, il gruppo di ricerca ha legato le proteine alle loro nanostrutture di DNA personalizzate come mezzo per creare una sorta di "codice a barre" che potrebbe essere utilizzato per identificare le molecole utilizzando i sensori a nanopori allo stato solido. Per utilizzare il codice a barre, hanno posizionato un frammento di DNA che era stato contrassegnato con una sostanza chimica che si lega solo agli oligomeri target, determinando un picco aggiuntivo, che ha consentito di isolare il target e la sua completa caratterizzazione.
Il team ha poi testato la propria tecnica utilizzando oligomeri di α-sinucleina, il tipo di proteine coinvolte nel ripiegamento nella malattia di Parkinson, ed è stato in grado di isolarli come desiderato in condizioni di laboratorio. Hanno anche misurato il tasso di formazione degli oligomeri. Hanno poi testato la tecnica in un ambiente più vicino alle condizioni biologiche e hanno scoperto che le prestazioni non ne risentivano. Suggeriscono che la loro tecnica offra la promessa di un futuro processo di screening per i pazienti a rischio di sviluppare disturbi neurodegenerativi.
Ulteriori informazioni: Sarah E. Sandler et al, Caratterizzazione digitale multiplex di oligomeri proteici mal ripiegati tramite nanopori a stato solido, Journal of the American Chemical Society (2023). DOI:10.1021/jacs.3c09335
Informazioni sul giornale: Giornale dell'American Chemical Society
© 2023 Rete Scienza X