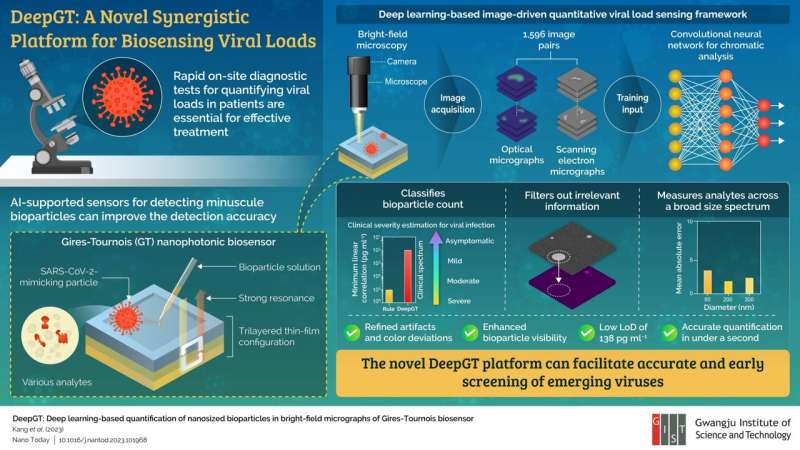
Studi recenti hanno scoperto che i biosensori Gires-Tournois (GT), un tipo di risonatore nanofotonico, possono rilevare minuscole particelle virali e produrre micrografie colorate (immagini scattate al microscopio) di cariche virali. Ma soffrono di artefatti visivi e di non riproducibilità, che ne limitano l'utilizzo.
In una recente svolta, un team internazionale di ricercatori, guidato dal professor Young Min Song della Scuola di ingegneria elettrica e informatica presso l’Istituto di scienza e tecnologia di Gwangju in Corea, ha sfruttato l’intelligenza artificiale (AI) per superare questo problema. Il loro lavoro è stato pubblicato su Nano Today .
Le tecnologie diagnostiche rapide e in loco per identificare e quantificare i virus sono essenziali per pianificare strategie di trattamento per i pazienti infetti e prevenire l’ulteriore diffusione dell’infezione. La pandemia di COVID-19 ha evidenziato la necessità di test diagnostici accurati ma decentralizzati che non comportino processi complessi e dispendiosi in termini di tempo necessari per i test di laboratorio convenzionali.
Uno strumento diagnostico popolare per quantificare le cariche virali è l’imaging microscopico a campo chiaro. Tuttavia, le piccole dimensioni (~ 100 nm) e il basso indice di rifrazione (~ 1,5, lo stesso di un vetrino da microscopio) delle bioparticelle come i virus spesso rendono difficile la loro stima accurata e aumentano il limite di rilevamento (la più bassa concentrazione di carica virale che può essere rilevato in modo affidabile).
Nel loro nuovo studio, il team ha proposto uno strumento di biosensing sinergico chiamato “DeepGT”, che può sfruttare i vantaggi delle piattaforme di rilevamento GT e unirli con algoritmi basati sul deep learning per quantificare accuratamente le bioparticelle su scala nanometrica, compresi i virus, senza la necessità di campioni complessi. metodi di preparazione.
"Abbiamo progettato DeepGT per valutare oggettivamente la gravità di un'infezione o di una malattia. Ciò significa che non dovremo più fare affidamento esclusivamente su valutazioni soggettive per la diagnosi e l'assistenza sanitaria, ma avremo invece un approccio più accurato e basato sui dati per guidare le strategie terapeutiche ," spiega il Prof. Song, rivelando la motivazione alla base del loro studio.
Il team ha progettato un biosensore GT con una configurazione a film sottile a tre strati e lo ha biofunzionalizzato per consentire il rilevamento colorimetrico in seguito all'interazione con gli analiti target. Le capacità di rilevamento sono state verificate simulando il meccanismo di legame tra le cellule ospiti e il virus utilizzando bioparticelle appositamente preparate che imitavano la SARS-CoV-2, il ceppo di coronavirus che ha causato la pandemia di COVID-19.
Successivamente, i ricercatori hanno addestrato una rete neurale convoluzionale (CNN) utilizzando oltre un migliaio di micrografie ottiche ed elettroniche a scansione della superficie del biosensore GT con diversi tipi di nanoparticelle. Hanno scoperto che DeepGT era in grado di perfezionare gli artefatti visivi associati alla microscopia in campo chiaro ed estrarre informazioni rilevanti, anche a concentrazioni virali fino a 138 pg ml –1 .
Inoltre, ha determinato il conteggio delle bioparticelle con elevata precisione, caratterizzato da un errore assoluto medio di 2,37 su 1.596 immagini rispetto a 13,47 per gli algoritmi basati su regole, in meno di un secondo. Potenziato dalle prestazioni delle CNN, il sistema di biosensing può anche indicare la gravità dell'infezione da asintomatica a grave in base alla carica virale.
DeepGT presenta quindi un modo efficiente e preciso per schermare virus in un'ampia gamma di dimensioni senza essere ostacolato dal limite minimo di diffrazione nella luce visibile. "Il nostro approccio fornisce una soluzione pratica per il rilevamento e la gestione rapidi delle minacce virali emergenti, nonché per il miglioramento della preparazione della sanità pubblica riducendo potenzialmente l'onere complessivo dei costi associati alla diagnostica", conclude il prof. Song.
Ulteriori informazioni: Jiwon Kang et al, DeepGT:Quantificazione basata sull'apprendimento profondo di bioparticelle di dimensioni nanometriche in micrografie in campo chiaro del biosensore Gires-Tournois, Nano Today (2023). DOI:10.1016/j.nantod.2023.101968
Informazioni sul giornale: Nano oggi
Fornito dall'Istituto di scienza e tecnologia di Gwangju