Più filamenti di DNA complementari possono essere ricotti termicamente nelle entità desiderate per progettare nanostrutture di DNA. In un nuovo studio ora pubblicato su Nature Nanotechnology , Caroline Rossi-Gendron e un team di ricercatori in chimica, scienza dei materiali e biologia in Francia e Giappone hanno utilizzato un tampone privo di magnesio contenente cloruro di sodio, cocktail complessi di filamenti di DNA e proteine per autoassemblarsi isotermamente a temperatura ambiente o fisiologica in nanostrutture definite dall'utente tra cui nanogriglie, origami di DNA e assemblaggi di piastrelle a filamento singolo.
Questo autoassemblaggio si basava sulla termodinamica, procedendo attraverso molteplici percorsi di ripiegamento per creare nanostrutture altamente configurabili. Il metodo ha consentito l'autoselezione della forma più stabile in un ampio pool di filamenti di DNA competitivi. È interessante notare che gli origami di DNA possono spostarsi isotermicamente da una forma inizialmente stabile a una radicalmente diversa attraverso uno scambio di filamenti costitutivi. Ciò ha ampliato la raccolta di forme e funzioni ottenute tramite l'autoassemblaggio isotermico per creare le basi per nanomacchine adattive e facilitare la scoperta di nanostrutture evolutive.
L'autoassemblaggio avviene quando entità presenti in natura o progettate razionalmente possono incorporare le informazioni necessarie per interagire spontaneamente e auto-organizzarsi in sovrastrutture funzionali di interesse. Tipicamente, i materiali sintetici autoassemblati risultano dall'organizzazione di un singolo componente ripetitivo per creare un assemblaggio sopramolecolare stabile contenente micelle o cristalli colloidali con un insieme prescritto di proprietà utili. Tali costrutti hanno una riconfigurabilità limitata, il che rende estremamente difficile produrre le strutture desiderate.
La nanotecnologia del DNA strutturale esplora il principio dell'accoppiamento di basi dipendente dalla sequenza tra singoli filamenti di DNA sintetico per superare questa sfida e assemblare sovrastrutture diverse ed elaborate di forma, dimensione e specificità funzionale previste su larga scala con una gamma di applicazioni. Le strutture multicomponente sono generalmente derivate da un processo di ricottura termica, in cui la miscela di DNA viene inizialmente riscaldata al di sopra della temperatura di fusione e raffreddata lentamente per evitare trappole cinetiche e garantire l'ibridazione del DNA specifica per sequenza.
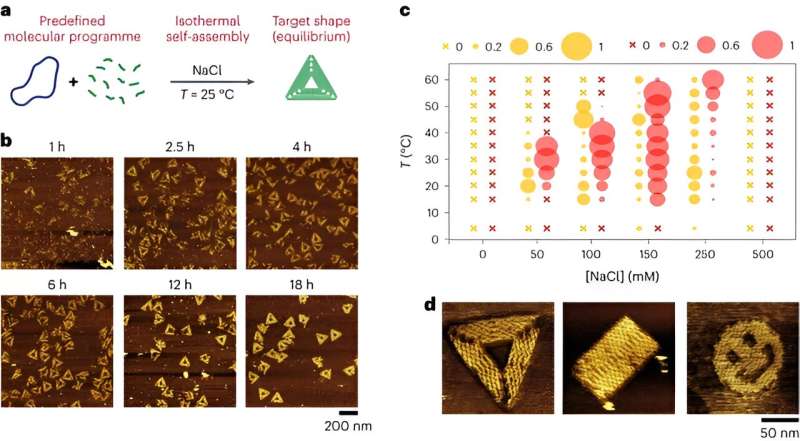
La ricottura termica può ostacolare la possibilità di formazione spontanea di nanostrutture in condizioni fisse. In questo lavoro, Rossi-Gendron e colleghi hanno quindi descritto che il metodo principale della nanotecnologia del DNA strutturale dipende dallo stesso principio di autoassemblaggio del DNA isotermico generico per creare nanostrutture di DNA elaborate definite dall'utente come origami di DNA e nanogriglie di DNA. Il team di ricerca ha studiato la complessità strutturale dei progetti di origami di DNA e delle nanogriglie autoripetenti utilizzando la microscopia a forza atomica per rivelare la molteplicità dei percorsi di piegatura nelle forme di origami 2D autoassemblanti.
Origami di DNA tramite autoassemblaggio in cloruro di sodio
Il team ha completato una serie di esperimenti in un ambiente di autoassemblaggio isotermico regolato termodinamicamente per completare la trasformazione della forma. Hanno ottenuto questo risultato assemblando una miscela di origami di DNA senza pretrattamento termico e incubato i costrutti per diverse ore in un tampone convenzionale. Come osservato in precedenza, indipendentemente dal tempo di incubazione, i risultati non hanno mostrato la formazione di oggetti dalla forma corretta.
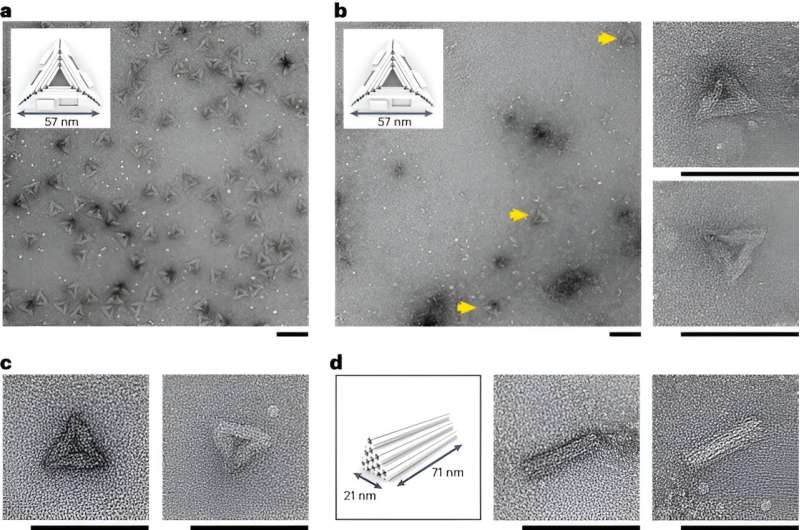
Il team ha optato per un tampone alternativo integrato con sali monovalenti per promuovere lo scambio e la riconfigurazione dei punti metallici e notare la notevole formazione di triangoli affilati correttamente piegati a temperatura ambiente nel giro di poche ore. Questi risultati erano coerenti per tutte le concentrazioni saline intermedie. I ricercatori hanno mostrato come l'autoassemblaggio isotermico nel buffer potrebbe essere guidato elettrostaticamente per generare una varietà di nanostrutture personalizzate in un'ampia finestra di temperatura.
Hanno esplorato il concetto di autoassemblaggio isotermico degli origami 3D per evidenziare la possibilità di autoassemblaggio spontaneo a temperatura ambiente o corporea senza pretrattamento termico per creare una varietà di morfologie per esemplificare la versatilità dell'autoassemblaggio. Tuttavia, la resa molto bassa dei costrutti ne ha evidenziato l'attuale limite che può essere superato ottimizzando la progettazione della nanostruttura.
Rossi-Gendron e colleghi hanno studiato ulteriormente i meccanismi di autoassemblaggio isotermico ideando un metodo per seguire il percorso di ripiegamento degli origami di DNA 2D in tempo reale. Il lavoro ha dimostrato che il raggiungimento della struttura di equilibrio per un singolo origami non dipendeva da uno specifico percorso di piegatura, ma si basava invece su più percorsi, fino a raggiungere la forma di equilibrio target.
Le strutture parzialmente piegate hanno mostrato diversi stati di piegatura iniziali, il che implica che più percorsi di piegatura non si basavano sull'autoassemblaggio assistito dalla superficie. I risultati concludono che la formazione di origami isotermici è un processo regolato termodinamicamente in base al quale le strutture raggiungono uno stato di equilibrio tramite l'autoassemblaggio. Dopo aver esposto le forme degli origami a una serie di elementi competitivi, il team ha notato come l'autoassemblaggio ha portato all'evoluzione spontanea dalla forma dell'origami a un costrutto stabile radicalmente diverso per creare un risultato di cambiamento di forma termodinamicamente favorito.
In questo modo, Rossi-Gendron e colleghi hanno utilizzato un tampone salino generico e una miscela altamente multicomponente di filamenti di DNA per autoassemblarsi spontaneamente a temperatura costante in un intervallo di temperature per formare oggetti dalla forma corretta come origami o nanogriglie di DNA. Hanno raggiunto questi risultati a temperatura ambiente per l'autoassemblaggio graduale guidato termodinamicamente. I risultati hanno indicato la possibilità di funzioni dinamiche in ambienti ambientali e sistemi viventi con temperature fisse per la scoperta di nanostrutture utilizzando grandi librerie di componenti del DNA.
Ulteriori informazioni: Caroline Rossi-Gendron et al, Autoassemblaggio isotermico di nanostrutture di DNA multicomponente ed evolutive, Nature Nanotechnology (2023). DOI:10.1038/s41565-023-01468-2
Paul W. K. Rothemund, Ripiegare il DNA per creare forme e modelli su scala nanometrica, Natura (2006). DOI:10.1038/nature04586
Informazioni sul giornale: Nanotecnologia naturale , Natura
© 2023 Rete Scienza X