Le librerie combinatorie sono una componente chiave del kit di strumenti del chimico per lo screening dei ligandi. Le librerie combinatorie dinamiche aggiungono una nuova dimensione collegando sintesi e screening. Ora, Scienziati britannici hanno sviluppato una libreria combinatoria dinamica per lo screening di strutture di ligandi supramolecolari che riconoscono le superfici proteiche e potrebbero interferire con l'interazione proteina-proteina. Lo studio è stato pubblicato su Giornale europeo di chimica organica .
La chimica combinatoria è stata adottata dall'industria farmaceutica perché consente ai chimici di creare grandi quantità di molecole e testarle per le proprietà desiderate, Per esempio, affinità di legame a un bersaglio terapeuticamente importante. Sintesi e screening sono collegati nella chimica combinatoria dinamica poiché le librerie combinatorie dinamiche possono spostare la distribuzione del loro prodotto in risposta al legame del bersaglio. Questo processo di amplificazione del prodotto genera strutture supramolecolari altrimenti di difficile accesso e consente la rapida identificazione di architetture di legame, Per esempio, inibitori che si legano al sito attivo di un enzima.
Però, le superfici proteiche sono grandi strutture tridimensionali con chiazze di diversa idrofobicità, acidità, e basicità, trovano in posizioni disparate sulla loro superficie. Perciò, lo screening degli inibitori delle interazioni proteina-proteina deve coinvolgere ligandi più complessi di quelli necessari per il riconoscimento del sito attivo "Avevamo bisogno di identificare una chimica reversibile e una metodologia analitica che fosse compatibile con l'idea di fare chimica combinatoria dinamica per il riconoscimento della superficie delle proteine, " dice Andrew J. Wilson, l'autore corrispondente dello studio che è stato condotto presso l'Università di Leeds.
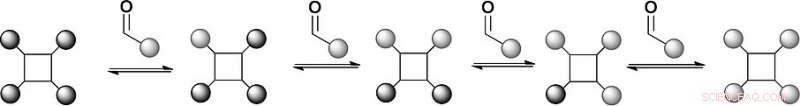
Gli scienziati hanno scelto un'impalcatura di tetrafenilporfirina con quattro idrazidi e hanno sostituito le benzaldeidi come bracci di riconoscimento che si collegherebbero in modo reversibile con le idrazidi per formare idrazoni. Le porfirine sono impalcature stabilite per il riconoscimento della superficie proteica, ma la chimica dell'idrazone è sinteticamente impegnativa. "Abbiamo un'impalcatura con quattro idrazoni in modo da poter scambiare chimica su quattro posizioni diverse, " dice Wilson. Inoltre, la chimica reversibile che forma idrazone di solito avviene a un pH acido, ma le proteine preferiscono un pH neutro. Gli scienziati hanno affrontato questo problema aggiungendo un catalizzatore a base di anilina, che ha spinto la reazione di scambio di idrazone verso l'equilibrio termodinamico a pH prossimo al neutro.
Nella prima configurazione di questa libreria di modelli, i ricercatori hanno provato due ligandi aldeidici sostituiti, che ha formato un equilibrio con l'idrazone derivato dalla benzaldeide. L'analisi della spettrometria di massa ha rivelato che la distribuzione del prodotto era all'equilibrio termodinamico, e i ligandi infatti scambiati per reazione reversibile. Ciò significa che sono state stabilite le condizioni per una libreria combinatoria dinamica per il riconoscimento della superficie delle proteine. Prossimo, gli scienziati hanno in programma di elaborare questo approccio per studiare la selezione diretta dalle proteine dei mimetici della superficie delle proteine.