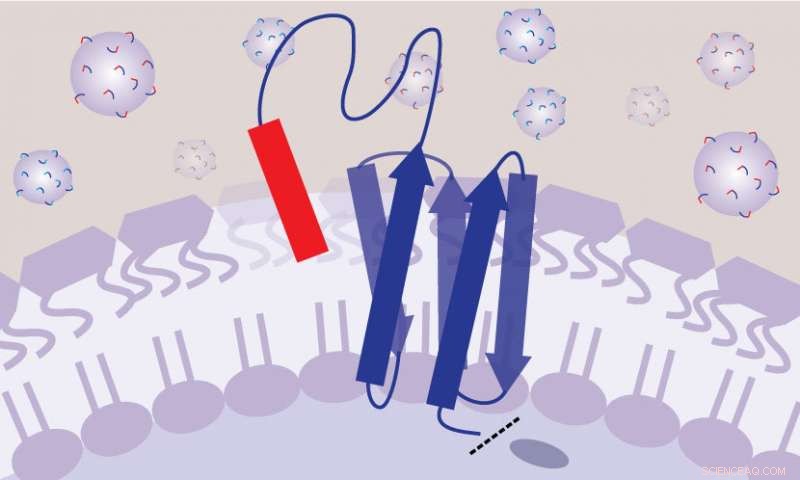
Nel metodo SLAY, ogni batterio è geneticamente modificato per produrre una molecola sulla sua superficie cellulare che è in parte peptide e in parte tether, come un tetherball del parco giochi. Questa disposizione consente ai peptidi di imitare i farmaci fluttuanti nel corpo umano. Credito:Ashley Tucker
I biologi dell'Università del Texas ad Austin hanno sviluppato un metodo per lo screening rapido di centinaia di migliaia di potenziali farmaci per combattere le infezioni, un'innovazione che promette di combattere la crescente piaga dei batteri resistenti agli antibiotici. Il metodo prevede l'ingegneria dei batteri per produrre e testare molecole potenzialmente tossiche per se stesse.
Una descrizione del metodo appare nell'edizione cartacea del 25 gennaio della rivista Cellula .
Nessuna nuova classe di antibiotici è stata scoperta in 40 anni:molti di quelli più accessibili in natura sono già stati trovati, e il processo per crearne e testarne di nuovi da zero è lento e laborioso, ma la medicina moderna ne ha assolutamente bisogno. Secondo l'Organizzazione Mondiale della Sanità, gli antibiotici hanno aggiunto circa 20 anni alla durata media della vita umana. Ma i loro benefici protettivi stanno svanendo man mano che i batteri evolvono la resistenza agli antibiotici.
Nella loro prova di concetto, la squadra dell'UT Austin, guidato da Bryan Davies, proiettato circa 800, 000 molecole chiamate peptidi per vedere se avevano effetti antimicrobici, nel senso che hanno ucciso i batteri nocivi. Di quelli, diverse migliaia di batteri E. coli uccisi, rendendoli potenziali contatti per gli antibiotici. Alcuni antibiotici attualmente in uso sono peptidi. Sarà necessaria una ricerca di follow-up per determinare quale, se del caso, delle migliaia di nuovi lead sono veramente efficaci e sicuri negli esseri umani, ma i ricercatori hanno dimostrato che almeno una di queste molecole, soprannominato P7, uccide anche altre forme di batteri patogeni ed è sicuro nei topi.
Con questo metodo, chiamato SLAY (Display antimicrobico localizzato in superficie), una persona può eseguire lo screening di centinaia di migliaia di peptidi simili in modo più rapido ed economico rispetto ai metodi esistenti. Davies vorrebbe che il metodo diventasse uno strumento standard nella ricerca globale di nuovi antibiotici.
"E se avessimo un migliaio di gruppi che usano tutti questo sistema per seguire i propri interessi e i propri peptidi?" ha detto Davies, assistente professore di bioscienze molecolari. "Una volta abilitata una community di quelle dimensioni, quindi penso che tu abbia maggiori possibilità di trovare effettivamente un nuovo antibiotico che funzioni".
Un progresso chiave in questo lavoro è stato capire come far produrre ai batteri molecole che potrebbero essere tossiche per se stessi e controllare come queste molecole interagiscono con i loro batteri ospiti.
"Abbiamo pensato, non sarebbe fantastico se un batterio potesse sintetizzare il composto per noi, perché i batteri sono economici e facili da coltivare, e poi testare il composto su se stesso e riferire e dirci, era un antimicrobico o no?" ha detto Davies.
La loro soluzione è stata quella di ingegnerizzare geneticamente i batteri per produrre una molecola sulla superficie cellulare che è in parte peptide e in parte tether, come una palla da gioco e il suo tether, con un'estremità fissata alla membrana cellulare e l'altra estremità libera di fluttuare. Ciò consente al peptide di muoversi e di entrare in contatto con la superficie della cellula batterica, come se fluttuasse liberamente come una droga nel tuo sangue, ma senza interagire con altri batteri vicini.
Assicurandosi che ogni versione del tetherball interagisca solo con i batteri che l'hanno prodotta, i ricercatori potrebbero quindi fare un grande salto di efficienza. Potrebbero creare centinaia di migliaia di ceppi di batteri, ognuno geneticamente modificato per produrre una versione leggermente diversa del tetherball, e mettere tutti questi ceppi nella stessa provetta per farli crescere. Eseguendo centinaia di migliaia di esperimenti contemporaneamente, il loro metodo consente di risparmiare un'enorme quantità di spazio, tempo e costo.
Parte di questo processo si basa su una tecnica sviluppata da George Georgiou di UT Austin negli anni '90 che induce i batteri a produrre proteine o peptidi sulle loro superfici.
Per scoprire quali tetherball (peptidi) eliminano i loro ospiti, gli scienziati utilizzano il sequenziamento dei geni per identificare quali versioni vengono prodotte dai batteri all'inizio e quali alla fine.
In seguito alla scoperta che P7 uccide i patogeni, il team ora prevede di creare migliaia di sottili variazioni di questa molecola, detti derivati, ed eseguili attraverso lo stesso processo di screening per cercare una versione ancora più efficace.
La borsista post-dottorato Ashley Tucker ha guidato il lavoro sperimentale per dimostrare l'uso della piattaforma.
Davies, Tucker e UT Austin hanno depositato domande di brevetto per il metodo SLAY e per le sequenze genetiche specifiche per le migliaia di peptidi antimicrobici che hanno scoperto finora.