
Le proteine progettate con uno strumento software ultrarapido chiamato ProteinMPNN avevano molte più probabilità di piegarsi come previsto. Credito:Ian Haydon, UW Medicine Institute for Protein Design
Negli ultimi due anni, l'apprendimento automatico ha rivoluzionato la previsione della struttura delle proteine. Ora, tre articoli in Scienza descrivere una rivoluzione simile nella progettazione delle proteine.
Nei nuovi articoli, i biologi della University of Washington School of Medicine mostrano che l'apprendimento automatico può essere utilizzato per creare molecole proteiche in modo molto più accurato e rapido di quanto fosse possibile in precedenza. Gli scienziati sperano che questo progresso porti a molti nuovi vaccini, trattamenti, strumenti per la cattura del carbonio e biomateriali sostenibili.
"Le proteine sono fondamentali in tutta la biologia, ma sappiamo che tutte le proteine che si trovano in ogni pianta, animale e microbo costituiscono molto meno dell'uno per cento di ciò che è possibile. Con questi nuovi strumenti software, i ricercatori dovrebbero essere in grado di trovare soluzioni a lungo -sfide in corso in medicina, energia e tecnologia", ha affermato l'autore senior David Baker, professore di biochimica presso la University of Washington School of Medicine e destinatario del Breakthrough Prize 2021 in Life Sciences.
Le proteine sono spesso indicate come i "mattoni della vita" perché sono essenziali per la struttura e la funzione di tutti gli esseri viventi. Sono coinvolti praticamente in ogni processo che avviene all'interno delle cellule, inclusa la crescita, la divisione e la riparazione. Le proteine sono costituite da lunghe catene di sostanze chimiche chiamate amminoacidi. La sequenza di amminoacidi in una proteina determina la sua forma tridimensionale. Questa forma intricata è fondamentale per il funzionamento della proteina.
Recentemente, potenti algoritmi di apprendimento automatico, tra cui AlphaFold e RoseTTAFold, sono stati addestrati per prevedere le forme dettagliate delle proteine naturali basate esclusivamente sulle loro sequenze di amminoacidi. L'apprendimento automatico è un tipo di intelligenza artificiale che consente ai computer di apprendere dai dati senza essere programmati in modo esplicito. L'apprendimento automatico può essere utilizzato per modellare problemi scientifici complessi che sono troppo difficili da comprendere per gli esseri umani.
Per andare oltre le proteine presenti in natura, i membri del team di Baker hanno suddiviso la sfida della progettazione delle proteine in tre parti e hanno utilizzato nuove soluzioni software per ciascuna.
L'intelligenza artificiale ha allucinato questi assemblaggi proteici simmetrici, in un modo simile ad altri A.!. strumenti generativi che producono output sulla base di semplici prompt. Credito:Ian Haydon, UW Medicine Institute for Protein Design
Innanzitutto, deve essere generata una nuova forma proteica. In un articolo pubblicato il 21 luglio sulla rivista Scienza , il team ha dimostrato che l'intelligenza artificiale può generare nuove forme proteiche in due modi. La prima, soprannominata "allucinazione", è simile a DALL-E o ad altre IA generative. strumenti che producono output sulla base di semplici prompt. La seconda, denominata "inpainting", è analoga alla funzione di completamento automatico che si trova nelle moderne barre di ricerca.
In secondo luogo, per accelerare il processo, il team ha ideato un nuovo algoritmo per la generazione di sequenze di amminoacidi. Descritto nel numero del 15 settembre di Scienza , questo strumento software, chiamato ProteinMPNN, viene eseguito in circa un secondo. È più di 200 volte più veloce del miglior software precedente. I risultati sono superiori agli strumenti precedenti e il software non richiede personalizzazione da parte di esperti per l'esecuzione.
"Le reti neurali sono facili da addestrare se si dispone di una tonnellata di dati, ma con le proteine non abbiamo tutti gli esempi che vorremmo. Abbiamo dovuto entrare e identificare quali caratteristiche di queste molecole sono le più importanti. è stato un po' di tentativi ed errori", ha affermato lo scienziato del progetto Justas Dauparas, un borsista post-dottorato presso l'Institute for Protein Design
In terzo luogo, il team ha utilizzato AlphaFold, uno strumento sviluppato da DeepMind di Alphabet, per valutare in modo indipendente se le sequenze di amminoacidi che hanno inventato potevano piegarsi nelle forme previste.
"Il software per la previsione delle strutture proteiche fa parte della soluzione, ma da solo non può inventare nulla di nuovo", ha spiegato Dauparas.
"ProteinMPNN è per la progettazione delle proteine ciò che AlphaFold era per la previsione della struttura delle proteine", ha aggiunto Baker.

Dettaglio di una proteina progettata utilizzando uno strumento rapido chiamato ProteinMPNN, un altro progresso nell'uso dell'intelligenza artificiale e dell'apprendimento automatico nella progettazione delle proteine. Credito:Ian Haydon, UW Medicine Institute for Protein Design
In un altro articolo apparso su Scienza Il 15 settembre, un team del laboratorio Baker ha confermato che la combinazione di nuovi strumenti di apprendimento automatico potrebbe generare in modo affidabile nuove proteine che funzionano in laboratorio.
"Abbiamo scoperto che le proteine prodotte utilizzando ProteinMPNN avevano molte più probabilità di ripiegarsi come previsto e potremmo creare assemblaggi proteici molto complessi utilizzando questi metodi", ha affermato lo scienziato del progetto Basile Wicky, un borsista post-dottorato presso l'Institute for Protein Design.
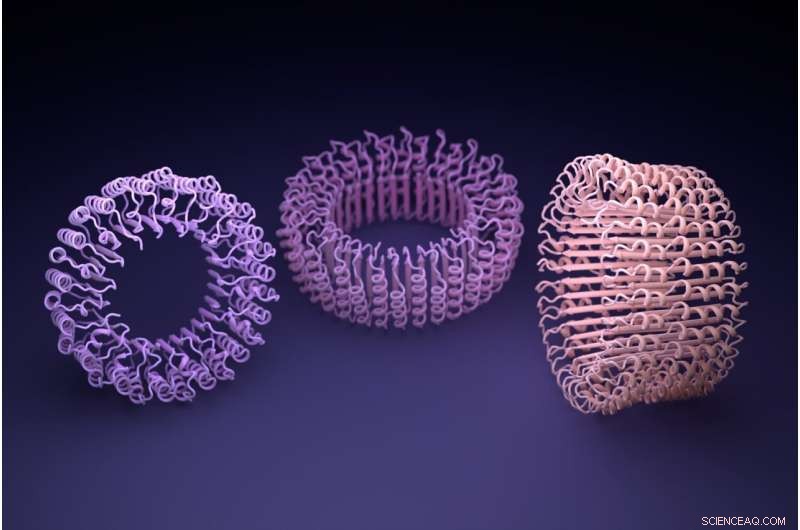
Tra le nuove proteine prodotte c'erano anelli su nanoscala che secondo i ricercatori potrebbero diventare parti per nanomacchine personalizzate. Sono stati usati microscopi elettronici per osservare gli anelli, che hanno diametri circa un miliardo di volte più piccoli di un seme di papavero.
"Questo è l'inizio dell'apprendimento automatico nella progettazione delle proteine. Nei prossimi mesi lavoreremo per migliorare questi strumenti per creare proteine ancora più dinamiche e funzionali", ha affermato Baker.
Le risorse informatiche per questo lavoro sono state donate da Microsoft e Amazon Web Services. + Esplora ulteriormente