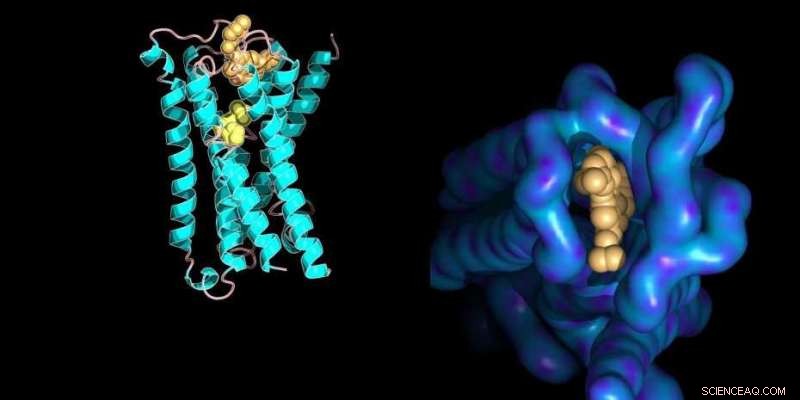
Modello molecolare di un recettore di membrana proteico legato ai suoi ligandi molecolari con dettagli strutturali del sito di legame allosterico (a destra). Credito:Ilaria Piazza / ETH Zurigo / Voce database PDB 4MQT
I ricercatori dell'ETH di Zurigo hanno utilizzato un nuovo approccio per scoprire interazioni precedentemente sconosciute tra proteine e piccole molecole metaboliche nelle cellule batteriche. La tecnica può essere utilizzata anche per testare l'effetto dei farmaci.
La scienza ora ha una nuova "omica" - un'interattomica, che si occupa delle interazioni tra proteine e piccole molecole. Essendosi precedentemente concentrato sulla genomica o sulla proteomica, i biologi del sistema possono ora aggiungere l'interattomica proteina-metabolita al mix. La genomica si occupa dell'analisi sistematica di tutti i geni di un organismo, mentre la proteomica si occupa dell'intero insieme di proteine all'interno di un'unità biologica.
Con l'interattomica proteina-metabolita, il gruppo guidato da Paola Picotti, professore di Biologia dei sistemi molecolari al Politecnico di Zurigo, ha ora aggiunto un altro livello agli "omici" esistenti. I ricercatori hanno recentemente pubblicato uno studio sulla rivista Cell, in cui per la prima volta hanno analizzato e quantificato sistematicamente le interazioni di tutte le proteine con i metaboliti (piccole molecole metaboliche) a livello dell'intero proteoma, stabilire i rapporti tra loro.
Diversi siti di taglio
I ricercatori hanno dimostrato quante proteine ed enzimi presenti in una cellula batterica di E. coli interagiscono con i metaboliti. Fare così, hanno usato un approccio noto come proteolisi limitata (LiP), combinato con l'analisi spettrometrica di massa.
I ricercatori hanno estratto il fluido cellulare, che contiene proteine, dalle cellule batteriche. Hanno quindi aggiunto un metabolita a ciascun campione e gli hanno permesso di interagire con le proteine. Finalmente, tagliano le proteine in pezzi più piccoli (peptidi) usando "forbici molecolari". In totale, i ricercatori hanno testato in questo modo 20 diversi metaboliti e le loro interazioni con le proteine.
Quando una proteina interagisce con un metabolita, se si deposita nel sito attivo della proteina o si attacca a un altro sito, la struttura proteica è alterata. Le "forbici molecolari" poi lo tagliano in punti diversi dalla struttura originaria, determinando un diverso insieme di peptidi.
Utilizzando lo spettrometro di massa, i ricercatori hanno misurato tutti i pezzi presenti nel campione e hanno inserito i dati ottenuti in un computer per ricostruire le differenze e i cambiamenti strutturali, e dove nella proteina questi sono localizzati.
Scoperte centinaia di nuove interazioni
Conoscenza dell'interattoma proteina-metabolita (le interazioni tra proteine e metaboliti e le reti molecolari (segnale) che ne fanno parte), era in precedenza molto limitato rispetto a quanto si sapeva sulle interazioni tra proteine diverse o tra proteine e DNA o RNA. Lo studio ha ora notevolmente aumentato questa conoscenza.
Utilizzando questo approccio con E. coli, Picotti e il suo team hanno scoperto circa 1, 650 diverse interazioni proteina-metabolita, di cui oltre 1, 400 erano precedentemente sconosciuti. Sono venuti alla luce anche migliaia di siti di legame su proteine a cui i metaboliti possono legarsi. "Sebbene il metabolismo di E. coli e delle molecole associate sia già molto noto, siamo riusciti a scoprire molte nuove interazioni e i corrispondenti siti di legame, " dice Picotti. Questo dimostra l'enorme potenziale del metodo:"I dati che produciamo con questa tecnica aiuteranno a identificare nuovi meccanismi regolatori, enzimi sconosciuti e nuove reazioni metaboliche nella cellula".
Il cambiamento strutturale regola l'attività
Nel loro studio, i ricercatori mostrano anche che piccole molecole metaboliche preferiscono legarsi (e quindi regolare) proteine la cui concentrazione è più o meno costante nel tempo. Ciò suggerisce che il legame dei metaboliti alle proteine e i cambiamenti nelle concentrazioni proteiche sono due percorsi complementari attraverso i quali le cellule regolano l'attività proteica.
Le proteine possono essere attivate o inattivate in tempi relativamente brevi da un cambiamento strutturale mediato dai metaboliti. "Questo tipo di cambiamento strutturale può essere invertito più rapidamente, " spiega Picotti. Dal punto di vista della cellula, questo spesso ha senso, perché utilizzare il percorso che comporta un cambiamento di concentrazione significa che la cellula deve smantellare o ricostruire le proteine, che costa più tempo, energia e risorse.
Picotti e i suoi colleghi sono stati anche in grado di dimostrare che molti enzimi sono meno esigenti di quanto si pensasse in precedenza:possono apparentemente legarsi e alterare chimicamente diversi metaboliti. Fino ad ora, si presumeva che gli enzimi fossero per lo più specifici di poche molecole molto simili.
L'industria farmaceutica è molto interessata al nuovo approccio, in quanto può essere utilizzato per testare l'interazione dei farmaci con le proteine cellulari e identificare i bersagli di un farmaco. I ricercatori potrebbero studiare a quali proteine e a quali siti si lega questo farmaco, come altera la loro struttura e quindi influenza la loro attività. Ciò faciliterebbe e accelererebbe i test e lo sviluppo di nuovi farmaci.