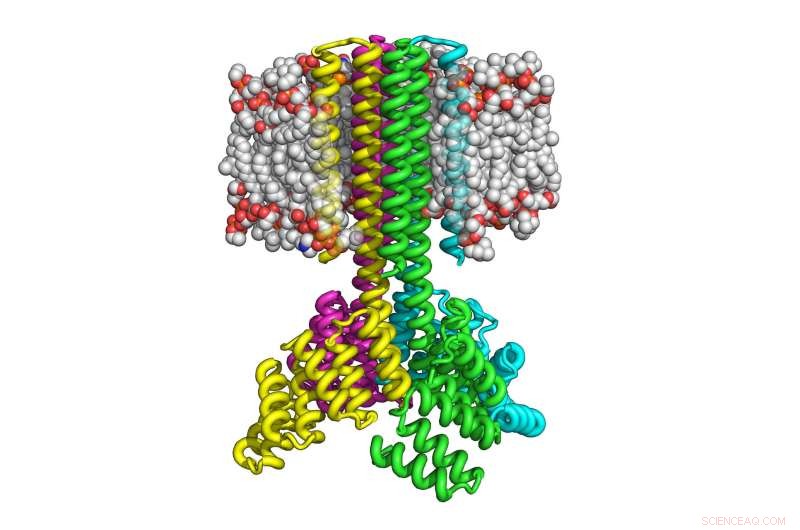
Questa illustrazione mostra come quattro copie della proteina transmembrana progettata al computer si sono assemblate per formare un tetramero a forma di razzo con un'ampia base citoplasmatica che si incanala in otto eliche transmembrana e che si orienta correttamente nella membrana. Credito:University of Washington Institute for Protein Design
Ora è possibile creare complessi, proteine transmembrana progettate da zero, gli scienziati riferiscono questa settimana. Il vantaggio, guidato da ingegneri molecolari presso l'Università di Washington Institute for Protein Design, consentirà ai ricercatori di creare proteine transmembrana non presenti in natura per svolgere compiti specifici.
Nel mondo vivente, le proteine transmembrana si trovano incorporate nella membrana di tutte le cellule e degli organelli cellulari. Sono essenziali per il loro normale funzionamento. Per esempio, molte proteine transmembrana naturali fungono da gateway per il movimento di sostanze specifiche attraverso una membrana biologica. Alcune proteine transmembrana ricevono o trasmettono segnali cellulari. A causa di tali ruoli, molti farmaci sono progettati per colpire le proteine transmembrana e alterare la loro funzione.
"I nostri risultati aprono la strada alla progettazione di proteine di membrana multispan che potrebbero imitare le proteine presenti in natura o avere una struttura completamente nuova, funzione e usi, "ha detto David Baker, un professore di biochimica della School of Medicine dell'Università di Washington e direttore dell'UW Institute of Protein Design che ha guidato il progetto. La ricerca è riportata nel numero del 1 marzo della rivista Scienza . Peilong Lu, un membro anziano del laboratorio Baker, è l'autore principale del documento.
Ma capire come vengono messe insieme le proteine transmembrana e come funzionano si è rivelato impegnativo. Poiché agiscono mentre sono incorporati all'interno della membrana cellulare, le proteine transmembrana si sono dimostrate più difficili da studiare rispetto alle proteine che operano nella soluzione acquosa che costituisce il citoplasma delle cellule o nel fluido extracellulare.
Nel nuovo studio, Lu e i suoi colleghi usavano un programma per computer, sviluppato nel laboratorio Baker e chiamato Rosetta, che può prevedere la struttura in cui si piegherà una proteina dopo che è stata sintetizzata. L'architettura di una proteina è cruciale perché la struttura di una proteina determina la sua funzione.
La forma di una proteina si forma da complesse interazioni tra gli amminoacidi che compongono la catena proteica e tra gli amminoacidi e l'ambiente circostante. In definitiva, la proteina assume la forma che meglio bilancia tutti questi fattori in modo che la proteina raggiunga lo stato energetico più basso possibile.
Il programma Rosetta utilizzato da Lu e dai suoi colleghi può prevedere la struttura di una proteina tenendo conto di queste interazioni e calcolando lo stato energetico complessivo più basso. Non è insolito per il programma creare decine di migliaia di strutture modello per una sequenza di amminoacidi e quindi identificare quelle con lo stato energetico più basso. È stato dimostrato che i modelli risultanti rappresentano accuratamente la struttura che la sequenza assumerà probabilmente in natura.
Determinare la struttura delle proteine transmembrana è difficile perché porzioni di proteine transmembrana devono passare attraverso l'interno della membrana, che è fatto di grassi oleosi chiamati lipidi.
Nei fluidi acquosi, residui di amminoacidi che hanno catene laterali polari - componenti che possono avere una carica in determinate condizioni fisiologiche o che partecipano al legame idrogeno - tendono a trovarsi sulla superficie della proteina dove possono interagire con l'acqua, che ha cariche laterali negative e positive alla sua molecola. Di conseguenza, i residui polari sulle proteine sono detti idrofili, o "amante dell'acqua".
residui non polari, d'altra parte, tendono a trovarsi imballati all'interno del nucleo proteico lontano dal fluido acquoso polare. Tali residui sono chiamati idrofobici o "timorosi dell'acqua". Di conseguenza, l'interazione tra i residui amanti dell'acqua e timorosi dell'acqua della proteina ei fluidi acquosi circostanti aiuta a guidare il ripiegamento delle proteine e stabilizza la struttura finale della proteina.
Nelle membrane, però, il ripiegamento delle proteine è più complicato perché l'interno lipidico della membrana non è polare, questo è, non ha separazione di cariche elettriche. Questo significa che per essere stabile la proteina deve essere apolare, residui timorosi d'acqua sulla sua superficie, e impacchetta il suo polare, residui di acqua all'interno. Quindi deve trovare un modo per stabilizzare la sua struttura creando legami tra i residui idrofili all'interno del suo nucleo.
La chiave per risolvere il problema, dice Lu, consisteva nell'applicare un metodo sviluppato dal laboratorio Baker per progettare le proteine in modo che il polo polare, i residui idrofili si adattano in modo tale che abbastanza formerebbero interazioni polari-polari che possono legare insieme la proteina dall'interno.
"Mettere insieme queste "reti di legami a idrogeno sepolte" è stato come mettere insieme un puzzle, " ha detto il panettiere.
Con questo approccio, Lu e i suoi colleghi sono stati in grado di produrre le proteine transmembrana progettate all'interno di batteri e cellule di mammifero utilizzando fino a 215 amminoacidi. Le proteine risultanti si sono rivelate altamente stabili termicamente e in grado di orientarsi correttamente sulla membrana. Come le proteine transmembrana naturali, le proteine sono multipass, nel senso che attraversano la membrana più volte, e assemblare in complessi multiproteici stabili, come dimeri, trimeri e tetrameri.
"Abbiamo dimostrato che ora è possibile progettare con precisione complessi, proteine transmembrana multipasso che possono essere espresse nelle cellule. Ciò consentirà ai ricercatori di progettare proteine transmembrana con strutture e funzioni completamente nuove, " disse Lu.