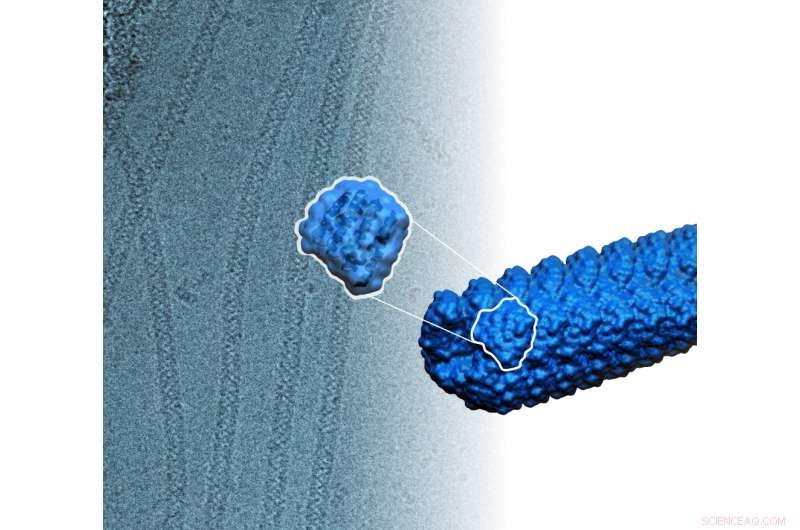
Nuovo, proteine progettate al computer si autoassemblano in filamenti più di mille volte più sottili di un capello umano. Credito:Ian C. Haydon/Institute for Protein Design
Per la prima volta, scienziati hanno creato, da zero, filamenti proteici autoassemblanti.
Questi sono stati costruiti da subunità proteiche identiche che si agganciano spontaneamente per formare lunghi, elicoidale, strutture filiformi.
Nel mondo naturale, i filamenti proteici sono componenti essenziali di diverse parti strutturali e mobili nelle cellule viventi, così come molti tessuti del corpo.
Questi includono i citoscheletri che danno alle cellule la loro forma, i microtubuli cellulari che orchestrano la divisione cellulare, e la proteina più comune nel nostro corpo, collagene, che dà forza e flessibilità alla nostra cartilagine, pelle e altri tessuti.
"Essere in grado di creare filamenti proteici da zero, o de novo, ci aiuterà a comprendere meglio la struttura e la meccanica dei filamenti proteici naturali e ci consentirà anche di creare materiali completamente nuovi, diversi da quelli che si trovano in natura, "ha detto David Baker, un professore di biochimica presso la University of Washington School of Medicine, e direttore dell'UW Institute for Protein Design, che ha diretto il progetto. È anche un investigatore dell'Howard Hughes Medical Institute.
Questi componenti proteici di nuova concezione si incastrano spontaneamente in filamenti. Le proteine progettate sono relativamente piccole, composto da circa 180-200 amminoacidi e misura solo su un nanometro di lunghezza, ma assemblare in filamenti stabili più di 10, 000 nanometri di lunghezza. I filamenti possono anche essere armeggiato per farli crescere o smontare. Credito:Institute for Protein Design/UW Medicine
Tali materiali potrebbero includere fibre artificiali che eguagliano o superano la forza della seta di ragno, che in peso è più forte dell'acciaio, ha detto il panettiere. Ha anche menzionato la possibilità di circuiti a filo su scala nanometrica.
Per progettare i filamenti, i ricercatori hanno utilizzato un programma per computer sviluppato nel laboratorio Baker, chiamata Rosetta, che può predire la forma di una proteina dalla sua sequenza di amminoacidi.
Per funzionare correttamente, le proteine devono assumere una forma precisa. Questo ripiegamento è guidato dalle proprietà dei singoli amminoacidi e dal modo in cui interagiscono tra loro e con l'ambiente fluido circostante. Le forze di attrazione e repulsione spingono la proteina a fermarsi in una forma con il livello energetico più basso.
Calcolando quale forma dovrebbe bilanciare queste forze di attrazione e repulsione per produrre il livello di energia totale più basso, Rosetta può prevedere, con un alto grado di precisione, la forma che assumerà una proteina in natura.
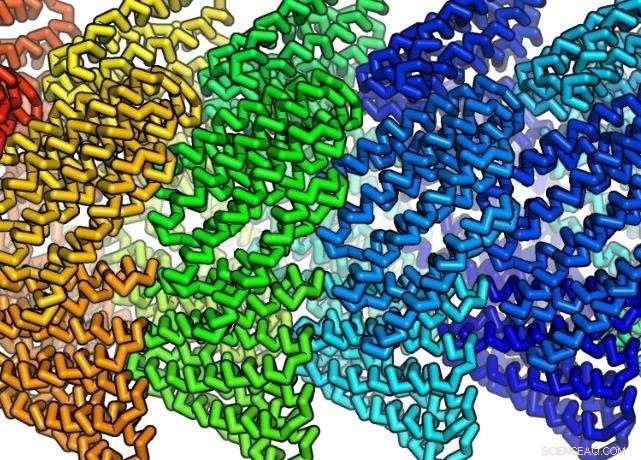
Usando Rosetta, i ricercatori hanno deciso di progettare piccole proteine che avevano amminoacidi sulla loro superficie che le avrebbero indotte ad attaccarsi l'una all'altra. Ciò ha permesso loro di assemblarsi in un'elica allineandosi come i gradini di una scala a chiocciola. Affinché l'elica sia stabile, la proteina progettata lega altre copie posizionate sopra e sotto di essa mentre l'elica si avvolge intorno, livello su livello.
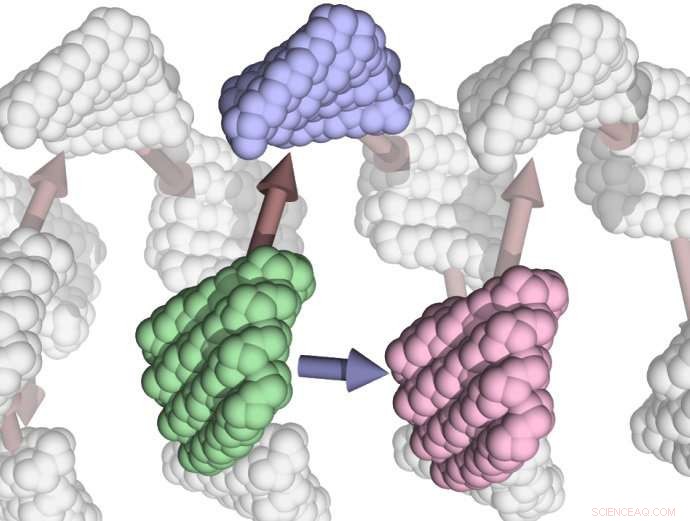
Concezione artistica di unità proteiche progettate al computer che iniziano ad autoassemblarsi per formare filamenti. Credito:Institute for Protein Design
"Alla fine siamo stati in grado di progettare proteine che si incastrassero insieme come i Lego, " disse Hao Shen, un dottorato di ricerca candidato presso l'UW Molecular Engineering &Sciences Institute. Lui e Jorge Fallas, un insegnante di recitazione in biochimica presso la UW School of Medicine, sono i principali autori di un documento che descrive l'approccio.
Questo articolo sarà pubblicato online dalla rivista Scienza di giovedì, 8 novembre 2018.
Fallas ha detto che le proteine progettate sono relativamente piccole. Sono costituiti da circa 180-200 amminoacidi e misurano solo circa un nanometro di lunghezza, ma assemblare in filamenti stabili più di 10, 000 nanometri di lunghezza. Un nanometro è 1 miliardesimo di metro, o circa la larghezza di 10 atomi di idrogeno allineati fianco a fianco.
I ricercatori hanno anche dimostrato che, armeggiando con la concentrazione della proteina progettata in soluzione e aggiungendo cappucci che inibiscono la capacità del progetto di legarsi, potrebbero far crescere o smontare i filamenti.
"La capacità di programmare la dinamica della formazione dei filamenti ci darà informazioni su come l'assemblaggio e lo smontaggio dei filamenti sono regolati in natura, " ha detto Baker. "La stabilità di queste proteine suggerisce che potrebbero fungere da scaffold facilmente modificabili per una gamma di applicazioni che vanno dai nuovi test diagnostici alla nanoelettronica".