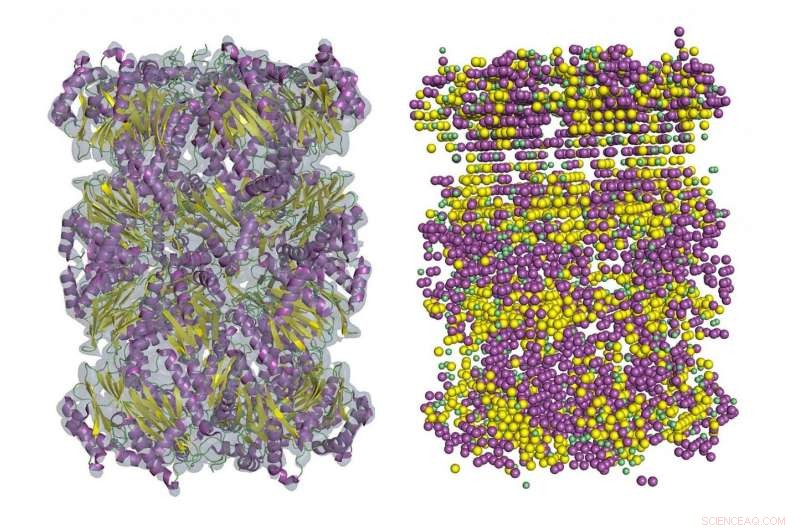
Un esempio del rilevamento della struttura secondaria nella mappa di densità cryo-EM utilizzando Emap2Sec. A sinistra è una mappa EM del proteasoma 20S archeale (ID EMDB:EMD-1733). A destra sono rilevate strutture secondarie da Emap2Sec. I punti in magenta sono le posizioni delle alfa eliche rilevate; i punti gialli vengono rilevati filamenti beta, e i punti verdi sono per le bobine rilevate (altre strutture). Credito:immagine della Purdue University/Daisuke Kihara
La microscopia crioelettronica è oggi il metodo più diffuso per determinare le strutture proteiche, che aiuta i ricercatori a sviluppare farmaci per diversi tipi di disturbi. Negli ultimi decenni, ha sostituito la cristallografia a raggi X perché può visualizzare proteine che non possono essere facilmente formate in grandi cristalli. La nuova tecnica è stata così rivoluzionaria da far vincere ai suoi sviluppatori il Premio Nobel 2017 per la chimica.
Il prodotto finale del crio-EM è una mappa della densità degli atomi nelle molecole biologiche, ma per raggiungere il livello di dettaglio di cui i ricercatori hanno bisogno, hanno bisogno di condurre ulteriori analisi. Un nuovo studio sulla rivista Metodi della natura delinea una tecnica per portare alla pari le mappe a bassa risoluzione.
L'approccio utilizzato dai ricercatori per farlo dipende dal livello di dettaglio con cui iniziano. Mappe da 2 a 3 ångström (Å, un'unità di lunghezza usata per esprimere la dimensione di atomi e molecole) sono generalmente considerati ad alta risoluzione. Però, mappe di questa qualità sono difficili da realizzare, e molti sono ancora comunemente prodotti nell'intervallo da 4 a 10 . Di tutte le proteine depositate nella Banca Dati di Microscopia Elettronica dal 2016-18, più del 50% è stato risolto a risoluzione intermedia.
"Se la risoluzione è migliore di tre, quindi gli strumenti convenzionali possono tracciare la posizione degli amminoacidi e costruire una mappa delle posizioni degli atomi. Ma spesso il cryo-EM non può darti una mappa a 3 , " disse Daisuke Kihara, professore di scienze biologiche e informatica alla Purdue University. "Nelle mappe di 5 o inferiori, di solito non puoi vedere affatto la connettività della catena."
Le proteine sono in realtà catene di amminoacidi, e il legame tra gruppi amminici e gruppi carbossilici a volte crea determinati modelli di piegatura. Questi modelli, note come alfa eliche e filamenti beta, formano la struttura secondaria della proteina.
Nelle mappe da 5 a 8 Å, sono generalmente visibili alcuni frammenti della struttura secondaria delle proteine, ma tracciare l'intera catena sarebbe molto difficile. Il nuovo metodo di Kihara, noto come Emap2sec, scopre strutture secondarie nelle mappe da 6 a 10 .
Emap2sec ha una rete neurale convoluzionale profonda al centro del suo algoritmo. Queste reti sono sistemi di deep learning utilizzati principalmente per classificare immagini, raggrupparli per somiglianza ed eseguire il riconoscimento degli oggetti. Funziona per l'identificazione della struttura proteica nelle mappe 3D perché il metodo "convolge" le caratteristiche della densità della mappa locale in immagini di una regione più ampia mentre le informazioni passano attraverso gli strati della rete neurale. La previsione locale viene effettuata nel contesto di un'ampia regione della mappa.
Le strutture secondarie identificate nelle mappe 3D aiutano i ricercatori ad assegnare strutture note di proteine che sono già state risolte nella mappa. Ciò significa che a volte hanno un punto di partenza, o almeno un indizio di come siano alcune delle strutture. Emap2sec può aiutare i ricercatori a inserire il loro pezzo nel puzzle in modo più rapido e semplice. Le informazioni sulla struttura identificate potrebbero anche essere utili per trovare errori nella modellazione della struttura.