I risultati pubblicati dai ricercatori dell'AU rivelano che lo spiegamento e il ripiegamento delle proteine mediati da tensioattivi sono processi complessi con diverse strutture presenti, e i riarrangiamenti avvengono su scale temporali da sub-millisecondi a minuti. (Immagine:riprodotta con il permesso della Royal Society of Chemistry). Credito: scienze chimiche , copyright 2020 Royal Society of Chemistry.
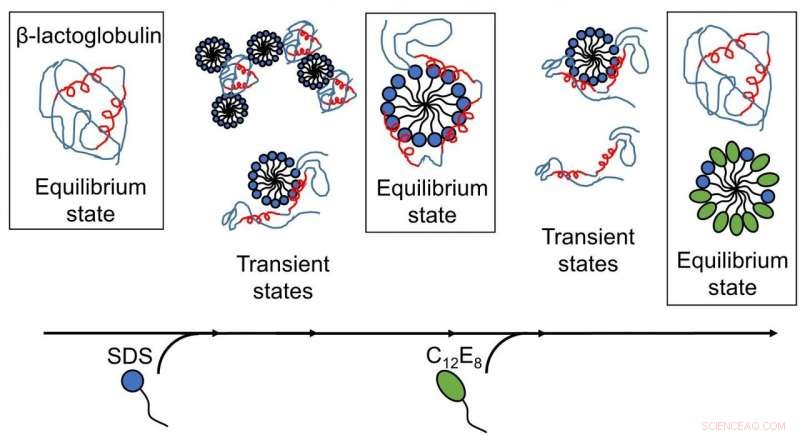
Comprendere le interazioni tra proteine e molecole di sapone (tensioattivi) è stato a lungo importante per l'industria, in particolare all'interno di detersivi e cosmetici. Il tensioattivo anionico sodio dodecilsolfato (SDS) è noto per dispiegare proteine globulari, mentre il tensioattivo non ionico ottaetilenglicole monododecil etere (C12E8) fa l'opposto, cioè aiuta le proteine a piegarsi di nuovo in forma.
Affinché i detersivi in polvere funzionino in modo efficiente, è importante che i tensioattivi non modifichino la struttura delle proteine (enzimi), poiché qualsiasi cambiamento nella struttura degli enzimi uccide la loro capacità di abbattere le macchie e rimuovere lo sporco. La maggior parte dei detersivi contiene miscele di tensioattivi che consentono agli enzimi di rimanere attivi. Anche, alcune biotecnologie sfruttano i tensioattivi in combinazione con le proteine.
Le proteine di membrana di solito si trovano nella membrana cellulare. Per estrarli da questo ambiente per diversi studi, devono essere solubilizzati dal tensioattivo. Il tensioattivo deve essere "delicato" e coprire solo la parte della proteina inserita nella membrana in modo che la loro struttura sia preservata. In contrasto, quando si caratterizza il peso molecolare delle proteine in laboratorio, una tecnica standard consiste nel dispiegarli mediante il tensioattivo aggressivo caricato negativamente, SDS, e monitorare come migrano in un gel polimerico in un campo elettrico. Questa tecnica funziona solo se il tensioattivo dispiega completamente le proteine e ne distrugge la struttura.
C'è ancora dibattito su quale tipo di interazione tra la proteina e il tensioattivo sia più importante. Sono le interazioni elettrostatiche tra le cariche del tensioattivo e la proteina, o sono semplicemente le proprietà dell'interfaccia degli aggregati (micelle) che i tensioattivi formano in acqua, quali sono responsabili dello sviluppo della proteina?
Mentre l'unfolding è stato studiato in dettaglio a livello proteico, in questi processi manca un quadro completo dell'interazione tra proteina e tensioattivo. Questa mancanza di conoscenza viene affrontata nel lavoro attuale utilizzando la proteina globulare -lattoglobulina (bLG) come proteina modello.
La giusta combinazione di tecniche sperimentali
È stata ottenuta una visione più approfondita dello spiegamento e del ripiegamento delle proteine, poiché le varie fasi delle interazioni tra tensioattivo e proteine sono state mappate in funzione del tempo. in primo luogo, la proteina modello, bLG, è stato miscelato con il tensioattivo anionico SDS mentre seguiva l'evoluzione temporale della formazione di complessi tra proteine e molecole di tensioattivo sulla scala temporale del millisecondo-minuto. Con questo i ricercatori hanno determinato la struttura dei complessi in evoluzione. Successivamente hanno mappato l'andamento temporale del processo di ripiegamento quando il tensioattivo non caricato (C12E8) è stato aggiunto a un campione contenente complessi di SDS e proteine.
Per osservare come la proteina si riorganizza durante il processo di spiegamento e ripiegamento indotto dai tensioattivi, tecniche spettroscopiche complementari, dicroismo circolare e fluorescenza del triptofano, sono stati utilizzati in combinazione con lo scattering di raggi X a piccolo angolo risolta nel tempo (SAXS).
Il dicroismo circolare e la fluorescenza del triptofano monitorano i cambiamenti nella struttura di bLG, mentre i cambiamenti nella forma complessiva dei complessi proteina-tensioattivo sono stati seguiti dal sincrotrone SAXS. Questa combinazione di tecniche non è stata utilizzata prima per studiare questi processi.
Processi complessi che durano da millisecondi a minuti
Il dispiegamento della proteina mediante SDS è stato un processo omogeneo, dove tutte le molecole proteiche seguono lo stesso percorso di dispiegamento. I complessi SDS (micelle) attaccano frontalmente le molecole proteiche e poi dispiegano gradualmente la proteina in modo che formi un guscio attorno alla micella SDS. Il ripiegamento inizia quando le micelle C12E8 "risucchiano" SDS dal complesso proteina-SDS per formare micelle SDS-C12E8 miste. Però, the actual refolding process seems to follow several routes, since multiple structures were found to form in parallel, namely protein-surfactant complexes (probably containing both SDS and C12E8), mixed micelles of SDS and C12E8, "naked" proteins unfolded like long polymeric chains, and properly folded proteins. The experiment allowed the interconversion between these species to be followed, so that it could be determined which of the processes are fast and which ones are slow. The folded protein could form both from the naked unfolded proteins (quickly) and from protein-surfactant complexes (more slowly). Così, the best way in which surfactants can help a protein to fold is to basically get out of the way and let the protein find its own way back to the folded state.
The results have provided deeper insight into the structural changes occurring at the protein-surfactant level. They revealed that surfactant-mediated unfolding and refolding of proteins are complex processes of rearrangements occurring on time scales from below milliseconds to minutes and involve intimate collaboration between surfactant complexes and proteins.