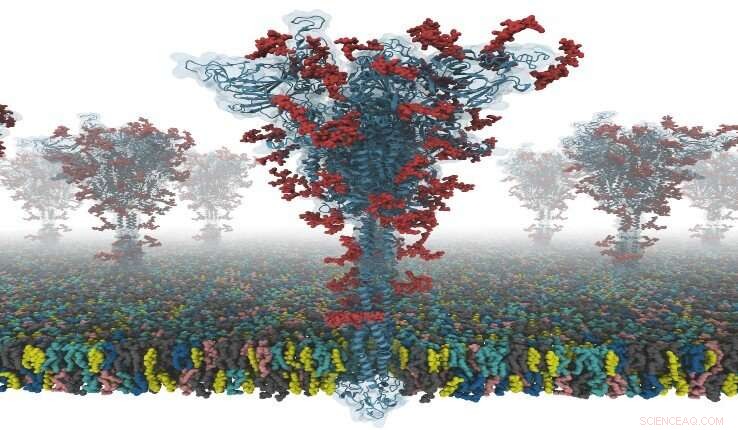
Un modello di una proteina S. Credito:Dr. Yeolkyo Choi/Lehigh
Il virus SARS coronavirus 2 (SARS-CoV-2) è la causa nota della malattia da coronavirus 2019 (COVID-19). La proteina "spike" o S facilita l'ingresso del virus nelle cellule ospiti.
Ora un gruppo di ricercatori della Seoul National University in Corea del Sud, Università di Cambridge nel Regno Unito, e Lehigh University negli Stati Uniti, hanno lavorato insieme per produrre i primi modelli di tutti gli atomi open source di una proteina S a lunghezza intera. I ricercatori dicono che questo è di particolare importanza perché la proteina S svolge un ruolo centrale nell'ingresso virale nelle cellule, rendendolo un obiettivo principale per lo sviluppo di vaccini e farmaci antivirali.
I dettagli possono essere trovati in un documento, "Sviluppo di un modello di proteina Spike SARS-CoV-2 completamente glicosilato in una membrana virale" appena pubblicato online in Il giornale di chimica fisica B .
Questa demo video illustra come costruire questo sistema di membrane dai loro modelli di proteine SARS-CoV-2 S. Il programma di costruzione del modello è ad accesso aperto e può essere trovato dalla home page di CHARMM-GUI facendo clic sul collegamento Archivio COVID-19, o facendo clic sul collegamento all'archivio nell'intestazione, quindi il link Proteine COVID-19 nella barra laterale sinistra.
Sviluppato da Wonpil Im, un professore nel Dipartimento di Scienze Biologiche e Bioingegneria della Lehigh University, CHARMM-GUI (GUI =interfaccia utente grafica) è un programma che simula in modo semplice sistemi biomolecolari complessi, preciso e rapido. Im lo descrive come un "microscopio computazionale" che consente agli scienziati di comprendere le interazioni a livello molecolare che non possono essere osservate in nessun altro modo. Maggiori informazioni su CHARMM-GUI possono essere trovate in questo video.
"I nostri modelli sono i primi modelli di proteina spike (S) SARS-CoV-2 a lunghezza intera completamente glicosilati che sono disponibili per altri scienziati, " dice Im. "Ho avuto la fortuna di collaborare con il Dr. Chaok Seok della Seoul National University in Corea e il Dr. Tristan Croll dell'Università di Cambridge nel Regno Unito. Il nostro team ha trascorso giorni e notti per costruire questi modelli con molta attenzione dal noto crio- Porzioni di struttura EM. La modellazione è stata molto impegnativa perché c'erano molte regioni in cui la modellazione semplice non è riuscita a fornire modelli di alta qualità".
Gli scienziati possono utilizzare i modelli per condurre ricerche di simulazione innovative e innovative per la prevenzione e il trattamento di COVID-19, secondo Im.
La struttura della proteina S è stata determinata con cryo-EM con RBD up (PDB ID:6VSB), e con RBD abbassato (ID PDB:6VXX). Ma, questo modello ha molti residui mancanti. Così, hanno prima modellato i residui di amminoacidi mancanti, e poi altri domini mancanti. Inoltre, hanno modellato tutti i potenziali glicani (o carboidrati) attaccati alla proteina S. Questi glicani impediscono il riconoscimento degli anticorpi, che rende difficile sviluppare un vaccino. Hanno anche costruito un sistema di membrana virale di una proteina S per la simulazione della dinamica molecolare.