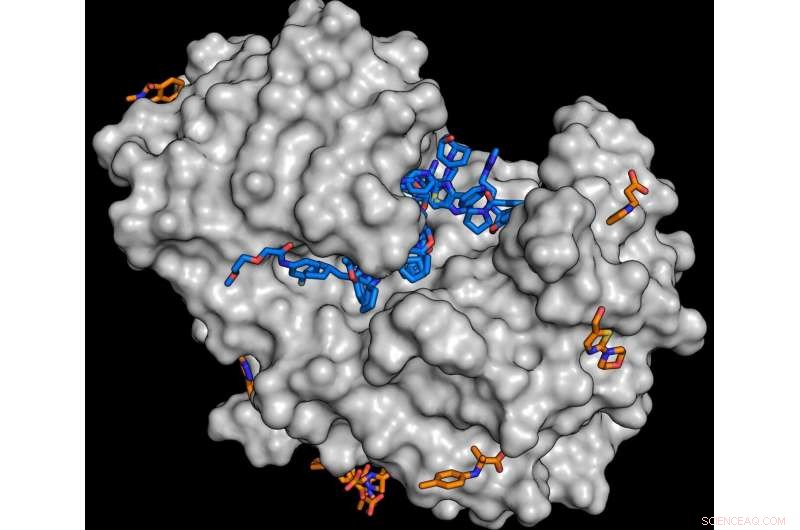
Per lo studio, l'enzima endotiapepsina (grigio) è stato combinato con molecole della libreria di frammenti. L'analisi mostra che numerose sostanze sono in grado di agganciarsi all'enzima (molecole blu e arancioni). Ogni sostanza trovata è un potenziale punto di partenza per lo sviluppo di molecole più grandi. Credito:J. Wollenhaupt/HZB
Al fine di accelerare lo sviluppo sistematico dei farmaci, il team MX dell'Helmholtz-Zentrum Berlin (HZB) e il Drug Design Group dell'Università di Marburgo hanno creato una nuova libreria di sostanze. Consiste di 1103 molecole organiche che potrebbero essere utilizzate come elementi costitutivi per nuovi farmaci. Il team MX ha ora convalidato questa libreria in collaborazione con il gruppo FragMAX al MAX IV. La libreria di sostanze dell'HZB è disponibile per la ricerca in tutto il mondo e svolge anche un ruolo nella ricerca di sostanze attive contro SARS-CoV-19.
Perché i farmaci siano efficaci, di solito devono ancorarsi alle proteine di un organismo. Come una chiave in una serratura, parte della molecola del farmaco deve inserirsi in recessi o cavità della proteina bersaglio. Ormai da diversi anni, il team del Dipartimento di Cristallografia Macromolecolare (MX) dell'HZB guidato dal Dr. Manfred Weiss insieme al Drug Design Group guidato dal Prof. Gerhard Klebe (Università di Marburg) ha quindi lavorato alla costruzione delle cosiddette librerie di frammenti. Questi sono costituiti da piccole molecole organiche (frammenti) con cui è possibile sondare e mappare le cavità funzionalmente importanti sulla superficie delle proteine. I cristalli proteici vengono saturati con i frammenti e quindi analizzati utilizzando una potente luce a raggi X. Ciò consente di ottenere informazioni strutturali tridimensionali a livelli di risoluzione atomica. Tra l'altro, è possibile scoprire quanto bene uno specifico frammento di molecola si aggancia alla proteina bersaglio. Lo sviluppo di queste librerie di sostanze è avvenuto nell'ambito del progetto di ricerca congiunto Frag4Lead ed è stato finanziato dal Ministero federale tedesco dell'istruzione e della ricerca (BMBF).
Il team MX (MX sta per Macromolecular Crystallography) ha ora pubblicato il progetto di una libreria di frammenti chimicamente diversificata chiamata libreria "F2X-Universal", che si compone di 1, 103 composti. Da questa libreria è stata estratta una selezione rappresentativa di 96 composti, che è indicato come lo schermo di immissione F2X. Nel corso della pubblicazione della biblioteca, questa selezione è stata ora testata e convalidata con successo dal team MX dell'HZB presso la sorgente di raggi X MAX IV a Lund, Svezia e a BESSY II.
Nello studio, i team HZB e MAX IV hanno verificato l'efficienza della libreria F2X Entry esaminando l'endotiapepsina e il complesso proteico Aar2/RnaseH come enzimi bersaglio. Nel passaggio successivo, il team MX utilizzerà l'intera libreria universale.
"Per lo studio in corso, gli esperti di screening dei frammenti presso HZB—BESSY II hanno lavorato a stretto contatto con il team del progetto FragMAX presso MAX IV", ha affermato il Dr. Uwe Müller del team MX di HZB che ha contribuito a impostare le tre linee di luce MX a BESSY II e la linea di luce BioMAX a MAX IV. "Ciò ha consentito a entrambi i partner di sviluppare ulteriormente le proprie piattaforme tecnologiche e di utilizzarle per l'imaging delle superfici funzionali di diverse proteine. Questa sarà un'ottima base per la futura collaborazione tra MAX IV e HZB".