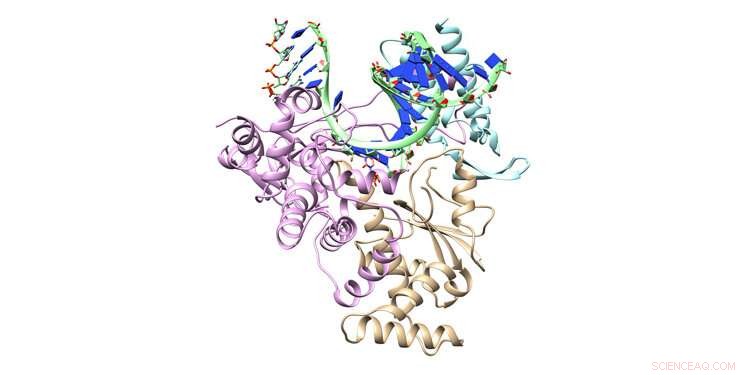
Il farmaco antivirale remdesivir costituisce la linea principale di difesa terapeutica approvata dalla FDA contro il virus COVID-19. I ricercatori dell'Università del Nord del Texas stanno utilizzando il supercomputer Frontera per modellare il modo in cui remdesivir blocca la riproduzione del coronavirus, nella speranza di sviluppare miglioramenti sul farmaco. Qui sono mostrate le strutture cristalline del modello complesso ternario RNA-polimerasi RNA-dipendente con RNA a doppio filamento e remdesivir trifosfato in ingresso. Credito:Gruppo di ricerca Cisneros, UNT
Le proteine chiave utilizzate dal coronavirus per la sua riproduzione sono state modellate sul supercomputer Frontera finanziato dall'NSF dal gruppo di ricerca Andres Cisneros dell'Università del North Texas. Gli obiettivi della ricerca includono la ricerca di modi per migliorare il remdesivir terapeutico COVID-19. Assegnazione Frontera finanziata dalla NSF assegnata a Cisneros attraverso il COVID-19 High Performance Computing Consortium.
A maggio 2020, la Food and Drug Administration degli Stati Uniti ha autorizzato il farmaco antivirale remdesivir per il trattamento di emergenza di COVID-19, una delle sole quattro terapie attualmente con questo status. Remdesivir ferma il macchinario chimico che il coronavirus usa per copiarsi, legandosi ad un enzima che fa l'assemblaggio. Mentre remdesivir ha mostrato risultati promettenti nell'aiutare i pazienti a riprendersi da COVID-19, gli scienziati stanno studiando modi per migliorarne l'efficacia.
Un team di scienziati guidato da G. Andres Cisneros dell'Università del North Texas sta modellando le parti chiave del coronavirus che utilizza per copiarsi. Le simulazioni vengono eseguite sui supercomputer Stampede2 e Frontera presso il Texas Advanced Computing Center (TACC).
"Siamo stati molto fortunati a ricevere un'assegnazione su Frontera per poter lavorare allo studio del meccanismo dei farmaci che prendono di mira due proteine specifiche nel COVID-19, " Ha detto Cisneros. Il suo lavoro indaga su come remdesivir e altri farmaci disponibili inibiscono le proteine NSP-12 e la principale proteasi, entrambi gli enzimi di cui il coronavirus ha bisogno per la replicazione. "Guardando come questi farmaci fanno il loro lavoro, forse queste informazioni possono essere utilizzate per migliorarle."
La proteina NSP-12 mette insieme i nucleotidi che compongono l'RNA virale, abbreviato in A, tu, G, e C, costruire set completi di materiale genetico per nuove copie di coronavirus. NSP-12 è in realtà parte di una struttura più ampia chiamata RNA polimerasi RNA-dipendente (RDRP) che copia l'RNA completo. Remdesivir si lega con RDRP, collegare i macchinari.
"Stiamo indagando su come avviene questo processo, " disse Cisneros. "Così facendo, forse potrebbe esserci un modo per noi e altri scienziati di trovare idee su se e come migliorare remdesivir".
L'altra proteina che Cisneros sta studiando è chiamata proteasi principale. Separa una poliproteina prodotta da SARS-CoV-2 tradotta dall'RNA virale in proteine funzionali che mettono "carne" sulle sue ossa virali. Ferma la proteasi, e impedisci al virus di formarsi. Questo lo rende un ottimo bersaglio farmacologico.
Cisneros ha spiegato che usa la matematica e la fisica di base delle equazioni di Newton e della meccanica quantistica per calcolare le proprietà delle proteine, compreso tutto ciò che è rilevante per il suo funzionamento, come l'RNA e l'acqua. Un approccio chiamato dinamica molecolare classica utilizza le equazioni di Newton per simulare come le proteine si muovono e interagiscono dinamicamente nel tempo. "Stiamo parlando di sistemi che simuliamo di centinaia di migliaia di atomi, " Disse Cisneros.
Simula anche le reazioni chimiche all'interno delle proteine per studiare come i farmaci bloccano l'RDRP o la proteasi. Un metodo ibrido chiamato QM/MM (meccanica quantistica/meccanica molecolare) consente di risparmiare tempo e denaro di calcolo concentrandosi maggiormente sulle interazioni nel sito attivo, usando le dinamiche molecolari rettilinee più approssimative per tutto il resto.
Il gruppo Cisneros ha sviluppato e mantiene un programma chiamato LICHEM che consente loro di utilizzare l'approccio QM/MM. "Una delle caratteristiche di LICHEM è che ci consente di utilizzare approcci per la parte di meccanica classica che includono una migliore descrizione della fisica che sta accadendo tra le molecole nell'ambiente classico, nello specifico, il potenziale dell'AMOEBA", ha affermato Cisneros. L'AMOEBA è sviluppato da Pengyu Ren di UT Austin, Jay Ponder dell'Università di Washington e Jean-Philip Piquemal dell'Università Sorbonne di Parigi con il contributo del gruppo Cisneros per i liquidi ionici.
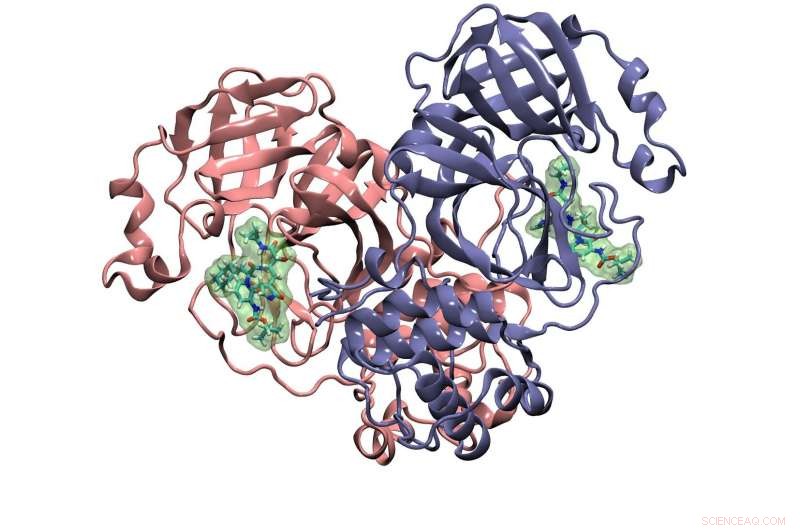
Un altro obiettivo di ricerca del gruppo Cisneros modellato su Frontera è una proteina chiamata proteasi principale. Scinde una poliproteina prodotta dal virus che costruisce le proteine funzionali delle copie di se stesso che genera. Struttura cristallina della proteasi principale del coronavirus con inibitore legato mostrato qui. Credito:Gruppo di ricerca Cisneros, UNT
"Frontera, con non solo la potenza di calcolo ma l'intercomunicazione tra i nodi, ci consente di eseguire questi calcoli QM/MM con valori molto più alti, non solo velocità, ma anche rendimento, " Ha detto Cisneros. Frontera li ha liberati per eseguire più sistemi alla volta. "Nel mio gruppo, Ho cinque scienziati diversi, dottorandi e dottori di ricerca, che funzionano su entrambi questi sistemi, ma in diversi pezzi del puzzle. Tutti possono accedere a queste risorse. è sicuramente molto utile, e apprezziamo molto l'assegnazione."
Ciò che ha fatto andare avanti Cisneros è stata la notizia nell'aprile del 2020 della segnalazione della struttura cristallina del SARS-CoV-2 RDRP. "Ho contattato il mio gruppo e ho detto loro che con queste informazioni, c'è qualcosa che possiamo fare per aiutare con la pandemia, " Egli ha detto.
Entro due giorni dalla notizia, Cisneros ha proposto con successo la sua ricerca sui bersagli farmacologici del coronavirus al COVID-19 High Performance Computing Consortium. Decine di strutture di supercalcolo nazionali e internazionali, industria, e organizzazioni tra cui TACC hanno offerto volontariamente le proprie risorse al consorzio a sostegno degli sforzi degli scienziati per combattere il coronavirus.
L'assegnazione è stata inizialmente assegnata solo su Stampede2 di TACC. il fiore all'occhiello del supercalcolo della National Science Foundation (NSF) che è classificato 21st più veloce al mondo e #2 per i sistemi accademici secondo la Top500. "Poi siamo stati contattati da TACC e siamo stati grati che ci fosse stato concesso l'accesso a Frontera. Ora abbiamo accesso a entrambi i sistemi, che è davvero fantastico, " Disse Cisneros.
Il supercomputer Frontera è il supercomputer accademico numero 1 più veloce e l'8 più veloce al mondo. Sia Frontera che Stampede2 sono finanziati dalla NSF.
"Siamo molto soddisfatti di questo sistema. Siamo stati in grado di trasferire parte delle conoscenze che avevamo da Stampede2 a Frontera, " Disse Cisneros. Uno dei suoi studenti neolaureati, Erik Vazquez Montelongo, impostare tutti i calcoli per LICHEM su Frontera in base a quanto appreso su Stampede2. "È stato davvero un vantaggio. Frontera per i nostri calcoli sta funzionando davvero bene. Siamo davvero contenti".
Uno dei postdoc in The Cisneros Group, Sehr Nazeem-Kahn, generato il modello per RDRP, il remdesivir e altri farmaci candidati, tutto nel sito attivo. Con quello in mano, hanno iniziato a eseguire simulazioni.
"Siamo stati molto felici di vedere che il suo modello era in realtà molto vicino alla struttura sperimentale. Questo è davvero utile per noi, perché convalida il modello che è stato costruito dal gruppo e mostra che siamo sulla strada giusta, " Ha aggiunto.
Attualmente, Il Dr. Naseem-Khan sta eseguendo simulazioni di dinamica molecolare di questo modello con remdesivir su Frontera. "Stiamo anche iniziando con i nostri calcoli QM/MM per RDRP. Nel caso della proteasi principale, c'erano anche strutture che dovevano essere modellate e successivamente confermate. Anche questo è stato molto soddisfacente, " Disse Cisneros.
Con quei dati di struttura, stanno esaminando sei diverse molecole di inibitore. "Uno di quelli, stiamo già iniziando i calcoli QM/MM su Frontera, e un altro su Stampede2, " Disse Cisneros. Se tutto va bene, spera di ottenere risultati nei prossimi cinque o sei mesi. "Sono calcoli molto costosi, " ha aggiunto. "Inoltre, l'esecuzione dell'analisi richiede tempo. Se dovessimo usare solo le risorse a casa, ci vorrebbero diversi anni».