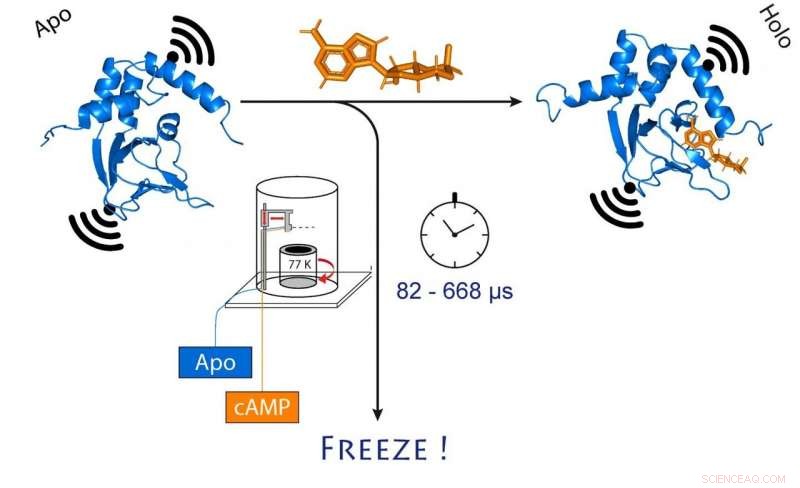
quando cAMP (arancione) si lega. Questo cambia anche la distanza tra i due magneti molecolari attaccati al canale (nero). Credito:(c) Tobias Hett / Universität Bonn und Forschungszentrum caesar
I ricercatori dell'Università di Bonn e del Centro di ricerca caesar sono riusciti a congelare le proteine ultravelocemente dopo un periodo di tempo ben definito. Sono stati in grado di seguire i cambiamenti strutturali su una scala temporale di microsecondi e con una precisione sub-nanometrica. Grazie alla sua elevata risoluzione spaziale e temporale, il metodo consente di tracciare rapidi cambiamenti strutturali negli enzimi e negli acidi nucleici. I risultati sono pubblicati nel Giornale dell'American Chemical Society.
Se vuoi sapere come appare la struttura spaziale di una biomolecola, hai un formidabile arsenale di strumenti a tua disposizione. I più popolari sono la microscopia elettronica e la diffrazione dei raggi X, che può rivelare anche i più piccoli dettagli di una proteina. Però, una limitazione significativa di questi metodi è che di solito forniscono immagini statiche, che spesso sono insufficienti per comprendere i processi biomolecolari in precisi termini meccanicistici. Perciò, un obiettivo a lungo termine di molti gruppi di ricerca in tutto il mondo è stato quello di monitorare i movimenti all'interno di una macromolecola come una proteina nel tempo mentre svolge il suo lavoro, proprio come in un film. I gruppi di ricerca guidati dal Prof. Dr. Olav Schiemann dell'Istituto di Chimica Fisica e Teorica dell'Università di Bonn e dal Prof. Dr. Benjamin Kaupp del Centro di Ricerca caesar della Max Planck Society si sono ora avvicinati a questo obiettivo obiettivo.
Hanno scelto un canale ionico per la loro indagine. Questa è una proteina che forma minuscoli pori nella membrana cellulare che sono permeabili alle particelle cariche chiamate ioni. "Questo canale è normalmente chiuso, "Spiega Schiemann. "Si apre solo quando un messaggero cellulare, chiamato cAMP, si lega ad esso. Volevamo sapere come funziona esattamente questo processo".
Mini magneti per misurare le distanze
Fare così, i ricercatori hanno prima mescolato la proteina del canale e il cAMP e poi hanno rapidamente congelato la soluzione. Nello stato congelato, la struttura della proteina può ora essere analizzata. Perché il loro metodo funzioni, avevano attaccato elettromagneti molecolari in due punti del canale. La distanza tra questi magneti può essere determinata con una precisione di pochi Angstrom (dieci miliardesimi di millimetro) utilizzando un sofisticato metodo chiamato PELDOR, che funziona come un righello molecolare. Negli ultimi anni, il metodo è stato significativamente raffinato e migliorato nel gruppo di Schiemann.
"Però, questo ci dà solo un'immagine statica del legame del cAMP al canale ionico, "Spiega Schiemann. "Abbiamo quindi ripetuto il processo di congelamento in tempi diversi dopo aver mescolato le due molecole. Ciò ha permesso di ricostruire i movimenti nella proteina dopo il legame con cAMP, proprio come un film, che si compone anche di una sequenza di immagini."
Al centro di questa procedura c'è un metodo sofisticato che consente di miscelare e congelare i campioni molto rapidamente in un momento preciso. La tecnica, chiamato "microsecondo congelamento hyperquenching" (abbreviato MHQ), è stato originariamente sviluppato presso l'Università di Delft, ma poi cadde in disuso. È stato riscoperto e decisamente affinato dal gruppo di Kaupp.
"Nel dispositivo MHQ, la molecola di cAMP e il canale ionico vengono miscelati a velocità ultraveloce, " spiega Kaupp. "Quindi la miscela viene sparata come un flusso sottilissimo su un cilindro di metallo molto freddo a -190 ° C, che ruota 7, 000 volte al minuto. È stato particolarmente difficile trasferire i campioni congelati per la misurazione PELDOR dalla piastra metallica in tubi di vetro sottili, e di tenerli congelati nel frattempo. Abbiamo dovuto progettare e costruire strumenti speciali per questo."
Surgelazione in 82 milionesimi di secondo
L'intero processo di miscelazione e congelamento richiede solo 82 microsecondi (un microsecondo equivale a un milionesimo di secondo). "Questo ci permette di visualizzare cambiamenti molto rapidi nella struttura spaziale delle proteine, " spiega Tobias Hett, uno dei due dottorandi che hanno contribuito in modo significativo al successo. Il vantaggio del metodo è la sua combinazione di alta risoluzione spaziale e temporale. "Questo rappresenta un importante passo avanti nello studio dei processi dinamici nelle biomolecole, " sottolinea Kaupp.
I ricercatori ora intendono utilizzare il loro metodo per dare un'occhiata più da vicino ad altre biomolecole. Sperano di acquisire nuove intuizioni, per esempio nel funzionamento di enzimi e acidi nucleici. L'importanza di tali intuizioni è illustrata al meglio dalla recente ondata mondiale di ricerche strutturali sulla SARS coronavirus-2:anche la cosiddetta proteina spike del virus subisce un cambiamento strutturale quando le cellule umane vengono infettate. Chiarire questo meccanismo fornirà preziose informazioni su come indirizzare il meccanismo di infezione con nuovi farmaci.
La preparazione dei campioni, l'esecuzione sperimentale, e l'analisi dei dati è molto complessa. I risultati dello studio riflettono quindi anche una collaborazione scientifica di successo con i ricercatori guidati dal Prof. Dr. Helmut Grubmüller dell'Istituto Max Planck di chimica biofisica di Göttingen e dal Prof. Dr. Heinz-Jürgen Steinhoff dell'Università di Osnabrück.