La retrosintesi mira a prevedere una serie di reagenti per la produzione di determinate molecole, che svolgono un ruolo significativo nel campo della biochimica, come la progettazione del percorso molecolare e la scoperta di farmaci. La maggior parte dei metodi esistenti beneficiano solo di un tipo di informazione anziché considerare ulteriormente i diversi aspetti dell'informazione molecolare.
Per affrontare questo problema, un gruppo di ricerca ha condotto uno studio, ora pubblicato su Frontiers of Computer Science .
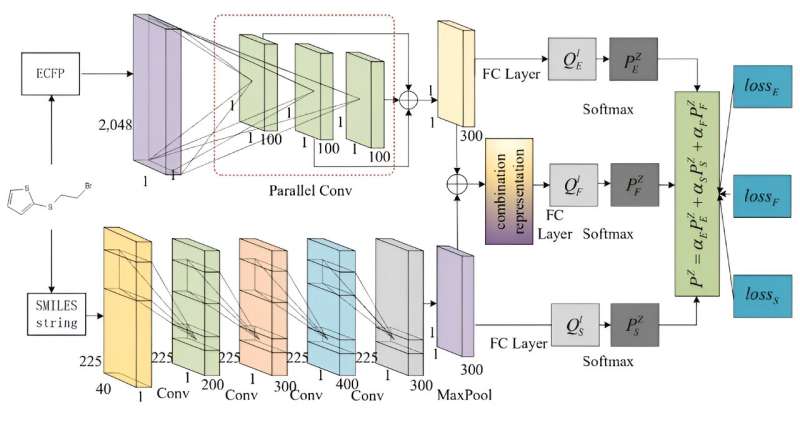
Il team ha proposto una rete multi-flusso per la previsione della retrosintesi descrivendo le molecole da più prospettive utilizzando i loro descrittori SMILES ed ECFP.
MSNR è costituito da tre moduli principali:(i) La CNN parallela e la CNN testuale che accetta gli ECFP e gli SMILES dopo la codifica one-hot come input per produrre le funzionalità profonde. (ii) La rappresentazione combinata è ideata fondendo i due tipi di caratteristiche profonde di ECFP e SMILES, che forniscono una prospettiva approfondita della rappresentazione molecolare. (iii) Sono stati implementati tre classificatori densi per prevedere la probabilità della presenza di reagenti per le molecole, che sfruttano le caratteristiche profonde estratte da diversi flussi rispettivamente come rappresentazione molecolare.
Unendo questi risultati di previsione multi-flusso con pesi variabili, il modello arriva a una previsione di retrosintesi finale. Inoltre, il modello viene addestrato utilizzando una funzione di perdita complessiva in grado di sfruttare le diverse informazioni disponibili da ciascun tipo di caratteristica profonda.
Ulteriori informazioni: Qiang Zhang et al, Una rete multi-flusso per la previsione della retrosintesi, Frontiere dell'informatica (2023). DOI:10.1007/s11704-023-3103-z
Fornito da Frontiers Journals