I ricercatori della Brown University hanno dimostrato di poter memorizzare una varietà di file di immagine:un disegno di Picasso, un'immagine del dio egizio Anubi e altri -- in matrici di miscele contenenti piccole molecole sintetizzate su misura. In tutto, i ricercatori hanno archiviato più di 200 kilobyte di dati, che dicono sia il più immagazzinato fino ad oggi usando piccole molecole. Credito:Brown University
Un team di ricercatori della Brown University ha compiuto progressi sostanziali nel tentativo di creare un nuovo tipo di sistema di archiviazione dei dati molecolari.
In uno studio pubblicato su Comunicazioni sulla natura , il team ha archiviato una serie di file di immagine:un disegno di Picasso, un'immagine del dio egizio Anubi e altri, in serie di miscele contenenti piccole molecole sintetizzate su misura. In tutto, i ricercatori hanno archiviato più di 200 kilobyte di dati, che dicono sia il più immagazzinato fino ad oggi usando piccole molecole. Non sono molti dati rispetto ai tradizionali mezzi di archiviazione, ma è un progresso significativo in termini di stoccaggio di piccole molecole, dicono i ricercatori.
"Penso che questo sia un sostanziale passo avanti, " disse Jacob Rosenstein, un assistente professore alla Brown's School of Engineering e autore dello studio. "Il gran numero di piccole molecole uniche, la quantità di dati che possiamo memorizzare, e l'affidabilità della lettura dei dati mostra una vera promessa per ampliarla ulteriormente."
Man mano che l'universo dei dati continua ad espandersi, molto lavoro è stato fatto per trovare nuovi e più compatti mezzi di stoccaggio. Codificando i dati nelle molecole, potrebbe essere possibile memorizzare l'equivalente di terabyte di dati in pochi millimetri di spazio. La maggior parte della ricerca sull'immagazzinamento molecolare si è concentrata su polimeri a catena lunga come il DNA, che sono noti portatori di dati biologici. Ma ci sono potenziali vantaggi nell'usare piccole molecole rispetto ai polimeri lunghi. Le piccole molecole sono potenzialmente più facili ed economiche da produrre rispetto al DNA sintetico, e in teoria hanno una capacità di stoccaggio ancora maggiore.
Il gruppo di ricerca Brown, supportato da una sovvenzione dell'Agenzia per i progetti di ricerca avanzata della difesa degli Stati Uniti (DARPA) guidata dalla professoressa di chimica Brenda Rubenstein, ha lavorato per trovare modi per rendere fattibile e scalabile l'archiviazione di dati di piccole molecole.
Per memorizzare i dati, il team utilizza piccole piastre di metallo disposte con 1, 500 minuscoli puntini di diametro inferiore a un millimetro. Ogni punto contiene una miscela di molecole. La presenza o l'assenza di molecole diverse in ciascuna miscela indicano i dati digitali. Il numero di bit in ogni miscela può essere grande quanto la libreria di molecole distinte disponibili per la miscelazione. I dati possono quindi essere letti utilizzando uno spettrometro di massa, che possono identificare le molecole presenti in ciascun pozzetto.
In un articolo pubblicato lo scorso anno, il team Brown ha dimostrato di poter archiviare file di immagine nell'intervallo dei kilobyte utilizzando alcuni metaboliti comuni, le molecole che gli organismi utilizzano per regolare il metabolismo. Per questo nuovo lavoro, i ricercatori sono stati in grado di espandere notevolmente le dimensioni della loro libreria, e quindi le dimensioni dei file che potevano codificare, sintetizzando le proprie molecole.
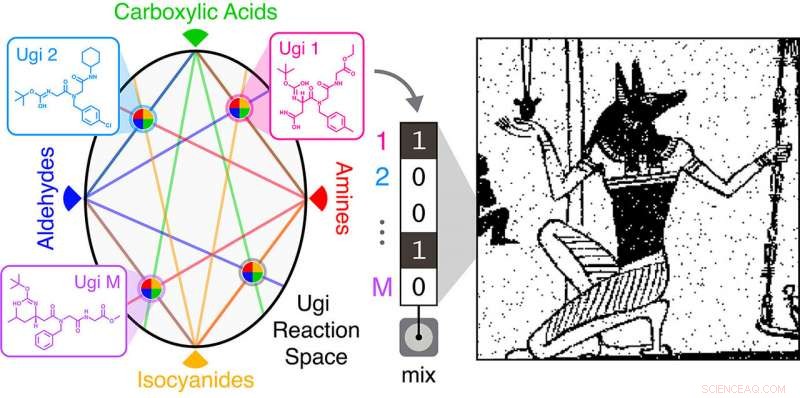
Il team ha realizzato le proprie molecole utilizzando le reazioni Ugi, una tecnica spesso utilizzata nell'industria farmaceutica per produrre rapidamente un gran numero di composti diversi. Le reazioni Ugi combinano quattro ampie classi di reagenti (un'ammina, un'aldeide o un chetone, un acido carbossilico, e un isocianuro) in una nuova molecola. Utilizzando diversi reagenti di ciascuna classe, i ricercatori potrebbero produrre rapidamente una vasta gamma di molecole distinte. Per questo lavoro, il team ha utilizzato cinque diverse ammine, cinque aldeidi, 12 acidi carbossilici, e cinque isocianuri in diverse combinazioni per creare 1, 500 composti distinti.
"Il vantaggio qui è la potenziale scalabilità della libreria, " Rubenstein ha detto. "Usiamo solo 27 componenti diversi per creare un 1, Libreria di 500 molecole in un giorno. Ciò significa che non dobbiamo uscire e trovare 1, 500 molecole uniche."
Da li, il team ha utilizzato sottobiblioteche di composti per codificare le proprie immagini. Una libreria di 32 composti è stata utilizzata per memorizzare un'immagine binaria del dio egizio Anubi. Una libreria di 575 composti è stata utilizzata per codificare un disegno di un violino di Picasso da 0,88 megapixel.
Il gran numero di molecole disponibili per le librerie chimiche ha inoltre consentito ai ricercatori di esplorare schemi di codifica alternativi che hanno reso più solida la lettura dei dati. Sebbene la spettrometria di massa sia estremamente precisa, non è perfetto. Così come con qualsiasi sistema utilizzato per memorizzare o trasmettere dati, questo sistema avrà bisogno di una qualche forma di correzione degli errori.
"Il modo in cui progettiamo le librerie e leggiamo i dati include informazioni extra che ci consentono di correggere alcuni errori, ", ha detto lo studente laureato Brown Chris Arcadia, primo autore del saggio. "Ciò ci ha aiutato a semplificare il flusso di lavoro sperimentale e ad ottenere ancora tassi di precisione fino al 99%".
C'è ancora molto lavoro da fare per portare questa idea su una scala utile, dicono i ricercatori. Ma la capacità di creare grandi librerie chimiche e usarle per codificare file sempre più grandi suggerisce che l'approccio può davvero essere scalato.
"Non siamo più limitati dalle dimensioni della nostra libreria chimica, che è davvero importante, " Rosenstein ha detto. "Questo è il più grande passo avanti qui. Quando abbiamo iniziato questo progetto alcuni anni fa, abbiamo avuto alcuni dibattiti sul fatto che qualcosa di questa scala fosse anche sperimentalmente fattibile. Quindi è davvero incoraggiante che siamo stati in grado di farlo".