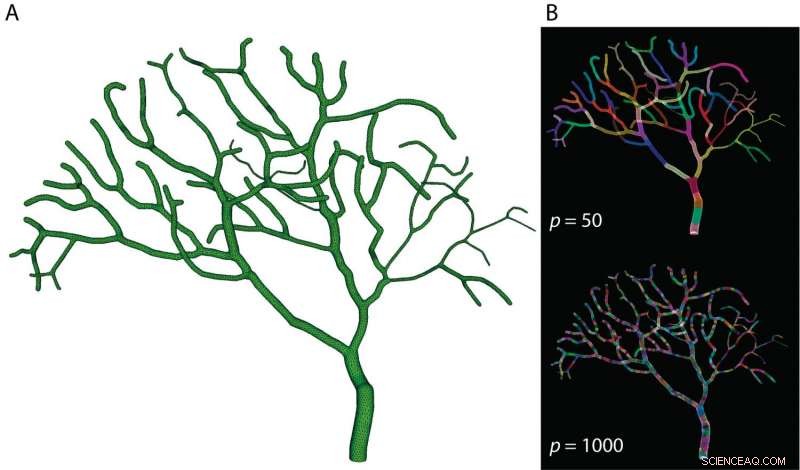
Scomponendo il modello parziale delle cellule di Purkinje (A) in 50 sezioni (B in alto) o 1000 sezioni (B in basso) ed eseguendo i calcoli di ciascuna sezione in parallelo su un supercomputer, I ricercatori dell'OIST hanno drasticamente ridotto il tempo di simulazione del modello. Credito:OIST
A differenza dei neuroscienziati sperimentali che si occupano di neuroni della vita reale, i neuroscienziati computazionali usano simulazioni di modelli per studiare come funziona il cervello. Mentre molti neuroscienziati computazionali usano modelli matematici semplificati di neuroni, i ricercatori dell'Unità di neuroscienze computazionali dell'Okinawa Institute of Science and Technology Graduate University (OIST) sviluppano un software che modella i neuroni in base ai dettagli delle interazioni molecolari con l'obiettivo di ottenere nuove informazioni sulla funzione neuronale. Finora le applicazioni del software erano limitate a causa dell'intensa potenza di calcolo richiesta per modelli neuronali così dettagliati, ma recentemente il dottor Weiliang Chen, Dottor Iain Hepburn, e il professor Erik De Schutter hanno pubblicato due articoli correlati in cui delineano l'accuratezza e la scalabilità del loro nuovo software di calcolo ad alta velocità, "PASSI Paralleli". I risultati combinati suggeriscono che Parallel STEPS potrebbe essere utilizzato per rivelare nuove intuizioni su come i singoli neuroni funzionano e comunicano tra loro.
La prima carta, pubblicato in Il Giornale di Fisica Chimica nell'agosto 2016, si concentra sull'assicurare che l'accuratezza di Parallel STEPS sia paragonabile ai metodi convenzionali. Negli approcci convenzionali, i calcoli associati alle reazioni chimiche neuronali e alla diffusione della molecola sono tutti calcolati su un'unità di elaborazione computazionale o "nucleo" in sequenza. Però, Il Dr. Iain Hepburn e colleghi hanno introdotto un nuovo approccio per eseguire calcoli di reazione e diffusione in parallelo che possono quindi essere distribuiti su più core di computer, mantenendo la precisione della simulazione a un livello elevato. La chiave era sviluppare un algoritmo originale separato in due parti:una che calcolasse gli eventi di reazione chimica e l'altra gli eventi di diffusione.
"Abbiamo testato una gamma di simulazioni di modelli da semplici modelli di diffusione a modelli biologici realistici e abbiamo scoperto che potevamo ottenere prestazioni migliorate utilizzando un approccio parallelo con una perdita minima di precisione. Ciò ha dimostrato la potenziale idoneità del metodo su scala più ampia, "dice il dottor Hepburn.
In un articolo correlato pubblicato su Le frontiere della neuroinformatica questo febbraio, Il Dr. Weiliang Chen ha presentato i dettagli di implementazione di Parallel STEPS e ne ha studiato le prestazioni e le potenziali applicazioni. Rompendo un modello parziale di una cellula di Purkinje - uno dei neuroni più grandi del cervello - in 50-1000 sezioni e simulando eventi di reazione e diffusione per ciascuna sezione in parallelo sul supercomputer Sango dell'OIST, Il Dr. Chen e colleghi hanno visto velocità di calcolo notevolmente aumentate. Hanno testato questo approccio sia su modelli semplici che su modelli più complicati di esplosioni di calcio nelle cellule di Purkinje e hanno dimostrato che la simulazione parallela potrebbe accelerare i calcoli di diverse centinaia di volte rispetto ai metodi convenzionali.
"Insieme, i nostri risultati mostrano che l'implementazione di Parallel STEPS ottiene miglioramenti significativi nelle prestazioni, e buona scalabilità, " afferma il Dr. Chen. "Modelli simili che in precedenza richiedevano mesi di simulazione possono ora essere completati in poche ore o minuti, il che significa che possiamo sviluppare e simulare modelli più complessi, e impara di più sul cervello in un lasso di tempo più breve."
Dr. Hepburn e Dr. Chen dell'Unità di neuroscienze computazionali dell'OIST, guidato dal professor Erik De Schutter, stanno collaborando attivamente con lo Human Brain Project, un'iniziativa mondiale con sede presso l'École Polytechnique Fédérale de Lausanne (EPFL) in Svizzera, sviluppare una versione più robusta di Parallel STEPS che incorpori la simulazione del campo elettrico delle membrane cellulari.
Finora STEPS è solo realisticamente in grado di modellare parti di neuroni ma con il supporto di Parallel STEPS, l'Unità di neuroscienze computazionali spera di sviluppare un modello in scala reale di un intero neurone e successivamente delle interazioni tra i neuroni in una rete. Collaborando con il team dell'EPFL e facendo uso del supercomputer IBM "Blue Gene/Q" che si trova lì, mirano a raggiungere questi obiettivi nel prossimo futuro.
"Grazie ai moderni supercomputer possiamo studiare gli eventi molecolari all'interno dei neuroni in un modo molto più trasparente di prima, " afferma il Prof. De Schutter. "La nostra ricerca apre strade interessanti nelle neuroscienze computazionali che collegano per la prima volta la biochimica con l'elettrofisiologia".