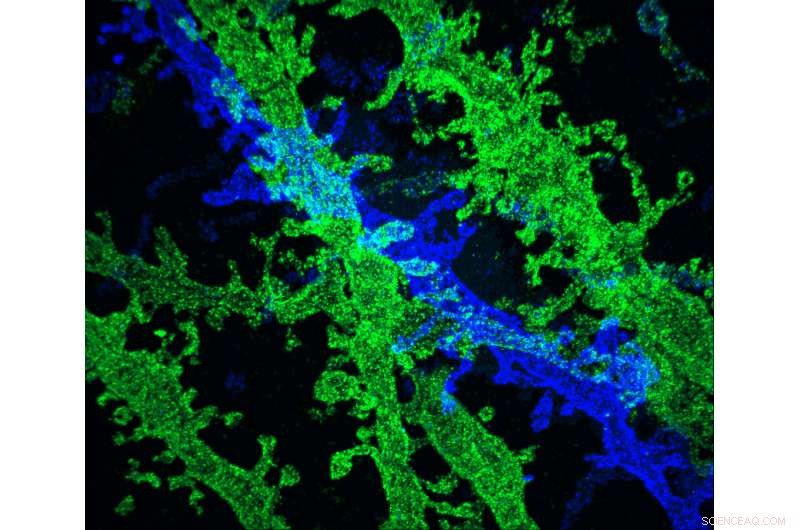
I ricercatori del MIT hanno sviluppato un modo per realizzare immagini ad altissima risoluzione di campioni di tessuto, ad una frazione del costo di altre tecniche che offrono una risoluzione simile. Credito:i ricercatori
I ricercatori del MIT hanno sviluppato un modo per realizzare immagini ad altissima risoluzione di campioni di tessuto, ad una frazione del costo di altre tecniche che offrono una risoluzione simile.
La nuova tecnica si basa sull'espansione del tessuto prima di visualizzarlo con un microscopio ottico convenzionale. Due anni fa, il team del MIT ha dimostrato che era possibile espandere i volumi dei tessuti di 100 volte, risultando in una risoluzione dell'immagine di circa 60 nanometri. Ora, i ricercatori hanno dimostrato che espandere il tessuto una seconda volta prima dell'imaging può aumentare la risoluzione a circa 25 nanometri.
Questo livello di risoluzione consente agli scienziati di vedere, Per esempio, le proteine che si raggruppano in schemi complessi nelle sinapsi cerebrali, aiutare i neuroni a comunicare tra loro. Potrebbe anche aiutare i ricercatori a mappare i circuiti neurali, dice Ed Boyden, professore associato di ingegneria biologica e scienze cerebrali e cognitive al MIT.
"Vogliamo essere in grado di tracciare il cablaggio di circuiti cerebrali completi, "dice Boyden, l'autore senior dello studio. "Se potessi ricostruire un circuito cerebrale completo, forse potresti creare un modello computazionale di come genera fenomeni complessi come decisioni ed emozioni. Dal momento che puoi mappare le biomolecole che generano impulsi elettrici all'interno delle cellule e che scambiano sostanze chimiche tra le cellule, potresti potenzialmente modellare le dinamiche del cervello."
Questo approccio potrebbe essere utilizzato anche per immaginare altri fenomeni come le interazioni tra cellule cancerose e cellule immunitarie, per rilevare gli agenti patogeni senza attrezzature costose, e per mappare i tipi di cellule del corpo.
L'ex postdoc del MIT Jae-Byum Chang è il primo autore dell'articolo, che appare nel numero del 17 aprile di Metodi della natura .
Doppia espansione
Per espandere i campioni di tessuto, i ricercatori li inseriscono in un denso, gel generato in modo uniforme in poliacrilato, un materiale molto assorbente che viene utilizzato anche nei pannolini. Prima che si formi il gel, i ricercatori etichettano le proteine cellulari che vogliono visualizzare, utilizzando anticorpi che si legano a bersagli specifici. Questi anticorpi portano "codici a barre" fatti di DNA, che a loro volta sono attaccate a molecole reticolanti che si legano ai polimeri che compongono il gel espandibile. I ricercatori quindi scompongono le proteine che normalmente tengono insieme il tessuto, consentendo ai codici a barre del DNA di espandersi l'uno dall'altro mentre il gel si gonfia.
Questi campioni ingranditi possono quindi essere etichettati con sonde fluorescenti che legano i codici a barre del DNA, e ripreso con microscopi confocali disponibili in commercio, la cui risoluzione è solitamente limitata a centinaia di nanometri.
Usando questo approccio, i ricercatori erano in precedenza in grado di raggiungere una risoluzione di circa 60 nanometri. Però, "le singole biomolecole sono molto più piccole di così, diciamo 5 nanometri o anche più piccoli, " Dice Boyden. "Le versioni originali della microscopia ad espansione erano utili per molte questioni scientifiche, ma non potevano eguagliare le prestazioni dei metodi di imaging a più alta risoluzione come la microscopia elettronica".
Nel loro studio di microscopia ad espansione originale, i ricercatori hanno scoperto che potevano espandere il tessuto di oltre 100 volte in volume riducendo il numero di molecole di reticolazione che trattengono il polimero in uno schema ordinato. Però, questo ha reso il tessuto instabile.
"Se si riduce la densità del reticolante, i polimeri non mantengono più la loro organizzazione durante il processo di espansione, "dice Boyden, che è membro del Media Lab del MIT e del McGovern Institute for Brain Research. "Perdi le informazioni."
Anziché, nel loro ultimo studio, i ricercatori hanno modificato la loro tecnica in modo che dopo la prima espansione tissutale, possono creare un nuovo gel che gonfia il tessuto una seconda volta, un approccio che chiamano "espansione iterativa".
Circuiti di mappatura
Utilizzando l'espansione iterativa, i ricercatori sono stati in grado di visualizzare i tessuti con una risoluzione di circa 25 nanometri, che è simile a quello ottenuto con tecniche ad alta risoluzione come la microscopia a ricostruzione ottica stocastica (STORM). Però, la microscopia ad espansione è molto più economica e più semplice da eseguire perché non sono necessarie attrezzature o prodotti chimici specializzati, dice Boyden. Il metodo è anche molto più veloce e quindi compatibile con Immagini 3D.
La risoluzione della microscopia ad espansione non corrisponde ancora a quella della microscopia elettronica a scansione (circa 5 nanometri) o della microscopia elettronica a trasmissione (circa 1 nanometro). Però, i microscopi elettronici sono molto costosi e non ampiamente disponibili, e con quei microscopi, è difficile per i ricercatori etichettare proteine specifiche.
Nel Metodi della natura carta, il team del MIT ha utilizzato l'espansione iterativa per visualizzare le sinapsi, le connessioni tra i neuroni che consentono loro di comunicare tra loro. Nel loro studio di microscopia ad espansione originale, i ricercatori sono stati in grado di visualizzare le proteine dell'impalcatura, che aiutano ad organizzare le centinaia di altre proteine presenti nelle sinapsi. Con il nuovo, risoluzione migliorata, i ricercatori sono stati anche in grado di vedere strutture su scala più fine, come la posizione dei recettori dei neurotrasmettitori situati sulle superfici delle cellule "postsinaptiche" sul lato ricevente della sinapsi.
"La mia speranza è che possiamo, negli anni a venire, iniziare davvero a mappare l'organizzazione di queste proteine di scaffolding e di segnalazione nella sinapsi, "dice Boyden.
La combinazione della microscopia ad espansione con un nuovo strumento chiamato multiplexing temporale dovrebbe aiutare a raggiungere questo obiettivo, lui crede. Attualmente, solo un numero limitato di sonde colorate può essere utilizzato per visualizzare diverse molecole in un campione di tessuto. Con il multiplexing temporale, i ricercatori possono etichettare una molecola con una sonda fluorescente, prendere un'immagine, e poi lavare via la sonda. Questo può essere ripetuto più volte, ogni volta usando gli stessi colori per etichettare molecole diverse.
"Combinando l'espansione iterativa con il multiplexing temporale, potremmo in linea di principio avere essenzialmente un colore infinito, imaging a risoluzione nanometrica su grandi volumi 3D, " Dice Boyden. "Le cose stanno diventando davvero entusiasmanti ora che queste diverse tecnologie potrebbero presto connettersi tra loro".
I ricercatori sperano anche di ottenere un terzo round di espansione, che credono potrebbe, in linea di principio, consentono una risoluzione di circa 5 nanometri. Però, in questo momento la risoluzione è limitata dalla dimensione degli anticorpi utilizzati per etichettare le molecole nella cellula. Questi anticorpi sono lunghi circa 10-20 nanometri, quindi per ottenere una risoluzione inferiore a quella, i ricercatori avrebbero bisogno di creare tag più piccoli o espandere prima le proteine l'una dall'altra e quindi fornire gli anticorpi dopo l'espansione.