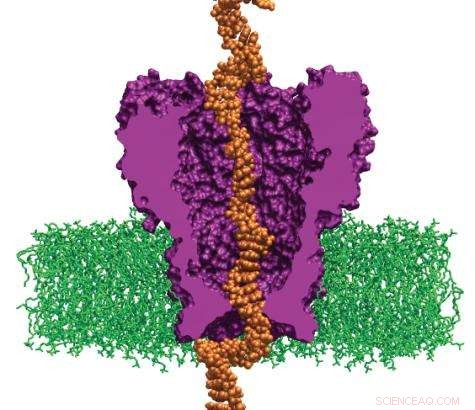
Gli scienziati simulano l'interazione del DNA con una proteina ingegnerizzata. Il sistema può rallentare i filamenti di DNA che viaggiano attraverso i pori abbastanza da leggere il genoma individuale di un paziente. (Immagine per gentile concessione di Aleksei Aksimentiev)
(PhysOrg.com) -- Il Progetto Genoma Umano ha aperto la strada alla genomica, lo studio del genoma di un organismo. La genomica personalizzata può stabilire la relazione tra le variazioni della sequenza del DNA tra gli individui e le loro condizioni di salute e le risposte a farmaci e trattamenti. Per rendere il sequenziamento del genoma una procedura di routine, però, il tempo deve essere ridotto a meno di un giorno e il costo a meno di $ 1, 000:un'impresa impossibile con le attuali conoscenze e tecnologie.
Nel 2008, un gruppo di ricerca guidato da Aleksei Aksimentiev, assistente professore nel dipartimento di fisica dell'Università dell'Illinois-Urbana-Champaign, ha iniziato un progetto per creare macchine per il sequenziamento del genoma personale che saranno più accessibili agli ospedali. Usando il giaguaro dell'Oak Ridge National Laboratory, uno dei supercomputer più veloci del mondo, Aksimentiev e il suo team stanno sviluppando un approccio a nanopori, che promette una drastica riduzione dei tempi e dei costi per il sequenziamento del DNA. La loro ricerca rivela la forma del DNA che si muove attraverso un singolo nanoporo, un poro proteico largo un miliardesimo di metro che attraversa una membrana. Quando il DNA passa attraverso il poro, la sequenza dei nucleotidi (mattoni del DNA) viene letta da un rivelatore.
"Il principale ostacolo al sequenziamento utilizzando le generazioni precedenti di nanopori biologici e sintetici era l'incapacità di identificare la sequenza del DNA alla risoluzione del singolo nucleotide, " ha detto Aksimentiev. "I nucleotidi sono passati troppo velocemente attraverso il nanoporo per consentire agli scienziati di sequenziare il DNA".
Il gruppo di Aksimentiev utilizza il nanoporo MspA, una proteina ingegnerizzata. La sua sequenza deve essere alterata per legarsi più fortemente al filamento di DNA in movimento. MspA è una piattaforma ideale per il sequenziamento del DNA perché ora gli scienziati possono misurare le dighe nei pori, che potrebbe rallentare il viaggio del DNA attraverso la proteina. Alterare la proteina MspA per ottimizzare le dighe richiede tempo e denaro in laboratorio, ma è semplice su un computer. Ad esempio, alterare la proteina in alcun modo, gli scienziati devono determinare se la particolare mutazione che introducono è stabile e se l'idea è ragionevole. Perciò, gli scienziati prima simulano MspA per decidere su una mutazione da indurre e per testare idee ad alto rischio prima di implementarle in un esperimento.
Il team di ricerca utilizza il codice NAMD, che calcola gli stati di energia minima degli atomi in un grande sistema biomolecolare ed è un indicatore di quali forme le molecole sarebbero più comode da assumere. Il team costruisce prima un modello della proteina MspA immersa in un doppio strato lipidico e una soluzione elettrolitica. Un filamento di DNA di una sequenza nucleotidica desiderata viene quindi infilato attraverso il nanoporo MspA. Successivamente gli scienziati simulano l'effetto di un campo elettrico che guida ioni e DNA attraverso il nanoporo MspA.
La simulazione utilizza la dinamica molecolare, o calcoli del moto di ogni atomo in un sistema molecolare seguendo le leggi fisiche della natura, imitare il sistema sperimentale. I risultati delle simulazioni possono essere direttamente confrontati con quelli degli esperimenti perché entrambi gli approcci misurano la corrente ionica, secondo Aksimentiev. Conoscendo le posizioni di ogni atomo e ione di DNA, gli scienziati ottengono un vantaggio:possono ottimizzare il sequenziamento dei nanopori utilizzando un design razionale per produrre un poro che aderisce più strettamente al DNA, rallentando il viaggio della molecola attraverso il poro ad una velocità che consente la risoluzione del singolo nucleotide.
Il lavoro di sequenziamento è finanziato dal National Human Genome Research Institute dei National Institutes of Health. Lo sviluppo del metodo del progetto è finanziato in parte dalla National Science Foundation. I collaboratori del progetto includono due gruppi sperimentali:uno guidato da Jens Gundlach dell'Università di Washington-Seattle e l'altro da Michael Niederweis dell'Università dell'Alabama-Birmingham.
La ricerca ha ricevuto 10 milioni di ore di elaborazione su Jaguar attraverso l'Innovative and Novel Computational Impact on Theory and Experiment, o INCITE, programma, che assegna allocazioni considerevoli su alcuni dei supercomputer più potenti del mondo a progetti che affrontano grandi sfide nel campo della scienza e dell'ingegneria. Con l'assegnazione INCITE, gli scienziati sono stati in grado di riprodurre le dighe nel nanoporo MspA per il tipo di nucleotidi di DNA ad esso confinati, rallentando il movimento della sequenza attraverso il nanoporo.
"Abbiamo effettuato uno studio pilota su diverse varianti del nanoporo MspA e osservato una notevole riduzione della velocità del filamento di DNA, " ha detto Aksimentiev. "Questi risultati molto preliminari suggeriscono che il raggiungimento di una riduzione di 100 volte della velocità del DNA, che dovrebbe essere sufficiente per leggere la sequenza di DNA con risoluzione del singolo nucleotide, è a portata di mano. Gli studi futuri saranno diretti verso questo obiettivo".
Il team spera di raggiungere l'obiettivo di questo progetto entro il 2013 e prevede di perseguire una serie di interessanti progetti spin-off, ha detto Aksimentiev. La capacità di rendere accessibile il sequenziamento del genoma consentirà programmi come il Cancer Genome Project, che caratterizza le mutazioni del DNA nelle cellule tumorali in vari tessuti in tutte le fasi dello sviluppo del cancro.