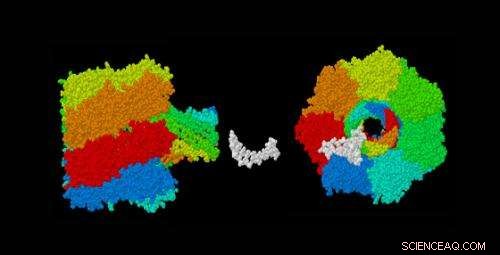
Questa è un'immagine del poro dell'alfa-emolisina (composto da 7 subunità identiche in 7 colori) e del DNA a singolo filamento di 12 mer (in bianco) sulla stessa scala per illustrare gli effetti del DNA sulla conduttanza quando si muove attraverso un nanoporo. Di seguito è riportata una vista ortogonale delle stesse molecole. Immagine creata il 5 ottobre 2008 da George Church utilizzando Rasmol_2.6 e coordinate da 7AHL.pdb e 1BNA.pdb Credito:George Church
Non è facile guidare lunghe catene di molecole - come il DNA - attraverso un "nanoporo" (un poro largo solo pochi milionesimi di millimetro) perché tendono ad aggrovigliarsi. Una simulazione effettuata da un gruppo internazionale di scienziati - tra cui i ricercatori della SISSA - ha suggerito una soluzione:meglio "tirare" dolcemente senza applicare troppa forza, in caso contrario la molecola potrebbe essere fermata a causa dell'eccessivo attrito. Questa è un'osservazione importante per creare metodi innovativi di sequenziamento del DNA.
Con il progredire della nanotecnologia, diventa sempre più importante conoscere nel dettaglio le dinamiche del nanomondo (il mondo alla scala del milionesimo di millimetro). Che succede, Per esempio, quando proviamo a guidare un polielettrolita (una lunga catena di molecole caricate elettricamente, come il DNA) attraverso un nanoporo se i nodi causano l'inceppamento del processo di traslocazione? Non è una domanda inutile, perché ora un nuovo metodo di sequenziamento del DNA per analizzare elettrochimicamente ogni singolo filamento guidandolo attraverso un nanoporo, viene sviluppato. Poiché quei fili tendono ad aggrovigliarsi se sono molto lunghi, Angelo Rosa della International School for Advanced Studies e i suoi colleghi si sono proposti di studiare teoricamente le dinamiche di questa traslocazione, effettuando una simulazione.
Il modello scelto dagli scienziati ha dimostrato che l'inceppamento non è causato dalla semplice presenza del nodo, ma dalla relazione tra l'attrito e la forza applicata per spingere la molecola nell'intercapedine." Il risultato non è così scontato se confrontato con quanto accade a livello macro, "ha spiegato Cristian Micheletti, ricercatore alla SISSA e uno degli autori del paper pubblicato su Lettere di revisione fisica . "I nodi introducono un attrito efficace che aumenta con la forza applicata e tira il polimero dall'altra parte del nanoporo. La traslocazione viene interrotta solo al di sopra di una forza di soglia".
"Secondo quanto abbiamo osservato nella simulazione, per evitare l'ostruzione del poro e l'arresto della traslocazione, la forza applicata dovrebbe essere controllata, senza tirare troppo" ha spiegato Rosa.
Questo studio è solo un primo passo. Per i dettagli quantitativi su questo processo (cos'è questa soglia e come dovrebbe essere misurata la forza per massimizzare l'efficacia di questo metodo di sequenziamento) saranno necessari approfondimenti sia a livello teorico (il modello sviluppato da Rosa, Di Ventra e Micheletti è mesoscopico, non atomistico) e a livello sperimentale.
Più in dettaglio…
Il nanoporesequencing è una tecnica innovativa, un'alternativa ai metodi più tradizionali come la PCA. Questo metodo consiste nel separare i due filamenti della base azotata che compongono la doppia elica del DNA e analizzarli uno per uno. Ogni filo viene guidato attraverso un nanoporo mentre vengono registrate le variazioni elettriche nella traslocazione. Questo è un metodo elettrochimico:le alterazioni del campo elettrico danno informazioni sulla composizione chimica della molecola spinta attraverso il poro e la composizione viene così ricostruita. Finora questo metodo ha dato buoni risultati con brevi frammenti di DNA, mentre si sono incontrate difficoltà per quelle più lunghe, a causa dei nodi. Ecco perché studi come Rosa, Di Ventra e Micheletti sono un passo importante per aumentarne l'efficienza.