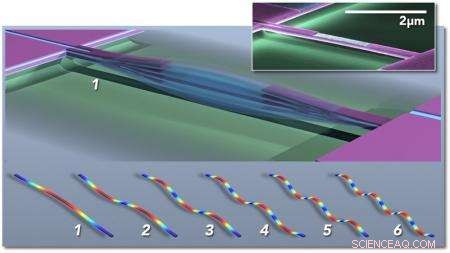
Sensore di massa basato su sistemi nanoelettromeccanici multimodali (NEMS); la figura principale rappresenta schematicamente una trave a doppia morsa che vibra in modo fondamentale (1). Di seguito sono mostrate le "istantanee" concettuali dei primi sei modi vibrazionali (1-6), i colori indicano una tensione da alta (rosso) a bassa (blu). L'inserto mostra una micrografia elettronica colorata di un risonatore NEMS piezoelettrico fabbricato nel Kavli Nanoscience Institute di Caltech. Credito:M. Matheny, L.G. Villanueva, P. Hung, J. Li e M. Roukes/Caltech
Basandosi sulla creazione del primo dispositivo meccanico in grado di misurare la massa delle singole molecole, uno alla volta, un team di scienziati del Caltech e i loro colleghi hanno creato nanodispositivi che possono anche rivelare la loro forma. Tali informazioni sono cruciali quando si cerca di identificare grandi molecole proteiche o complessi complessi di molecole proteiche.
"Puoi immaginare che con grandi complessi proteici composti da molti diversi, subunità più piccole ci sono molti modi per assemblarle. Questi possono finire per avere masse abbastanza simili pur essendo in realtà specie diverse con diverse funzioni biologiche. Ciò è particolarmente vero con gli enzimi, proteine che mediano le reazioni chimiche nel corpo, e proteine di membrana che controllano le interazioni di una cellula con il suo ambiente, " spiega Michael Roukes, il professore di fisica Robert M. Abbey, Fisiche applicate, e Bioingegneria al Caltech e l'autore corrispondente di un articolo che descrive la tecnologia apparso il 30 marzo nel numero online della rivista Nanotecnologia della natura .
Uno dei fondamenti della rivoluzione genomica è stata la capacità di replicare in massa molecole di DNA o RNA utilizzando la reazione a catena della polimerasi per creare i molti milioni di copie necessarie per il tipico sequenziamento e analisi. Però, la stessa tecnologia di produzione di massa non funziona per copiare le proteine. Proprio adesso, se vuoi identificare correttamente una particolare proteina, ne hai bisogno molto, in genere milioni di copie della sola proteina di interesse, con pochissime altre proteine estranee come contaminanti. La massa media di questa popolazione molecolare viene quindi valutata con una tecnica chiamata spettrometria di massa, in cui le molecole vengono ionizzate, in modo che ottengano una carica elettrica, e poi lasciate interagire con un campo elettromagnetico. Analizzando questa interazione, gli scienziati possono dedurre il rapporto tra massa molecolare e carica.
Ma la spettrometria di massa spesso non è in grado di discriminare differenze sottili ma cruciali nelle molecole con rapporti massa-carica simili. "Con la spettrometria di massa oggi, " spiega Roukes, "Le grandi molecole e i complessi molecolari vengono prima sminuzzati in tanti pezzi più piccoli, questo è, in frammenti molecolari più piccoli che gli strumenti esistenti possono gestire. Questi diversi frammenti vengono analizzati separatamente, e poi la bioinformatica, che implica simulazioni al computer, viene utilizzata per rimettere insieme il puzzle. Ma questo processo di riassemblaggio può essere ostacolato se pezzi di complessi diversi vengono mischiati insieme".
Con i loro dispositivi, Roukes e i suoi colleghi possono misurare la massa di una singola molecola intatta. Ogni dispositivo, che misura solo un paio di milionesimi di metro o meno, è costituito da una struttura vibrante chiamata risonatore del sistema nanoelettromeccanico (NEMS). Quando una particella o una molecola atterra sul nanodispositivo, la massa aggiunta cambia la frequenza alla quale la struttura vibra, proprio come mettere gocce di saldatura su una corda di chitarra cambierebbe la frequenza della sua vibrazione e il tono risultante. Gli spostamenti di frequenza indotti forniscono informazioni sulla massa della particella. Ma anche loro come descritto nel nuovo documento, può essere utilizzato per determinare la distribuzione spaziale tridimensionale della massa:cioè, la forma della particella.
"Una corda di chitarra non vibra solo a una frequenza, " dice Roukes. "Ci sono armoniche del suo tono fondamentale, o cosiddetti modi vibrazionali. Ciò che distingue una corda di violino da una corda di chitarra sono proprio le diverse mescolanze di queste diverse armoniche del tono fondamentale. Lo stesso vale qui. Abbiamo un sacco di toni diversi che possono essere eccitati contemporaneamente su ciascuno dei nostri nanodispositivi, e tracciamo molti toni diversi in tempo reale. Si scopre che quando la molecola atterra con orientamenti diversi, quelle armoniche sono spostate in modo diverso. Possiamo quindi utilizzare la teoria dell'imaging inerziale che abbiamo sviluppato per ricostruire un'immagine nello spazio della forma della molecola".
"La nuova tecnica scopre una capacità precedentemente non realizzata dei sensori meccanici, " afferma il professor Mehmet Selim Hanay della Bilkent University di Ankara, Tacchino, un ex ricercatore post-dottorato nel laboratorio di Roukes e co-primo autore dell'articolo. "In precedenza abbiamo identificato molecole, come l'anticorpo IgM, basandosi esclusivamente sui loro pesi molecolari. Ora, consentendo di dedurre contemporaneamente sia il peso molecolare che le informazioni sulla forma per la stessa molecola, la nuova tecnica può migliorare notevolmente il processo di identificazione, e questo è importante sia per la ricerca di base che per l'industria farmaceutica".
Attualmente, strutture molecolari vengono decifrate mediante cristallografia a raggi X, una tecnica spesso laboriosa che comporta l'isolamento, purificante, e poi cristallizzando le molecole, e quindi valutando la loro forma in base ai modelli di diffrazione prodotti quando i raggi X interagiscono con gli atomi che insieme formano i cristalli. Però, molte molecole biologiche complesse sono difficili se non impossibili da cristallizzare. E, anche quando possono essere cristallizzati, la struttura molecolare ottenuta rappresenta la molecola allo stato cristallino, che può essere molto diverso dalla struttura della molecola nella sua forma biologicamente attiva.
"Puoi immaginare situazioni in cui non sai esattamente cosa stai cercando:dove sei in modalità di scoperta, e stai cercando di capire la risposta immunitaria del corpo a un particolare agente patogeno, Per esempio, " dice Roukes. In questi casi, la capacità di eseguire il rilevamento di singole molecole e di ottenere il maggior numero possibile di informazioni separate su quella singola molecola migliora notevolmente le probabilità di effettuare un'identificazione univoca.
"Diciamo che il cancro inizia spesso con una singola cellula aberrante, e ciò significa che anche se potrebbe essere una di una molteplicità di cellule simili, c'è qualcosa di unico nella composizione molecolare di quella cellula. Con questa tecnica, abbiamo potenzialmente un nuovo strumento per capire cosa c'è di unico al riguardo, " Aggiunge.
Finora, la nuova tecnica è stata validata utilizzando particelle di dimensioni e forme note, come nanogoccioline polimeriche. Roukes e colleghi dimostrano che con i nanodispositivi all'avanguardia di oggi, l'approccio può fornire una risoluzione su scala molecolare, ovvero fornire la capacità di vedere i sottocomponenti molecolari dell'individuo, assemblaggi proteici intatti. Gli attuali sforzi del gruppo sono ora concentrati su tali esplorazioni.
Scott Kelber, un ex studente laureato nel laboratorio di Roukes, è l'altro co-primo autore dell'articolo, intitolato "Imaging inerziale con sistemi nanoelettromeccanici". Professor John Sader dell'Università di Melbourne, Australia, e un visiting associate in fisica al Caltech, è l'autore corrispondente. Altri coautori sono Cathal D. O'Connell e Paul Mulvaney dell'Università di Melbourne. Il lavoro è stato finanziato dal premio Pioneer di un National Institutes of Health Director, una Distinguished Visiting Professorship del Caltech Kavli Nanoscience Institute, la Fondation pour la Recherche et l'Enseignement Superieur di Parigi, e il programma di sovvenzioni dell'Australian Research Council.