Le entità replicanti. Credito:(c) Junghoon Kim et al. Nanotecnologia della natura , doi:10.1038/nnano.2015.87
(Phys.org)—È possibile progettare nanomateriali autoreplicanti? Potrebbe essere se prendiamo in prestito i mattoni della natura. Il DNA è una molecola autoreplicante in cui le sue parti componenti, nucleotidi, hanno interazioni chimiche specifiche che consentono la progettazione di strutture autoassemblate. Nei sistemi biologici, Il DNA si replica con l'aiuto di proteine. Però, Junghoon Kim, Junwye Lee, Shogo Hamada, Satoshi Murata, e Sung Ha Park della Sungkyunkwan University e della Tohoku University hanno progettato un sistema auto-replicabile controllabile che non richiede proteine. Il loro lavoro appare in Nanotecnologia della natura .
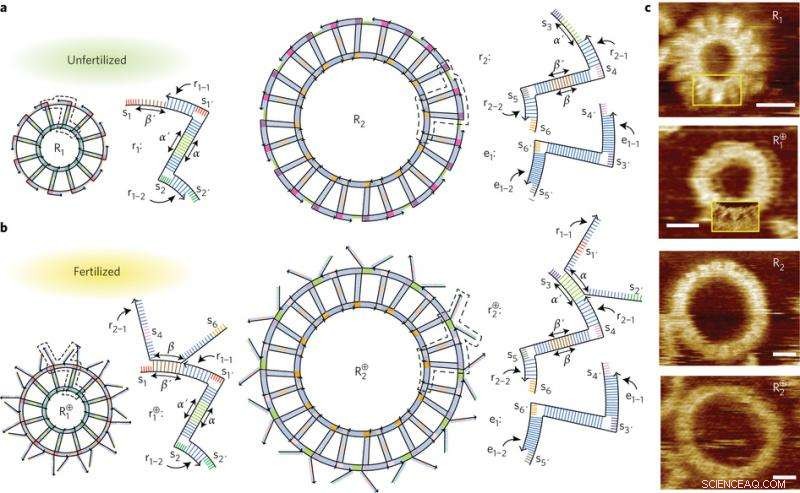
Per capire come funziona questo processo di autoreplicazione, è importante conoscere le diverse parti componenti. Kim et al. progettato due motivi a T del DNA, R 1 e r 2 , che sono DNA a doppio filamento composto da domini funzionali, etichettati alfa e beta, e le estremità "appiccicose" come punti di connessione. Hanno anche progettato un motivo di estensione. Dodici unità della r 1 motivo autoassemblante in un piccolo anello, R 1 , e dodici unità di r 2 più dodici motivi di estensione che si autoassemblano in un anello più grande, R 2 .
Questi componenti possono trovarsi in due stati diversi, "fertilizzata" o "non fecondata". Le strutture fertilizzate contengono le caratteristiche necessarie per la replicazione. La fecondazione avviene quando un dominio alfa o beta a singolo filamento di un r 1 o r 2 motivo si lega a un filamento avente un dominio alfa o beta complementare. Questo lascia una sporgenza a filamento singolo, o presa per i piedi, che si estende dall'anello o dal motivo originale. Gli appigli indicano che l'anello o il motivo è fecondato.
Questi appigli che si estendono dall'anello del DNA si legano a filamenti di invasori complementari. Quando questo accade, la struttura ibridata costituita dalla punta e dal filo invasore si stacca dall'anello iniziale, ed eventualmente, poiché questi pezzi si staccano a causa della migrazione dei rami, si autoassemblano in un altro anello.
Questo processo continua attraverso due diversi percorsi di replica. Un percorso cresce in modo esponenziale. L'altro percorso cresce secondo la sequenza di Fibonacci. Il particolare percorso seguito dipende da quali filamenti invasori vengono aggiunti al sistema.
Gli autori hanno verificato che le popolazioni dell'anello del DNA sono cresciute attraverso questo processo mediato dalla presa di piede con AFM e studi di assorbanza. Per gli studi AFM, hanno prelevato un piccolo campione da ciascuna fase e hanno determinato il numero medio di anelli presenti in quella fase. I dati sull'assorbanza sono stati aggiustati per determinare la concentrazione relativa degli anelli in ciascuna fase.
Hanno anche verificato che gli anelli figli erano il risultato della ricottura alle prese a singolo filamento dall'anello iniziale piuttosto che come risultato dell'autoassemblaggio di motivi di DNA residui in soluzione utilizzando l'elettroforesi su gel ed estraendo i prodotti del DNA da ciascuna fase. Le singole fasi sono state studiate con AFM e i filamenti invasori sono stati aggiunti a una soluzione durante ciascuna delle fasi per vedere se si formavano anelli.
Kim, et al. hanno dimostrato che l'autoreplicazione su scala nanometrica può avvenire utilizzando le proprietà termodinamiche dello spostamento del filamento mediato dalla presa e le capacità di autoassemblaggio dei motivi del DNA. In questo studio i motivi a T del DNA sintetico si autoassemblano in strutture che consentono reazioni sequenziali. Questa ricerca dimostra la possibilità di nanostrutture autoreplicanti funzionalmente programmabili.
© 2015 Phys.org