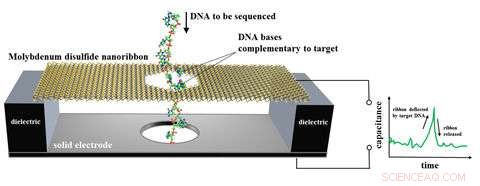
Il progetto proposto dal NIST per un sequenziatore di DNA basato su un sensore di movimento elettronico. Un nastro su scala nanometrica di bisolfuro di molibdeno è sospeso su un elettrodo metallico e immerso in acqua. DNA a singolo filamento, contenente una catena di basi (bit di codice genetico), è infilato attraverso un foro nel nastro, che si flette solo quando una base del DNA si accoppia e poi si separa da una base complementare fissata al foro. Il movimento della membrana viene rilevato come segnale elettrico. Le simulazioni numeriche e le stime teoriche mostrano che la membrana sarebbe accurata dal 79 all'86 percento nell'identificare le basi del DNA in una singola misurazione a velocità fino a circa 70 milioni di basi al secondo. Credito:NIST
Ricercatori del National Institute of Standards and Technology (NIST) e collaboratori hanno proposto un progetto per il primo sequenziatore di DNA basato su un nanosensore elettronico in grado di rilevare piccoli movimenti piccoli come un singolo atomo.
Il dispositivo proposto, un tipo di condensatore, che immagazzina la carica elettrica, è un minuscolo nastro di bisolfuro di molibdeno sospeso su un elettrodo di metallo e immerso nell'acqua. Il nastro è di 15,5 nanometri (nm, miliardesimi di metro) di lunghezza e 4,5 nm di larghezza. DNA a singolo filamento, contenente una catena di basi (bit di codice genetico), viene infilato attraverso un foro largo 2,5 nm nel nastro sottile. Il nastro si flette solo quando una base di DNA si accoppia e poi si separa da una base complementare fissata al foro. Il movimento della membrana viene rilevato come segnale elettrico.
Come descritto in un nuovo documento, il team del NIST ha effettuato simulazioni numeriche e stime teoriche per mostrare che la membrana sarebbe accurata dal 79 all'86 percento nell'identificare le basi del DNA in una singola misurazione a velocità fino a circa 70 milioni di basi al secondo. I circuiti integrati rileverebbero e misurerebbero i segnali elettrici e identificherebbero le basi. I risultati suggeriscono che un tale dispositivo potrebbe essere un veloce, sequenziatore di DNA accurato ed economico, secondo la carta.
Sequenziamento convenzionale, sviluppato negli anni '70, comporta la separazione, copiando, etichettare e riassemblare pezzi di DNA per leggere le informazioni genetiche. I metodi più recenti includono il sequenziamento automatizzato di molti frammenti di DNA contemporaneamente, ancora costoso, e nuovi concetti di "sequenziamento dei nanopori". Per esempio, lo stesso gruppo del NIST ha recentemente dimostrato l'idea di sequenziare il DNA facendolo passare attraverso un nanoporo di grafene, e misurare come le proprietà elettroniche del grafene rispondono allo sforzo.
L'ultima proposta del NIST si basa su un film sottile di bisolfuro di molibdeno, un materiale stratificato che conduce elettricità e viene spesso utilizzato come lubrificante. Tra gli altri vantaggi, questo materiale non si attacca al DNA, che può essere un problema con il grafene. Il team del NIST suggerisce che il metodo potrebbe funzionare anche senza un nanoporo, un design più semplice, facendo passare il DNA attraverso il bordo della membrana.
"Questo approccio risolve potenzialmente il problema con il DNA che si attacca al grafene se inserito in modo improprio, perché questo approccio non utilizza il grafene, periodo, Il teorico del NIST e autore principale Alex Smolyanitsky ha dichiarato:"Un'altra importante differenza è che invece di fare affidamento sulle proprietà del grafene o di qualsiasi materiale particolare utilizzato, leggiamo i movimenti elettricamente in modo più semplice formando un condensatore. Ciò rende qualsiasi membrana elettricamente conduttiva adatta all'applicazione."
L'esperto di nanomateriali Boris Yakobson della Rice University, un coautore sulla carta, suggerito l'idea del condensatore. Il supporto computazionale è stato fornito dall'Università di Groningen nei Paesi Bassi.
Il DNA ha quattro basi. Per le simulazioni, citosina (C), che si accoppia naturalmente con la guanina (G), è attaccato all'interno del poro. Quando un pezzo di DNA passa attraverso il poro, qualsiasi G nel filo si attacca temporaneamente alla C incorporata, tirando il nanonastro e segnalando l'elettrodo. La sequenza del DNA è determinata misurando come e quando i segnali elettrici variano nel tempo. Per rilevare tutte e quattro le basi, quattro nanonastri, ciascuno con una base diversa attaccata al poro, possono essere impilati verticalmente per creare un sensore DNA integrato.
Il nastro di bisolfuro di molibdeno è abbastanza flessibile da deformarsi in modo misurabile in risposta alle forze necessarie per rompere una coppia di DNA, ma abbastanza rigido per avere meno in corso, movimento insignificante del grafene, potenzialmente riducendo il rumore indesiderato nei segnali di sequenziamento. La deflessione del nastro è estremamente piccola, nell'ordine di un angstrom, le dimensioni di un atomo di idrogeno. La sua forza di trazione è dell'ordine di 50 piconewton, o trilionesimi di newton, abbastanza per rompere i delicati legami chimici tra le basi del DNA.
I ricercatori hanno stimato le prestazioni del dispositivo in un circuito integrato e hanno scoperto che le correnti di picco attraverso il condensatore erano misurabili (da 50 a 70 picoampere), anche per i piccoli nanonastri studiati. Si prevede che i picchi attuali saranno ancora maggiori nei sistemi fisici. La dimensione del dispositivo potrebbe essere modificata per rendere ancora più semplice misurare i segnali di sequenziamento.
Gli autori del NIST sperano di costruire una versione fisica del dispositivo in futuro. Per applicazioni pratiche, la tecnologia microfluidica di sequenziamento del DNA delle dimensioni di un chip potrebbe essere combinata con l'elettronica in un unico dispositivo abbastanza piccolo da essere tenuto in mano.