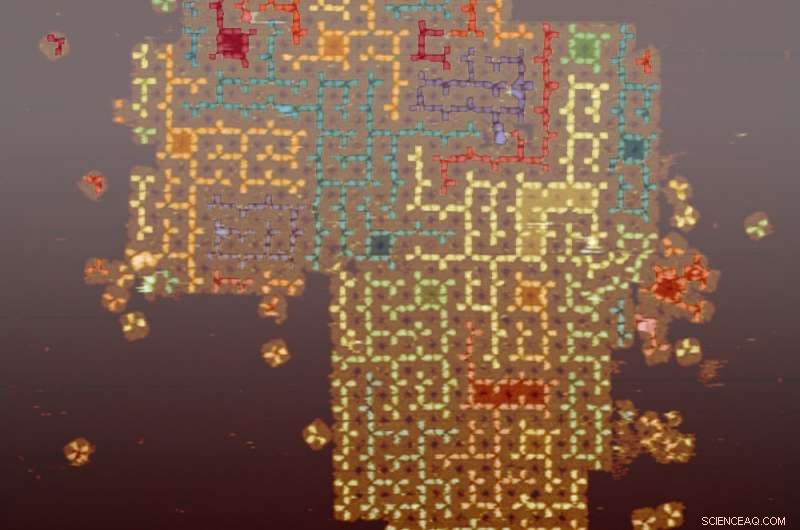
Immagine colorata al microscopio a forza atomica di strutture ad albero casuali autoassemblate sulla superficie di matrici di tessere di DNA. Ogni albero ha un singolo ciclo come "radice". Credito:Caltech / Grigory Tikhomirov, Philip Petersen e Lulu Qian.
Molti sistemi auto-organizzati in natura sfruttano una sofisticata miscela di processi deterministici e casuali. Non esistono due alberi esattamente uguali perché la crescita è casuale, ma un Redwood può essere facilmente distinto da un Jacaranda poiché le due specie seguono programmi genetici diversi. Il valore della casualità negli organismi biologici non è completamente compreso, ma è stato ipotizzato che consenta dimensioni del genoma più piccole, perché non tutti i dettagli devono essere codificati. La casualità fornisce anche la variazione alla base dell'evoluzione adattativa.
A differenza della biologia, l'ingegneria raramente sfrutta il potere della casualità per fabbricare strutture complesse. Ora, un gruppo di scienziati del Caltech ha dimostrato che la casualità nell'autoassemblaggio molecolare può essere combinata con regole deterministiche per produrre nanostrutture complesse a partire dal DNA.
Il lavoro, fatto nel laboratorio del Professore Associato di Bioingegneria Lulu Qian, appare nel numero del 28 novembre della rivista Nanotecnologia della natura .
Gli esseri viventi usano il DNA per immagazzinare informazioni genetiche, ma il DNA può anche essere usato come un robusto elemento chimico per l'ingegneria molecolare. Le quattro molecole complementari che compongono il DNA, chiamati nucleotidi, si legano insieme solo in modi specifici:gli A si legano ai T, e le G si legano alle C. Nel 2006, Paul Rothemund (BS '94), professore di ricerca di bioingegneria, scienze informatiche e matematiche, e sistemi di calcolo e neurali al Caltech, ha inventato una tecnica chiamata DNA origami che sfrutta la corrispondenza tra lunghi filamenti di nucleotidi di DNA, piegandoli in qualsiasi cosa, dalle opere d'arte su nanoscala ai dispositivi per la somministrazione di farmaci. Le strutture autoassemblate formate attraverso gli origami di DNA possono essere funzionali da sole o possono essere utilizzate come modelli per organizzare altre molecole funzionali, come i nanotubi di carbonio, proteine, nanoparticelle metalliche, e coloranti organici, con una programmabilità e una precisione spaziale senza precedenti.
Usando l'origami del DNA come elemento costitutivo, i ricercatori hanno realizzato nanostrutture di DNA più grandi, come matrici periodiche di tessere origami. Però, perché l'elemento costitutivo si ripete ovunque, la complessità dei modelli che possono essere formati su queste strutture più grandi è piuttosto limitata. Processi di assemblaggio completamente deterministici, che controllano il design di ogni singola piastrella e la sua posizione distinta nell'array, possono dare origine a modelli complessi, ma questi processi non si adattano bene. Al contrario, se sono coinvolti solo processi casuali e le caratteristiche globali dell'array non sono controllate da regole di progettazione, è impossibile creare modelli complessi con proprietà desiderate senza generare contemporaneamente una grande frazione di molecole indesiderate che vengono sprecate. Fino al lavoro di Qian e dei suoi colleghi, combinare processi deterministici con quelli casuali non era mai stato esplorato sistematicamente per creare complesse nanostrutture di DNA.

Sinistra, Le tessere Truchet hanno due archi che sono asimmetrici di rotazione. Destra, popolari giochi da tavolo ispirati alle tessere Truchet. Credito:per gentile concessione di L. Qian
"Cercavamo principi di autoassemblaggio molecolare che abbracciassero aspetti sia deterministici che casuali, " dice Qian. "Abbiamo sviluppato una semplice serie di regole che consentono alle tessere del DNA di legarsi in modo casuale ma solo in modelli controllati specifici".
L'approccio prevede la progettazione di modelli su singole piastrelle, modulando i rapporti delle diverse tessere, e determinare quali piastrelle possono unirsi durante l'autoassemblaggio. Ciò porta a caratteristiche emergenti su larga scala con proprietà statistiche sintonizzabili, un fenomeno che gli autori chiamano "disturbo programmabile".
"Le strutture che possiamo costruire hanno aspetti programmabilmente casuali, "dice Grigory Tikhomirov, uno studioso postdottorato senior in biologia e ingegneria biologica, e autore principale della carta. "Per esempio, possiamo creare strutture che hanno linee che prendono percorsi apparentemente casuali, ma possiamo garantire che non si intersechino mai e che alla fine si chiudano sempre in loop".
Oltre ai loop, il team ha scelto altri due esempi, labirinti e alberi, per dimostrare che molte proprietà non banali di queste strutture possono essere controllate da semplici regole locali. Hanno trovato questi esempi interessanti perché loop, labirinto, e le strutture ad albero esistono ampiamente in natura su più scale. Per esempio, i polmoni sono strutture ad albero su una scala da millimetri a centimetri, e i dendriti neurali sono strutture ad albero su scala da micrometrica a millimetrica. Le proprietà controllate che hanno mostrato includono le regole di ramificazione, le direzioni di crescita, la vicinanza tra reti adiacenti, e la distribuzione delle dimensioni.
Il gruppo si è inizialmente ispirato alle classiche piastrelle Truchet, che sono tessere quadrate con due archi di DNA diagonalmente simmetrici sulla superficie. Ci sono due orientamenti rotazionalmente asimmetrici del motivo ad arco. Consentendo una scelta casuale dei due orientamenti delle tessere in ciascuna posizione nell'array, il motivo continuerà attraverso le tessere vicine, o diventando loop di varie dimensioni o uscendo da un bordo dell'array.

Anello autoassemblato, labirinto, e strutture ad albero sulla superficie degli array di tessere di DNA. Riga superiore, labirinti casuali con giunzioni a tre e quattro vie di distanze variabili tra giunzioni adiacenti contro solo giunzioni a tre vie di una distanza fissa tra giunzioni adiacenti. riga centrale, alberi casuali (ogni albero ha un singolo ciclo come "radice") con rami più lunghi di lunghezza variabile rispetto a rami più corti di lunghezza fissa. riga inferiore, loop casuali con lunghezze sintonizzabili e numero di incroci. Credito:L. Qian
Per creare array Truchet su scala molecolare, il team ha utilizzato la tecnica dell'origami del DNA per piegare il DNA in tessere quadrate e quindi ha progettato le interazioni tra queste tessere per incoraggiarle ad autoassemblarsi in grandi matrici bidimensionali.
"Poiché tutte le molecole si scontrano mentre galleggiano in una provetta durante il processo di autoassemblaggio, le interazioni dovrebbero essere sufficientemente deboli da consentire alle tessere di riorganizzarsi ed evitare di essere intrappolate in configurazioni indesiderate, "dice Philip Petersen, uno studente laureato nel laboratorio Qian e co-primo autore della carta. "D'altra parte, le interazioni dovrebbero essere sufficientemente specifiche in modo che le interazioni desiderate siano sempre molto preferite rispetto a quelle indesiderate, interazioni spurie".
Diversi tipi di pattern globali emergono quando i riquadri sono contrassegnati con diversi pattern locali. Per esempio, se ogni tessera orientata in modo casuale porta una "T" anziché due archi, il modello globale è un labirinto con rami e anelli invece che solo anelli. Se le regole di autoassemblaggio vincolano il possibile orientamento relativo delle tessere a "T" adiacenti, è possibile garantire che oltre a una singola "radice, " i rami nei labirinti non si chiudono mai in anelli, producendo alberi. Per esplorare la piena generalità di questi principi, Il team di Qian ha sviluppato un linguaggio di programmazione per tessere casuali di origami del DNA.
"Con questo linguaggio di programmazione, il processo di progettazione inizia con una descrizione di alto livello delle tessere e degli array, che può essere automaticamente tradotto in diagrammi di array astratti e simulazioni numeriche, quindi passa al design delle tessere origami DNA, incluso il modo in cui le tessere interagiscono tra loro sui bordi. Finalmente, progettiamo sequenze di DNA, " dice Qian. "Con queste sequenze di DNA, è semplice per i ricercatori ordinare i filamenti di DNA, mescolarli in una provetta, attendere che le molecole si autoassemblano nelle strutture progettate durante la notte, e ottenere immagini delle strutture utilizzando un microscopio a forza atomica".
Il metodo del disordine programmabile del gruppo ha diverse applicazioni future. Per esempio, potrebbe essere utilizzato per costruire ambienti di test complessi per robot molecolari sempre più sofisticati:macchine su nanoscala basate sul DNA che possono muoversi su una superficie, raccogliere o rilasciare proteine o altri tipi di molecole come carichi, e prendere decisioni sulla navigazione e le azioni.
"Le potenziali applicazioni sono molto più ampie, " aggiunge Qian. Dagli anni '90, catene unidimensionali casuali di polimeri sono state utilizzate per rivoluzionare la sintesi chimica e dei materiali, consegna farmaci, e la chimica degli acidi nucleici creando vaste librerie combinatorie di molecole candidate e quindi selezionando o evolvendo le migliori in laboratorio. "Il nostro lavoro estende lo stesso principio alle reti bidimensionali di molecole e ora crea nuove opportunità per fabbricare dispositivi molecolari più complessi organizzati da nanostrutture di DNA, " lei dice.
Il documento è intitolato "Disturbo programmabile in tassellature casuali del DNA".