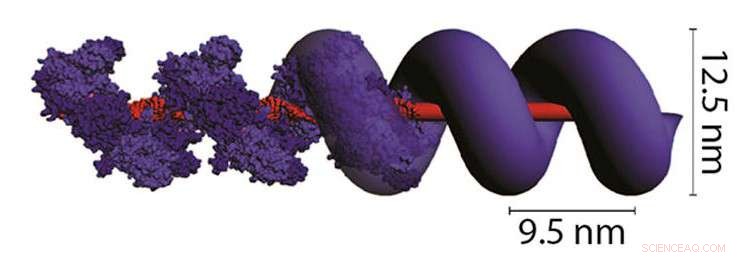
La proteina RecA (unità viola), avvolge e fortifica il DNA a doppia elica, consentendo agli scienziati di costruire grandi strutture con il materiale genetico. Credito:NIST
Il DNA è la sostanza della vita, ma è anche roba da nanotecnologia. Poiché le molecole di DNA con strutture chimiche complementari si riconoscono e si legano tra loro, filamenti di DNA possono combaciare come i mattoncini Lego per creare oggetti su scala nanometrica di forma e struttura complesse.
Ma i ricercatori devono lavorare con assemblaggi di DNA molto più grandi per realizzare un obiettivo chiave:costruire dispositivi in miniatura durevoli come biosensori e contenitori per la somministrazione di farmaci. È stato difficile perché le lunghe catene di DNA sono flosce e il metodo standard per assemblare lunghe catene è soggetto a errori.
Usando una proteina legante il DNA chiamata RecA come una sorta di armature su nanoscala, o barra d'armatura, per supportare l'impalcatura del DNA floscio, ricercatori del National Institute of Standards and Technology (NIST) hanno costruito molti dei più grandi rettangolari, forme lineari e altre mai assemblate dal DNA. Le strutture possono essere da due a tre volte più grandi di quelle costruite utilizzando tecniche standard di autoassemblaggio del DNA.
Inoltre, perché il nuovo metodo richiede meno pezzi chimicamente distinti per costruire strutture organizzate rispetto alla tecnica standard, noto come DNA origami, è probabile che riduca il numero di errori nella costruzione delle forme. Questo è un grande vantaggio per lo sforzo di produrre dispositivi affidabili basati sul DNA in grandi quantità, ha detto il ricercatore del NIST Alex Liddle.
Sebbene la capacità di RecA di legarsi al DNA a doppio filamento sia nota da anni, il team del NIST è il primo a integrare i filamenti di questa proteina nell'assemblaggio delle strutture del DNA. L'aggiunta di RecA offre un vantaggio particolare:una volta che un'unità della proteina si lega a un piccolo segmento di DNA a doppia elica, attira automaticamente altre unità per schierarsi al suo fianco, nello stesso modo in cui i magneti a barra si uniranno end-to-end. Come mattoni che riempiono una fondazione, RecA allinea l'intera lunghezza del filamento di DNA, allungamento, ampliandolo e rafforzandolo. un floscio, Un filamento di DNA largo 2 nanometri può trasformarsi in una struttura rigida più larga di quattro volte.
"Il metodo RecA estende notevolmente la capacità dei metodi di autoassemblaggio del DNA di costruire strutture più grandi e sofisticate, ", ha affermato Daniel Schiffels del NIST.
Schiffel, Liddle e la loro collega Veronika Szalai descrivono il loro lavoro in un recente articolo su ACS Nano .
Il nuovo metodo incorpora la tecnica dell'origami del DNA e la supera, secondo Liddle. Nell'origami del DNA, brevi filamenti di DNA che hanno una sequenza specifica di quattro coppie di basi sono usati come graffette per legare insieme lunghe sezioni di DNA. Per rendere lo scheletro magro del DNA più forte e più spesso, il filo può riavvolgersi su se stesso, utilizzando rapidamente la corda lunga.
Se l'origami del DNA riguarda la piegatura, Liddle ha paragonato il nuovo metodo della sua squadra alla costruzione di una stanza, a partire da una planimetria. La posizione del corto, pezzi di DNA a singolo filamento che fungono da punti metallici segnano gli angoli della stanza. Tra gli angoli giace un lungo, magro pezzo di DNA a singolo filamento. L'enzima DNA polimerasi trasforma una sezione del lungo pezzo di DNA a singolo filamento nella versione a doppio filamento della molecola, un passaggio necessario perché RecA si lega fortemente solo al DNA a doppia elica. Quindi RecA si assembla lungo tutto il doppio filo, rinforzando la struttura del DNA e limitando la necessità di ulteriori graffette per mantenerne la forma.
Con meno punti richiesti, il metodo RecA è probabilmente in grado di costruire strutture organizzate con meno errori rispetto agli origami del DNA, ha detto Liddle.