Credito:Wiley
Anche piccole quantità di virus possono avere conseguenze disastrose. L'identificazione dell'RNA può rivelare il tipo di virus presente. Sulla rivista è stata ora delineata una tecnica rapida e sensibile basata sul rilevamento ottico Angewandte Chemie . Scienziati tedeschi e finlandesi hanno dimostrato il legame di un bersaglio di RNA a una sonda fatta di nanotubi d'oro e una struttura di origami di DNA. Gli interruttori di chiralità attivati dal legame possono essere misurati mediante spettroscopia di dicroismo circolare.
Identificare l'agente patogeno, spesso un virus, che preoccupa un paziente è una delle maggiori sfide nel settore sanitario. Virus responsabili della febbre Zika, AIDS, e l'epatite C contengono sequenze di RNA mutanti. I medici devono sapere rapidamente quale tipo di virus hanno acquisito i loro pazienti, ma le attuali tecniche basate sulla moltiplicazione dell'RNA sono costose e richiedono tempo. Ora, Tim Liedl della Ludwigs-Maximilians-Universität di Monaco di Baviera, Germania, e i suoi colleghi, hanno sviluppato una strategia di rilevamento veloce basata sulla nanoplasmonica, origami del DNA, e una lettura ottica.
La luce può indurre onde plasmoniche in strutture metalliche nanometriche più piccole della lunghezza d'onda della luce incidente. Questa risonanza può portare a un'emissione di luce fortemente potenziata anche da strutture nanoscopiche, una caratteristica molto interessante per le applicazioni di biorilevamento. Liedl e colleghi hanno creato una sonda di rilevamento di dimensioni nanometriche per molecole di RNA.
La sonda, un apparato di dimensioni nanometriche fatto di DNA e nanobarre d'oro, è stato assemblato con la cosiddetta tecnica del DNA origami, che sfrutta le interazioni specifiche delle basi del DNA per piegare e incollare i singoli filamenti in qualsiasi forma desiderata. Gli autori hanno costruito due barre di eliche di DNA parallele collegate liberamente attraverso una cerniera al centro delle barre. Nanobarre d'oro sono state posizionate sopra ciascuna delle barre incrociate. Entrambi i bracci incrociati sono stati dotati di funzionalità alle estremità:gli scienziati hanno attaccato una singola sequenza di DNA integrata con un filamento di blocco a un braccio, e la sequenza di DNA complementare all'altra. In presenza di RNA bersaglio, che potrebbe essere una tipica sequenza di RNA virale, il filamento bloccante lascerebbe il suo DNA a favore dell'ibridazione dell'RNA, ed entrambe le singole sequenze di DNA formerebbero in modo complementare un doppio filamento per cui i due bracci della croce sono tirati insieme. Questo cambiamento strutturale introduce la chiralità nella sonda.
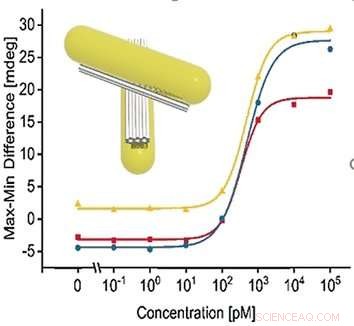
La chiralità può essere rilevata con il dicroismo circolare. E senza dubbio, i cambiamenti strutturali innescati dal legame all'RNA hanno indotto un segnale di dicroismo circolare rilevabile con uno spettrometro CD. Sono state riconosciute concentrazioni fino a 100 picomolari dell'RNA bersaglio, secondo gli autori. Gli scienziati sperano di stabilire questa tecnica nei sistemi lab-on-a-chip in cui sono necessari pochi passaggi per la preparazione del campione e dispositivi in miniatura a basso costo portano a risultati sensibili. I risultati preliminari sul siero di sangue con l'aggiunta di RNA virale erano promettenti.
Gli autori ammettono che i limiti di rilevazione non sono ancora sufficientemente bassi per essere clinicamente rilevanti. Però, credono che i miglioramenti dovrebbero essere possibili; Compreso, migliore protezione dei nanosensori dalle proteine del siero, un cambiamento verso metalli plasmonici con una migliore risonanza, e l'espansione dei siti di riconoscimento dell'RNA. Ciò potrebbe rendere la tecnica uno strumento diagnostico promettente che non è necessariamente limitato all'RNA virale.