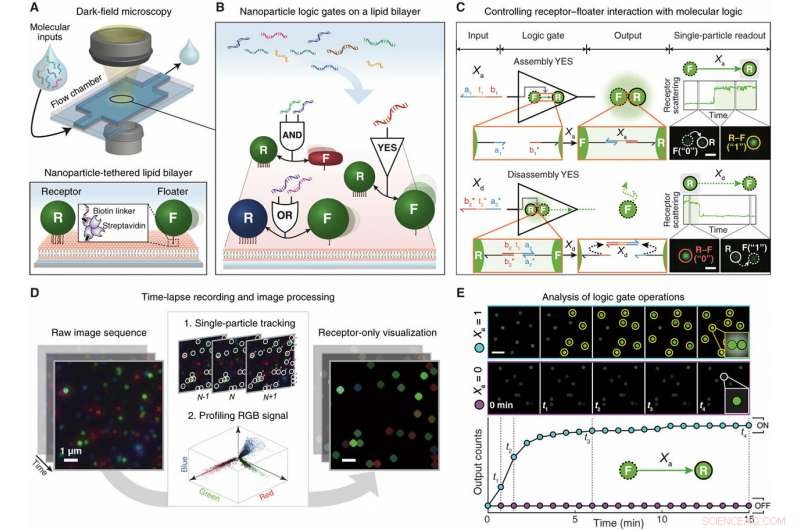
Calcolo logico a singola nanoparticella su LNT. (A) Schemi della piattaforma LNT. Due tipi di nanoparticelle modificate dal DNA, recettore immobile (R) e galleggiante mobile (F), sono collegati a un SLB e monitorati da DFM. (B) Coppie R-F come porte logiche booleane di nanoparticelle. Ogni porta logica prende il DNA come input e produce un assemblaggio o un disassemblaggio tra le due particelle come output. Le frecce bidirezionali indicano le interazioni R–F. I ligandi del DNA di superficie non sono raffigurati. (C) Porte SI a nanoparticelle singole. I domini funzionali sono rappresentati da colori e numeri in pedice con punte di freccia che indicano le loro estremità 3′. Gli asterischi indicano la complementarità. I cerchi luminosi dietro i dimeri R–F illustrano l'accoppiamento plasmonico. (D) Analisi dell'immagine. Un algoritmo di tracciamento a singola particella identifica innanzitutto i segnali del recettore da una sequenza di immagini grezze. in seguito, i segnali rilevati vengono campionati e utilizzati per generare un nuovo filmato in campo oscuro che visualizza solo i segnali del recettore. (E) Analisi cinetica. Per ogni condizione di input vengono fornite istantanee del solo recettore (in alto) e un grafico cinetico (in basso) della porta Assembly YES. Un grafico cinetico si ottiene contando cumulativamente il numero di recettori di commutazione di stato nel tempo. Credito:progressi scientifici, doi:10.1126/sciadv.aau2124
Le nanoparticelle possono essere utilizzate come substrati per il calcolo, con controllo algoritmico e autonomo delle loro proprietà uniche. Però, attualmente manca un'architettura scalabile per formare sistemi informatici basati su nanoparticelle. In un recente studio pubblicato su Progressi scientifici , Jinyoung Seo e collaboratori del Dipartimento di Chimica della Seoul National University in Corea del Sud, riportato su una piattaforma di nanoparticelle integrata con porte logiche e circuiti a livello della singola particella. Hanno implementato la piattaforma su un doppio strato lipidico di supporto. Ispirato dalle membrane cellulari in biologia che compartimentano e controllano le reti di segnalazione, gli scienziati hanno chiamato la piattaforma "nanotablet lipidico" (LNT). Per condurre nano-bio-informatica, hanno usato un doppio strato lipidico come circuito chimico e le nanoparticelle come unità di calcolo.
Su una nanocompressa lipidica in soluzione, Seo et al. ha stabilito che una singola porta logica di nanoparticelle rileva le molecole come input e attiva l'assemblaggio o il disassemblaggio delle particelle come output. Hanno dimostrato operazioni logiche booleane insieme a fan-in/fan-out di porte logiche e un circuito logico combinatorio come multiplexer nello studio. Gli scienziati prevedono che il nuovo approccio sarebbe in grado di modulare circuiti di nanoparticelle su doppi strati lipidici per progettare nuovi paradigmi e gateway nel calcolo molecolare, circuiti di nanoparticelle e nanoscienza di sistema, nel futuro.
La materia può essere fusa con il calcolo su molte scale di lunghezza, che vanno da goccioline di dimensioni micro in logica di bolle microfluidica e microparticelle a biomolecole e macchine molecolari. L'implementazione del calcolo nelle nanoparticelle rimane inesplorata, nonostante una vasta gamma di applicazioni che potrebbero beneficiare della capacità di controllare algoritmicamente l'utile fotonica, elettrico, magnetico, proprietà catalitiche e materiali delle nanoparticelle. Queste proprietà sono attualmente inaccessibili tramite sistemi molecolari. Idealmente, sistemi di nanoparticelle dotati di capacità di calcolo possono formare circuiti di nanoparticelle per svolgere autonomamente compiti complessi in risposta a stimoli esterni per combinare il flusso di materia e informazioni su scala nanometrica.
Un approccio esistente per utilizzare le nanoparticelle come substrati per il calcolo consiste nel funzionalizzare le particelle con ligandi sensibili agli stimoli. Un gruppo di tali nanoparticelle modificate eseguirà quindi operazioni logiche elementari che rispondono a una varietà di input chimici e fisici. Gli scienziati mirano a utilizzare una singola nanoparticella come nanoparti modulari ea implementare il calcolo desiderato in modo plug-and-play. Però, ci sono difficoltà nel cablaggio di più porte logiche integrate in fase di soluzione poiché è impegnativo controllare la diffusione degli ingressi, porte logiche e output nello spazio 3D. Per risolvere questa sfida, gli scienziati si sono ispirati alla membrana cellulare; un equivalente biologico di un circuito stampato che può ospitare una varietà di proteine recettoriali come unità computazionali. In natura, le proteine compartimentate interagiscono con i recettori come una rete per svolgere funzioni complesse. Le membrane possono anche consentire il verificarsi di processi di calcolo parallelo e quindi gli scienziati dei materiali sono stati ispirati a ricablare il fenomeno biologico.

Imaging time-lapse in campo oscuro di una porta YES dell'Assemblea di nanoparticelle. Credito:progressi scientifici, doi:10.1126/sciadv.aau2124.
Bioispirato dalle membrane cellulari, in questo studio, Seo et al. ha dimostrato una piattaforma di calcolo delle nanoparticelle basata su doppio strato lipidico. Come prova di principio, hanno usato nanoparticelle plasmoniche a dispersione di luce per costruire componenti di circuiti, DNA come ligandi di superficie e input molecolari insieme alle interazioni biotina-streptavidina per legare le nanoparticelle al doppio strato lipidico. Dopo aver fissato le nanoparticelle a un doppio strato lipidico supportato (SLB), hanno fornito diverse caratteristiche chiave negli esperimenti;
Gli scienziati hanno implementato il nano-biocalcolo all'interfaccia di nanostrutture e biomolecole, dove l'informazione molecolare in soluzione (input) è stata tradotta in un assemblaggio/disassemblaggio dinamico di nanoparticelle su un doppio strato lipidico (output). Come componente chiave di un LNT, Seo et al. progettato una camera di flusso con un doppio strato lipidico rivestito sul fondo del substrato.
La visualizzazione del solo recettore di un film in campo oscuro. Credito:progressi scientifici, doi:10.1126/sciadv.aau2124.
Per costruire nanocompresse lipidiche nel setup sperimentale, gli scienziati hanno utilizzato tre componenti chiave:piccole vescicole unilamellari (SUV), camere a flusso di vetro e nanoparticelle plasmoniche funzionalizzate con DNA. Le nanoparticelle modificate dal DNA aderiscono al doppio strato lipidico per formare porte logiche e circuiti che elaborano le informazioni molecolari. Gli scienziati hanno classificato le nanoparticelle funzionalizzate in recettori immobili (reporter per il calcolo) o floater mobili (vettori di informazioni per il calcolo). In tale contesto, i galleggianti erano "fili" che trasportavano le informazioni delle porte a monte nelle porte a valle attraverso una robusta diffusione laterale. Hanno caratterizzato le nanoparticelle per convalidare le loro proprietà del materiale prima di costruire i circuiti sperimentali.
Seo et al. ha utilizzato la microscopia in campo oscuro (DFM) per misurare le prestazioni delle porte logiche delle nanoparticelle in risposta agli input molecolari in soluzione. Quando le sequenze di immagini in campo oscuro sono state ottenute dalle operazioni logiche, lo scienziato li ha elaborati e quantificati utilizzando una pipeline di analisi delle immagini personalizzata.
Del tutto, gli scienziati hanno progettato porte logiche booleane di nanoparticelle e operazioni di assemblaggio e disassemblaggio di porte YES a nanoparticelle singole in tempo reale. Le porte YES a nanoparticelle singole hanno costituito gli esempi più semplici nello studio. Per rilevare i segnali di dispersione di una porta logica di nanoparticelle, gli scienziati hanno fatto affidamento sull'accoppiamento plasmonico tra due particelle centrali che componevano il cancello. Per formare le nanoparticelle, Seo et al. nanotubi d'oro sintetizzati con gusci d'argento, nanosfere d'oro e nanosfere d'argento su semi d'oro detti rossi, nanoparticelle verdi e blu per esibire il rosso, segnali di dispersione verde e blu nello studio. Gli scienziati hanno rappresentato il comportamento delle nanoparticelle a controllo logico in un modo semplice, grafico della reazione delle nanoparticelle per mostrare una reazione di assemblaggio da un galleggiante a un recettore e una reazione di disassemblaggio, fornendo una visione intuitiva del comportamento di ogni porta logica delle nanoparticelle.
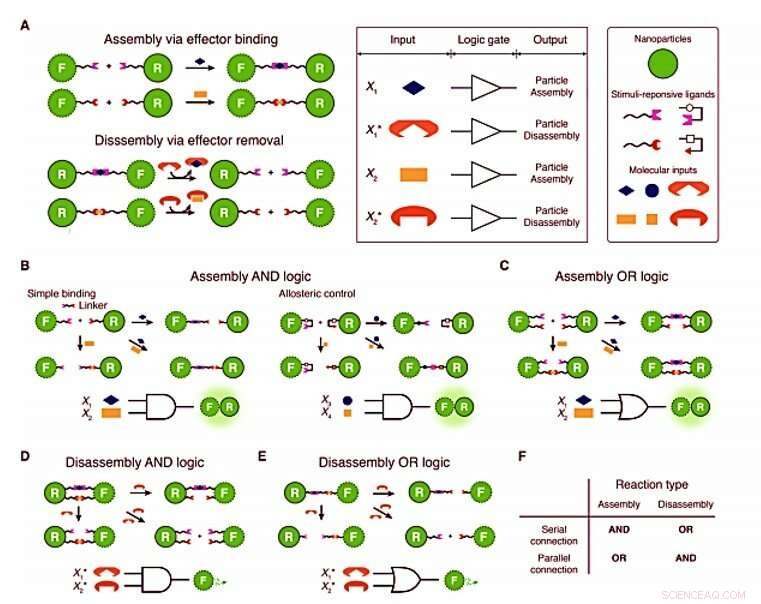
Principi di progettazione per porte logiche di nanoparticelle. (A) Riassunto grafico del concetto generalizzabile. Vengono fornite l'illustrazione delle porte YES di assemblaggio/disassemblaggio delle nanoparticelle mediate da effettori (a sinistra) e la tabella della verità per il concetto (a destra). La coppia effettore-ligando selettivo e la coppia effettore-chelatore sono necessarie per la costruzione di porte logiche di assemblaggio/disassemblaggio. Per costruire una porta logica usando due nanoparticelle, “bonding” interactions in the receptor–floater interface need to be programmed in such a way that the bonds are formed (via assembly) or cleaved (via disassembly) only if two molecular inputs satisfy AND or OR logic. (B) Two-input Assembly AND gate. (C) Two-input Assembly OR gate. Assembly reactions are controlled by AND logic when the bond-forming interaction require the serial activation by the two inputs and by OR logic when the bond-forming interaction is controlled in parallel. (D) Two-input Disassembly AND gate. (E) Two-input Disassembly OR gate. Allo stesso modo, Disassembly reactions are modulated by AND logic via parallel disconnection and by OR logic via serial disconnection. (F) Table summary. These illustrations describe the generalized concept of the interface programming. In questo studio, we used sequence recognition and strand displacement of DNA as the mechanisms to implement the logic. Nello specifico, we used single-stranded DNA molecules as effectors, thiolated oligonucleotides as ligands, and a strand displacement as chelation mechanism. We foresee that this design rules can be potentially applied to other ligand systems and core nanostructures. Credito:progressi scientifici, doi:10.1126/sciadv.aau2124.
The scientists used a sufficiently high density of nanoparticles and incorporated single-particle tracking algorithms to profile the scattering signals and visualize the receptor signals alone in a dark background. To qualitatively understand the overall computing performance of a single nanoparticle logic gate, they used the "receptor-only" view. The results showed that the population of nanoparticle logic gates switched into the ON state in response to performing a YES logic operation. The scientists deduced that a population of nanoparticle logic gates produced high output counts only when the molecular input met TRUE conditions.
To demonstrate two-input, single-nanoparticle logic gates, Seo et al. similarly developed:Assembly AND, Assembly OR, Disassembly AND, and Disassembly OR gates via "interface programming". The scientists showed that the design principles for interface programming were straightforward and could be generalized among circuits. They expanded the interface programming to enable nanoparticle logic gates to process INHIBIT logic.
The scientists then increased the complexity of reactions at the receptor-floater interface but noticed incomplete reactions or spurious interactions occuring in the system. Such anomalous interactions indicated that they could not rely on programming particle interfaces as before to construct complex circuits. Anziché, they introduced a conceptually distinct approach termed nanoparticle "network programming" to allow two single-particle logic gates to be combined with AND or, OR logic. In the resulting network programming of wired nanoparticle logic gates, the scientists showed the strategy could be implemented to build complex multilayer cascades readily without extensive optimization. Seo et al. successfully implemented the nanoparticle multiplexer to show the ability to design and operate nanoparticle circuits on LNTs in a highly modular and controlled manner.
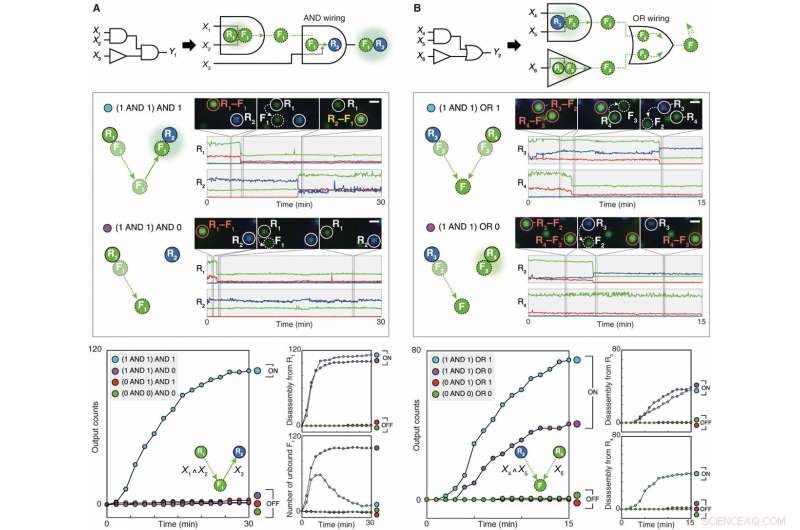
Wiring of nanoparticle logic gates via network programming. (A) Wiring with AND logic. Two logic gates (Disassembly AND gate and Assembly YES gate) are designed to operate in series for AND wiring. The floater F1, which is bound to the first receptor R1 in its initial state, acts as a Disassembly AND logic gate and subsequently as an Assembly YES gate with the second receptor R2. The generation of R2–F1 dimers is an output of the (X1 AND X2) AND X3 circuit. (B) Wiring with OR logic. Two logic gates (Disassembly AND gate and Disassembly YES gate) are designed to operate in parallel for OR wiring. The two gates both release G-NFs as outputs. The generation of the G-NFs is an output of the (X3 AND X4) OR X5 circuit. Circuit diagrams (top), single-particle dark-field analysis (middle), and kinetics analysis of circuits (lower left) and intermediate reactions (lower right). Credito:progressi scientifici, doi:10.1126/sciadv.aau2124
Scientists can expand on the demonstrated scope of lipid bilayer-based nanoparticle computation to advance the existing molecular computing technologies to operate nanoparticle circuits. They can also integrate lipid bilayers with DNA nanostructures to open the development of new molecular circuits by expanding on dynamic inter-origami interactions for more complex and practical molecular computations. Current limits of the experimental setup prevent the construction of arbitrarily large circuits. These can be overcome to generate broader design space for circuit buildup with new modes of communication, dynamic reconfiguration and DNA walkers.
Seo et al. envision that the molecular computing network can be analogously built in a similar approach to silicon-based computers that have improved through the years. The scientists can advance the experimental setup by increasing the nanoparticle density, to increase the computing capacity and expand parallelism, so that each nanoparticle may independently perform its own computation. Per applicazioni pratiche, the lipid nanotablet will play a pivotal role in building dynamic, autonomous nanosystems in molecular diagnostics and smart sensors; to sense multiple stimuli and trigger the appropriate response. If such nanocircuits are introduced into living cell membranes, scientists can create novel bioengineered nano-bio interfaces as biologic-inorganic hybrid systems. The particles can also be used separately to study membrane-associated phenomena in living cells. In questo modo, by facilitating communication between nanosystems and cellular systems, the scientists will be able to activate new pathways to navigate complex and dynamic theranostic applications.
© 2019 Scienza X Rete