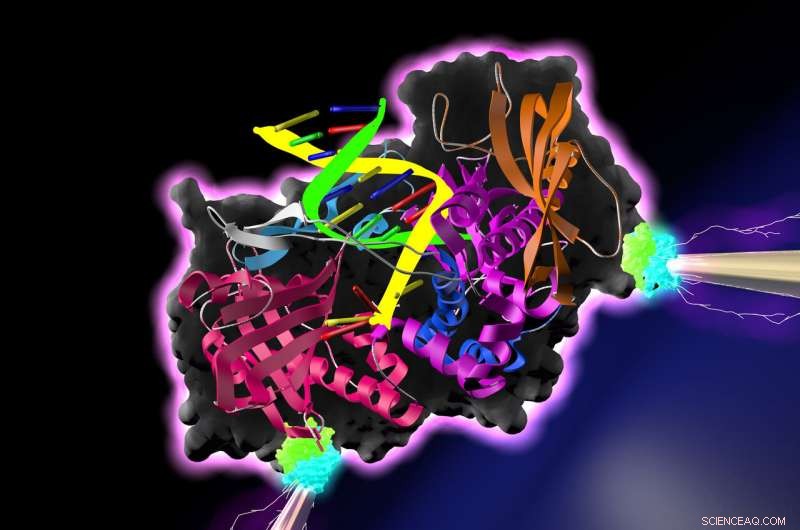
Una DNA polimerasi, un enzima che sintetizza molecole di DNA da blocchi di nucleotidi, è in bilico tra una coppia di elettrodi. Il legame dei nucleotidi da parte della polimerasi provoca picchi di conduttanza, che possono essere caratteristici delle specifiche molecole legate dalla polimerasi. Tale dispositivo, in teoria, potrebbe svolgere rapidamente, preciso, sequenziamento del DNA a basso costo, e può avere molte applicazioni aggiuntive nella diagnostica medica, produzione industriale e altri settori. Credito:laboratorio di Lindsay
In mezzo allo zoo di biomolecole essenziali alla vita, gli enzimi sono tra i più vitali. Senza queste proteine specializzate, che accelerano la velocità delle reazioni chimiche, migliaia di processi vitali essenziali, dalla crescita e digestione cellulare alla respirazione e alla funzione nervosa, sarebbe impossibile.
In una nuova ricerca, Stuart Lindsay e i suoi colleghi indagano su un'impresa scoperta di recente eseguita da enzimi, e molto probabilmente, tutte le proteine. In condizioni adeguate, possono agire come superbi conduttori di elettricità, consentendo loro di essere incorporati in una gamma di dispositivi elettronici. "È un modo per collegare la straordinaria diversità chimica degli enzimi direttamente a un computer, "Dice Lindsay.
Mentre il ruolo della conduttanza proteica in natura rimane una questione di mistero e speculazione, sfruttare questo fenomeno per l'uso umano probabilmente aprirà nuove strade per i dispositivi di rilevamento biochimici, produzione industriale intelligente e nuove innovazioni nella diagnostica medica.
Forse più emozionante, la conduttanza elettrica attraverso un tipo speciale di enzima può segnalare un progresso significativo per il sequenziamento del DNA. Utilizzando una DNA polimerasi, il lettore di DNA ad alta risoluzione della natura, in un tale dispositivo potrebbe potenzialmente consentire il sequenziamento fulmineo di interi genomi umani con una precisione senza precedenti a costi molto bassi. Il nuovo studio "apre il vaso di Pandora di guardare la funzione di qualsiasi enzima in un chip di computer".
Attualità
Gli autori del nuovo studio descrivono i trucchi che hanno usato per fissare una DNA polimerasi a una coppia di elettrodi e i conseguenti picchi di corrente associati all'enzima che si lega e rilascia successivamente nucleotidi del DNA bersaglio. La riuscita dimostrazione della conduttanza enzimatica apre la strada al montaggio di array di proteine su chip per computer, dove possono agire come processori biologici paralleli per una varietà di compiti.
"Gli enzimi sono molecole incredibili che svolgono reazioni chimiche che altrimenti non avverrebbero, " dice Lindsay. Per dare un senso al potere di queste molecole, alcune reazioni essenziali ai processi vitali, svolgersi migliaia di volte al secondo, richiederebbero millenni per verificarsi in assenza di enzimi.
Lindsay dirige il Biosedign Center for Single Molecule Biophysics presso l'Arizona State University. La ricerca primaria del centro si concentra sulla scienza al nesso tra medicina molecolare e nanotecnologia.
I risultati del suo gruppo appaiono nella prossima edizione della rivista ACS Nano .
Proteine come conduttori
Fino a poco tempo fa, le proteine erano considerate strettamente come isolanti del flusso di corrente elettrica. Ora, sembra, le loro proprietà fisiche insolite possono portare a una condizione in cui sono in bilico sensibile tra un isolante e un conduttore. (Un fenomeno noto come criticità quantistica potrebbe essere al centro del loro comportamento peculiare.)
Infatti, nelle ricerche precedenti, Lindsay ha dimostrato una forte conduttanza elettrica attraverso una proteina catturata tra una coppia di elettrodi. La nuova ricerca porta le indagini sulla conduttanza proteica un passo avanti. In precedenza, la proteina è stata collegata tramite i suoi due cosiddetti siti attivi. Queste sono le regioni di una proteina che legano molecole selezionate, spesso determinando un cambiamento conformazionale nella complessa struttura 3-D della molecola e il completamento del compito assegnato alla proteina.
Questa volta, la biomolecola è stata collegata in modo sensibile agli elettrodi mediante siti di legame alternati sull'enzima, lasciando i siti attivi disponibili per legare le molecole e svolgere la funzione proteica naturale.
Kindle della natura
La molecola enzimatica scelta per gli esperimenti è una delle più importanti per la vita. Conosciuta come DNA polimerasi, questo enzima si lega ai nucleotidi successivi in un tratto di DNA e genera una catena complementare di nucleotidi, uno per uno. Questa versatile nanomacchina viene utilizzata nei sistemi viventi per copiare il DNA durante la replicazione cellulare e per riparare rotture o altri danni al DNA.
Lo studio descrive le tecniche per fissare la DNA polimerasi agli elettrodi in modo da generare forti segnali di conduttanza per mezzo di due sostanze chimiche leganti specializzate note come biotina e streptavidina. Quando un elettrodo è stato funzionalizzato con questa tecnica, piccoli picchi di conduttanza sono stati generati mentre la DNA polimerasi si legava e rilasciava successivamente ciascun nucleotide, come una mano che afferra e rilascia una palla da baseball. Quando entrambi gli elettrodi sono stati dotati di streptavidina e biotina, segnali di conduttanza molto più forti, misura 3-5 volte più grande, sono stati osservati.

Stuart Lindsay dirige il Biodesign Center for Single Molecule Biophysics. È anche professore di Regents e Nadine e Edward Carson professore di fisica e chimica. Credito:Biodesign Institute presso ASU
L'idea di sfruttare una polimerasi per eseguire il sequenziamento rapido del DNA è stata con Lindsay per un po'. Aveva pensato di usarlo in dispositivi precedenti da lui creati in cui sezioni di DNA venivano alimentate attraverso strette giunzioni di tunnel. "Non sarebbe bello se potessi mettere un paio di elettrodi all'interno delle polimerasi perché la polimerasi afferra il DNA e lo fa passare attraverso la giunzione. Se avessi un meccanismo di lettura incorporato nella polimerasi, hai la macchina per il sequenziamento ideale."
Il nuovo metodo spera di adottare un approccio diverso, utilizzando l'esperienza di lettura della velocità della polimerasi per fornire una lettura dei nucleotidi attraverso picchi di conduttanza specifici per ciascuna delle 4 basi del DNA. In pratica, è necessario superare una serie di ostacoli di progettazione. Il corretto fissaggio della polimerasi per la conduttanza elettrica è un affare delicato e ha comportato molti tentativi ed errori. I siti di legame devono essere progettati in domini specifici che non influenzano il ripiegamento e la funzione delle proteine e le connessioni devono essere escogitate per impedire all'enzima stesso di entrare in contatto con gli elettrodi. Anche l'uso della biotina per legare la molecola sembra essere critico per l'elevata conduttanza. La biotina che lega una tasca della streptavidina sembra aiutare a guidare il trasporto di elettroni in profondità all'interno della proteina, massimizzando così la conduttanza.
Separare i segnali di conduttanza che registrano ciascuna base di DNA successiva dal rumore di fondo e dai movimenti casuali dei punti di contatto per l'enzima si è anche dimostrato impegnativo e sofisticati algoritmi di apprendimento automatico vengono utilizzati per chiarire le letture della conduttanza. Lindsay crede che molti di questi problemi di rumore saranno risolti quando le polimerasi saranno incorporate in chip adeguatamente isolati e sigillati che tengono l'enzima rigidamente in posizione.
frontiere degli enzimi
Il primo genoma umano completo è stato una pietra miliare per la scienza e la medicina. Lo sforzo erculeo del Progetto Genoma Umano ha consumato 13 anni di lavoro al costo di un miliardo di dollari. Ora potrebbero aprirsi le porte di una nuova era della bioelettronica proteica, con molte sorprese probabilmente in serbo.
Se gli ostacoli tecnici rimanenti possono essere superati, Il sequenziamento del DNA potrebbe essere effettuato alla velocità vertiginosa di una DNA polimerasi funzionale, o circa un centinaio di nucleotidi al secondo. "Se metti 10, 000 molecole su un chip—non è una cosa difficile da fare—sequenzerete un intero genoma in meno di un'ora, "Lindsay dice