
Applicazione in vitro del test utilizzando più tipi di cellule con simulazione ed estrazione della velocità. (A) Dati di fluorescenza e configurazione del test che rappresentano la fluorescenza QD (QSH) non lavata per i pozzetti non lavati che contengono reni (MES13), fegato (AML12), endoteliale (C166), e macrofagi (J774a.1) tipi di cellule. AU, unità arbitrarie. (B) Concentrazioni di assorbimento della fluorescenza grezza lavata (nero) e calibrata (rosso) (cerchi, misurato) rispetto agli attacchi di simulazione (linee) per il fegato, endoteliale, macrofago, e tipi di cellule renali. (C) Riepilogo delle costanti di velocità ottimizzate dall'algoritmo genetico per l'adattamento del modello ai set di dati misurati. (D) Immagini di microscopia confocale dipendenti dal tempo dell'assorbimento di QD, che mostra la saturazione (frecce a doppia faccia) per ogni tipo di cellula nello studio (il blu e il rosso rappresentano il nucleo cellulare Hoechst e la fluorescenza QSH, rispettivamente). Cartoon è una rappresentazione visiva della cinetica di interazione delle cellule QD. Credito: Progressi scientifici , doi:10.1126/sciadv.aax2642
I bioingegneri possono progettare farmaci intelligenti per terapie basate su anticorpi e nanomateriali per ottimizzare l'efficienza dei farmaci per prodotti sempre più efficienti, studi preclinici in fase iniziale. Il farmaco ideale avrà la massima efficienza nei siti dei tessuti bersaglio per il trasporto dal sistema vascolare tissutale all'ambiente cellulare. I ricercatori possono utilizzare simulazioni biologiche accoppiate ad approcci in vitro per prevedere la loro esposizione in modo rapido ed efficiente per prevedere la biodistribuzione dei farmaci all'interno di singole cellule di tessuto animale vivo senza fare affidamento su studi sugli animali. In un nuovo studio ora pubblicato su Progressi scientifici , Edward Price e Andre J. Gesquiere hanno utilizzato con successo un'analisi in vitro e un modello di fluidodinamica computazionale (CFD) per tradurre la cinetica cellulare in vitro in simulazioni del corpo intero su più specie e tipi di nanomateriali. Il lavoro ha permesso loro di prevedere la distribuzione dei farmaci all'interno delle singole cellule tissutali e il team si aspetta che questo lavoro venga perfezionato, ridurre e sostituire i test sugli animali fornendo agli scienziati una nuova prospettiva sullo sviluppo dei farmaci.
Le nanomedicine (NM) sotto forma di anticorpi e nanomateriali sintetici possono integrare la medicina convenzionale a piccole molecole attraverso il targeting attivo dei tessuti, tempi di circolazione variabili e stabilità, accoppiato alla biodistribuzione regolabile. I team di ricerca fanno molto affidamento su modelli animali per quantificare le dosi erogate, sollevando questioni di etica e impennate di tempo e di costi. Dal punto di vista scientifico, la distruzione dell'architettura tissutale condotta durante l'omogeneizzazione dei tessuti per la quantificazione può eliminare la conoscenza critica del trasporto della nanomedicina all'interno delle cellule tissutali e del sistema vascolare. Un farmaco può raggiungere le cellule esibendo una farmacocinetica plasmatica ottimale per raggiungere il sistema vascolare tissutale. L'architettura e il diametro delle nanoparticelle consentono il trasporto critico del farmaco attraverso le pareti dei microvasi in un ambiente cellulare infetto o normale.
Nel presente lavoro, Price e Gesquiere hanno accoppiato un approccio di simulazione in vitro/in silico per prevedere la biodistribuzione NM all'interno delle specie precliniche (modelli animali e cellulari) a livello della singola cellula. Hanno tenuto conto delle differenze nella dimensione delle nanoparticelle, specie animali e diametri delle dimensioni dei pori del sistema vascolare utilizzando un modello fluidodinamico. Il team ha quantificato il contenuto di NM cellulare in situazioni in cui i risultati erano difficili da tradurre in studi sugli animali a causa della mancanza di prospettive sistematiche. Hanno anche affrontato problemi sperimentali esistenti in cui le cellule inducevano la degradazione della fluorescenza delle nanoparticelle per produrre falsi negativi.
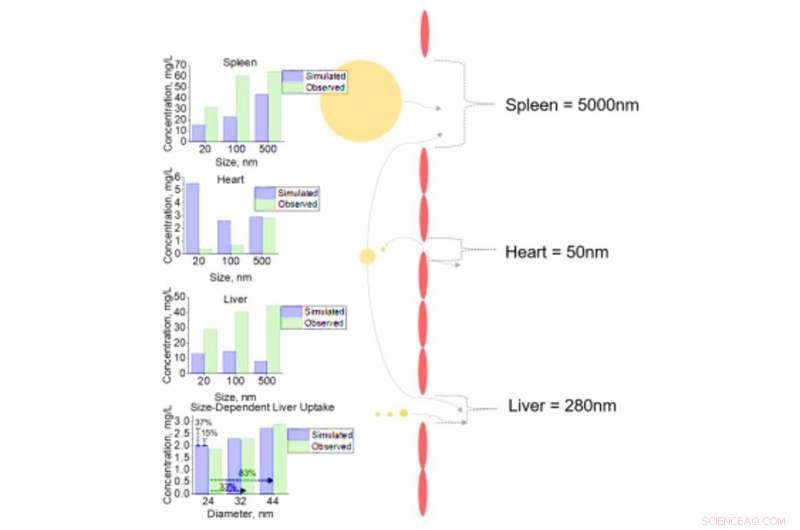
Analisi cellulare dell'assorbimento di NM in tessuti variabili per NM con diametri diversi. Captazione cellulare di NM con diametri di 20, 100, e 500nm per la milza, cuore, e fegato con fenestra di diametro 5000, 50, e 280nm rispettivamente. Analisi di sensibilità delle variazioni incrementali del diametro NM (24, 32, e 44 nm) con successivo assorbimento di cellule epatiche. Tutti gli output simulati (previsti) vengono confrontati con i dati osservati dai set di dati cellulari della letteratura. Credito: Progressi scientifici , doi:10.1126/sciadv.aax2642
Per convalidare il loro lavoro in vitro, il team ha simulato l'analisi lisosomiale accoppiata alla spettroscopia di assorbimento atomico (AAS), e simulazioni in vivo comprovate confrontando i risultati con la letteratura pubblicata sui dati sugli animali del corpo intero per i ratti, topi e primati non umani. Gli scienziati hanno utilizzato punti quantici (QD) come sistema modello grazie al loro potenziale nel rilevamento delle nanoparticelle, considerando anche i loro limiti. Prezzo et al. ha tenuto conto delle complessità in una piattaforma NM per estendere le capacità alle nanoparticelle a base di anticorpi e metallo/polimero.
Quando una nanoparticella circola attraverso l'apporto di sangue animale per entrare nell'ambiente tissutale, interagirà con le cellule dei tessuti tramite adsorbimento, desorbimento, processi di interiorizzazione o di assorbimento attivo. Gli scienziati hanno esposto le nanoparticelle a QD (punti quantici) non tossici per rilevare in modo fluorescente le loro interazioni con le cellule comunemente incontrate dai NM come i macrofagi, cellule endoteliali e cellule epiteliali, dopo iniezione endovenosa. Il team di ricerca ha quantificato la concentrazione di nanoparticelle che interagiscono con le cellule in vitro e ha costruito simulazioni di cinetica cellulare per valutare la cinetica della velocità e le interazioni tra cellule NM.
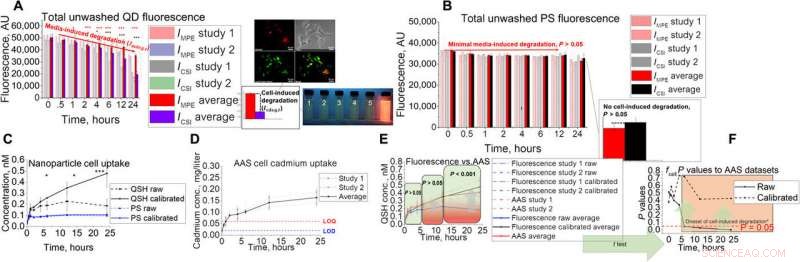
Sviluppo di test in vitro e validazione per AAS. (A e B) Configurazione del test in vitro con il suo segnale di fluorescenza non lavato accoppiato per (A) QD e (B) PS. Le immagini mostrano la colocalizzazione Hepa1-6 di QD nei compartimenti lisosomiali e istantanee di fluorescenza sotto illuminazione ultravioletta in condizioni di esposizione lisosomiale simulata con pH 2,5, 3.0, 3.5, 4.0, 4.5, e 7.4 (da 1 a 6, rispettivamente). (C a E) studi di captazione QD utilizzando la tecnica in vitro con (C) fluorescenza, (D) AS, (E) combinato, e i risultati statistici (F) utilizzando il test t a due code quando si confronta l'assorbimento di fluorescenza grezzo e calibrato con i dati AAS. Gli asterischi in figura rappresentano la significatività al * (P <0.05), ** (P <0,01), e *** (P <0,001) livelli. Credito: Progressi scientifici , doi:10.1126/sciadv.aax2642
La simulazione conteneva un (i) mezzo, (ii) membrana cellulare e (iii) compartimenti dello spazio cellulare interconnessi tramite equazioni di trasferimento di massa di base e costanti di velocità del primo ordine. Hanno ottimizzato il sistema utilizzando un algoritmo genetico in MATLAB e hanno notato che la costante del tasso di assorbimento della membrana è più alta per i macrofagi e più bassa per le cellule endoteliali e del fegato. Dei tipi cellulari, il processo di interiorizzazione era anche relativamente più alto per le cellule del tessuto epatico e più basso per le cellule endoteliali. L'imaging confocale ha supportato visivamente questi dati con un rapido assorbimento e saturazione dei macrofagi entro un'ora dall'esposizione, mentre le cellule del tessuto epiteliale ed endoteliale sono state ulteriormente ritardate.
Il team ha dimostrato la presenza di fattori di degradazione cellulare all'interno di saggi in vitro utilizzando simulazioni di cinetica cellulare per aiutare a tradurre in modo efficiente i dati in vitro in studi sugli animali. La spettroscopia di adsorbimento atomico (AAS) ha convalidato i risultati dell'assorbimento del QD tramite saggi di fluorescenza e ha evidenziato l'importanza del calcolo della degradazione indotta dalle cellule e dal mezzo. Poiché le nanomedicine (NM) situate all'interno dei tessuti possono accumularsi negli interstizi, vascolare o all'interno di una varietà di tipi di cellule, il team ha tradotto la cinetica cellulare in vitro in simulazioni di animali in silico per tenerne conto. Hanno condotto due studi pilota e valutato le tendenze nell'assorbimento del tessuto intero in funzione delle dimensioni e dell'assorbimento di NM quantificato a livello della singola cellula. Architettonicamente, ogni simulazione di compartimento tissutale conteneva quattro sottocompartimenti per rappresentare l'epiteliale, cellule endoteliali e macrofagiche.
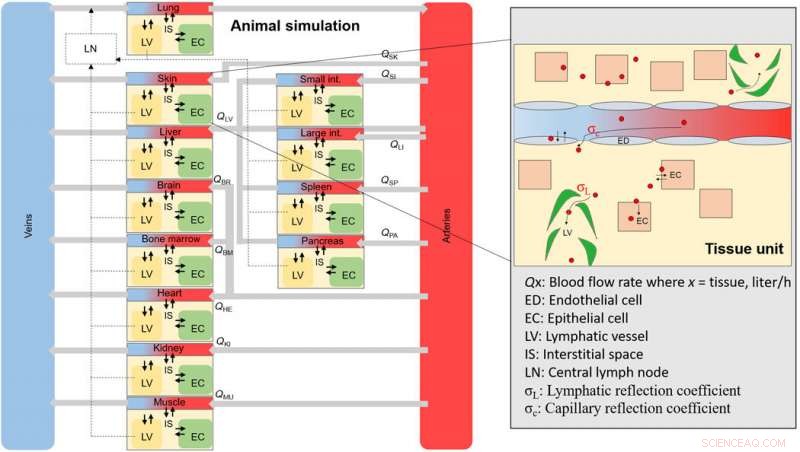
Uno schema generale della simulazione NM animale. Schema dei tessuti complessivi e dei compartimenti sanguigni che compongono l'intero animale. Ogni compartimento tissutale è interconnesso attraverso i tassi di flusso sanguigno e contiene sotto-compartimenti (area ingrandita), dove un NM si distribuirà all'internalizzazione attraverso le finestre endoteliali. Credito: Progressi scientifici , doi:10.1126/sciadv.aax2642
Poiché il fegato e la milza sono bersagli comuni per l'accumulo di NM, hanno usato questi tessuti come casi di studio per comprendere e catturare, la sensibilità e l'accuratezza della simulazione. Per esempio, la simulazione ha mostrato che le dimensioni NM aumentate hanno superato le dimensioni dei pori del tessuto della maggior parte dei tipi di tessuto per incanalare verso i tessuti con dimensioni dei pori più grandi come il fegato. A questo punto, le cellule endoteliali del fegato hanno interagito con gli NM per un rapido legame di membrana e saturazione entro 24 ore. Quando la MN entrava nell'interstizio del tessuto, interagiva rapidamente con le cellule del tessuto epiteliale (per il fegato) e macrofagico (per la milza). I risultati hanno rafforzato la fiducia nel potere predittivo della simulazione e nella sua capacità di prevedere per la prima volta il contenuto di farmaci di dimensioni variabili a livello di singola cellula per gli animali, rigorosamente sulla base dei soli dati in vitro.
Gli scienziati hanno quindi indicato il potere predittivo e la capacità di traslazione della simulazione confrontando il contenuto misurato a livello di tessuto per più tipi e specie di NM, compresi i topi, topi e contenuto cellulare (epiteliale, endoteliali e macrofagi). Hanno testato le prestazioni predittive su più variabili in base alle linee guida e agli standard dell'Organizzazione mondiale della sanità del consorzio accademico e farmaceutico per lo sviluppo e la sicurezza dei farmaci. Le medie previste dal modello e le medie osservate sono correlate linearmente per indicare previsioni ragionevoli del modello in tutte le specie e tipi di NM.
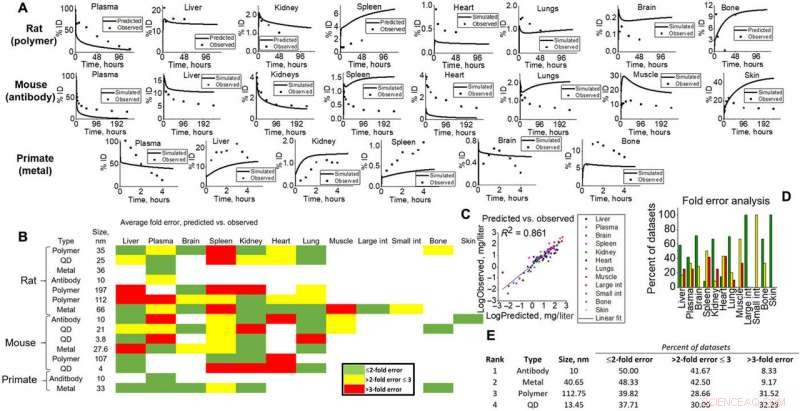
Output di simulazione per la convalida di vari studi sugli animali. (A) Esempi a livello di tessuto previsti (linea) rispetto a quelli osservati (punti) per tutti i tipi di specie (ratti, i topi, e NHP) per prove visive delle capacità del modello. (B) Mappa termica dell'analisi dell'errore di piega calcolata per tutti i tessuti, nm, e tipi di specie nella simulazione. Verde, giallo, e il rosso rappresentano <2 volte, <3 volte, e>3 volte gli errori. L'errore di piegatura è stato calcolato in base alle equazioni fornite in Materiali e metodi, dove le medie dei set di dati simulati sono state confrontate con quelle osservate. (C) Tutte le medie dei punti dati specifiche per i tipi di tessuto per le simulazioni e osservate sono state tracciate l'una contro l'altra per ottenere un adattamento lineare (R2 =0,861). (D) Gli errori di piegatura specifici del tessuto vengono mostrati per visualizzare l'accuratezza del modello per ciascun tessuto in questo studio. (E) Gli output simulati da NM sono stati classificati (da 1 a 4) in base all'analisi dell'errore di piegatura. Credito: Progressi scientifici , doi:10.1126/sciadv.aax2642
In questo modo, Edward Price e Andre J. Gesquiere hanno sviluppato una piattaforma praticabile per ridurre e perfezionare i test sugli animali durante lo sviluppo della nanomedicina (NM). I dati concentrati in vitro accoppiati alle simulazioni della cinetica cellulare hanno fornito interazioni cellula-NM per mostrare un eccellente potenziale traslazionale. I risultati non hanno richiesto l'adattamento ai set di dati sulla biodistribuzione animale, poiché tutti i parametri sono stati ottimizzati su dati in vitro o calcolati utilizzando un modello fluidodinamico. Il processo ha funzionato anche in condizioni di degradazione NM nell'ambiente biologico. Le tecniche combinate in vitro e in silico aiuteranno la futura progettazione di farmaci intelligenti per aiutare gli scienziati a formare discussioni migliori e informate riducendo al contempo i test sugli animali vivi. Price e Gesquiere si aspettano che questo lavoro serva a un nuovo approccio per le simulazioni predittive del trasporto della nanomedicina.
© 2020 Scienza X Rete